Mol:FLIAABGS0006
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 38 41 0 0 0 0 0 0 0 0999 V2000 | + | 38 41 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.4594 0.5126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4594 0.5126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9031 0.1915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9031 0.1915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3468 0.5126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3468 0.5126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2093 0.1916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2093 0.1916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2093 -0.4744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2093 -0.4744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7861 -0.8074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7861 -0.8074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3628 -0.4744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3628 -0.4744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3628 0.1916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3628 0.1916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7861 0.5246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7861 0.5246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9029 -0.4505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9029 -0.4505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3468 -0.7716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3468 -0.7716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7861 -1.4729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7861 -1.4729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9391 -0.8071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9391 -0.8071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9391 -1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9391 -1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4905 -1.7621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4905 -1.7621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0418 -1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0418 -1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0418 -0.8071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0418 -0.8071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4905 -0.4888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4905 -0.4888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5633 0.3089 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.5633 0.3089 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -3.2170 -0.1482 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.2170 -0.1482 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.7184 0.0457 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.7184 0.0457 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.1988 0.0400 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.1988 0.0400 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.5869 0.4006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5869 0.4006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0963 0.2177 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.0963 0.2177 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.9503 0.5324 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9503 0.5324 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5439 -0.5784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5439 -0.5784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4328 -0.4338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4328 -0.4338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3317 0.5363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3317 0.5363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3573 -1.4123 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3573 -1.4123 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3317 1.1964 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3317 1.1964 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7911 1.5085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7911 1.5085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1627 1.1457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1627 1.1457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5441 1.5028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5441 1.5028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9033 1.1329 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9033 1.1329 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5441 2.0014 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5441 2.0014 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7911 2.0625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7911 2.0625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9078 -1.9438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9078 -1.9438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7738 -2.4439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7738 -2.4439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 4 1 0 0 0 0 | + | 9 4 1 0 0 0 0 |
| − | 2 10 1 0 0 0 0 | + | 2 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 5 1 0 0 0 0 | + | 11 5 1 0 0 0 0 |
| − | 6 12 2 0 0 0 0 | + | 6 12 2 0 0 0 0 |
| − | 7 13 1 0 0 0 0 | + | 7 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 19 20 1 1 0 0 0 | + | 19 20 1 1 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 22 1 1 0 0 0 0 | + | 22 1 1 0 0 0 0 |
| − | 11 29 1 0 0 0 0 | + | 11 29 1 0 0 0 0 |
| − | 28 30 1 0 0 0 0 | + | 28 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 33 35 2 0 0 0 0 | + | 33 35 2 0 0 0 0 |
| − | 31 36 2 0 0 0 0 | + | 31 36 2 0 0 0 0 |
| − | 16 37 1 0 0 0 0 | + | 16 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 37 38 | + | M SAL 2 2 37 38 |
| − | M SBL 2 1 40 | + | M SBL 2 1 40 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 40 3.2359 -1.65 | + | M SVB 2 40 3.2359 -1.65 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 33 35 34 | + | M SAL 1 3 33 35 34 |
| − | M SBL 1 1 36 | + | M SBL 1 1 36 |
| − | M SMT 1 COOH | + | M SMT 1 COOH |
| − | M SVB 1 36 -1.5441 1.5028 | + | M SVB 1 36 -1.5441 1.5028 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FLIAABGS0006 | + | ID FLIAABGS0006 |
| − | KNApSAcK_ID C00010117 | + | KNApSAcK_ID C00010117 |
| − | NAME Bichanin A 7-O-glucoside-6''-malonate | + | NAME Bichanin A 7-O-glucoside-6''-malonate |
| − | CAS_RN 34232-17-2 | + | CAS_RN 34232-17-2 |
| − | FORMULA C25H24O13 | + | FORMULA C25H24O13 |
| − | EXACTMASS 532.121690854 | + | EXACTMASS 532.121690854 |
| − | AVERAGEMASS 532.45026 | + | AVERAGEMASS 532.45026 |
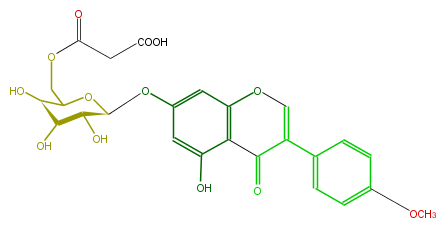
| − | SMILES O=C(CC(O)=O)OCC([C@H](O)4)O[C@H]([C@H]([C@H]4O)O)Oc(c1)cc(O3)c(C(C(=C3)c(c2)ccc(OC)c2)=O)c1O | + | SMILES O=C(CC(O)=O)OCC([C@H](O)4)O[C@H]([C@H]([C@H]4O)O)Oc(c1)cc(O3)c(C(C(=C3)c(c2)ccc(OC)c2)=O)c1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
38 41 0 0 0 0 0 0 0 0999 V2000
-1.4594 0.5126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9031 0.1915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3468 0.5126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2093 0.1916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2093 -0.4744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7861 -0.8074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3628 -0.4744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3628 0.1916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7861 0.5246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9029 -0.4505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3468 -0.7716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7861 -1.4729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9391 -0.8071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9391 -1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4905 -1.7621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0418 -1.4438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0418 -0.8071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4905 -0.4888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5633 0.3089 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-3.2170 -0.1482 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.7184 0.0457 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.1988 0.0400 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.5869 0.4006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0963 0.2177 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.9503 0.5324 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5439 -0.5784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4328 -0.4338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3317 0.5363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3573 -1.4123 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3317 1.1964 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7911 1.5085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1627 1.1457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5441 1.5028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9033 1.1329 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5441 2.0014 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7911 2.0625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9078 -1.9438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7738 -2.4439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 4 1 0 0 0 0
2 10 1 0 0 0 0
10 11 2 0 0 0 0
11 5 1 0 0 0 0
6 12 2 0 0 0 0
7 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
19 20 1 1 0 0 0
20 21 1 1 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
24 28 1 0 0 0 0
22 1 1 0 0 0 0
11 29 1 0 0 0 0
28 30 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
33 35 2 0 0 0 0
31 36 2 0 0 0 0
16 37 1 0 0 0 0
37 38 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 37 38
M SBL 2 1 40
M SMT 2 OCH3
M SVB 2 40 3.2359 -1.65
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 33 35 34
M SBL 1 1 36
M SMT 1 COOH
M SVB 1 36 -1.5441 1.5028
S SKP 8
ID FLIAABGS0006
KNApSAcK_ID C00010117
NAME Bichanin A 7-O-glucoside-6''-malonate
CAS_RN 34232-17-2
FORMULA C25H24O13
EXACTMASS 532.121690854
AVERAGEMASS 532.45026
SMILES O=C(CC(O)=O)OCC([C@H](O)4)O[C@H]([C@H]([C@H]4O)O)Oc(c1)cc(O3)c(C(C(=C3)c(c2)ccc(OC)c2)=O)c1O
M END