Mol:FLIFHXNF0011
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 32 36 0 0 0 0 0 0 0 0999 V2000 | + | 32 36 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.8722 0.2250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8722 0.2250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8722 -0.4174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8722 -0.4174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3159 -0.7385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3159 -0.7385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7596 -0.4174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7596 -0.4174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7596 0.2250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7596 0.2250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3159 0.5462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3159 0.5462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2033 -0.7385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2033 -0.7385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3530 -0.4174 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.3530 -0.4174 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.3530 0.2250 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.3530 0.2250 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.2033 0.5462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2033 0.5462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9091 -0.7384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9091 -0.7384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9091 -1.4252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9091 -1.4252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5039 -1.7686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5039 -1.7686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0987 -1.4252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0987 -1.4252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0987 -0.7384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0987 -0.7384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5039 -0.3950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5039 -0.3950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2033 -1.3807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2033 -1.3807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5309 0.2250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5309 0.2250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9093 0.5462 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.9093 0.5462 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.3530 -1.1167 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3530 -1.1167 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4495 1.1745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4495 1.1745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0883 1.2417 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.0883 1.2417 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -2.3496 0.6548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3496 0.6548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4094 1.7977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4094 1.7977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0890 2.3527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0890 2.3527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0501 1.7977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0501 1.7977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8930 1.2445 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8930 1.2445 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3159 -1.3807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3159 -1.3807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0865 -1.9402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0865 -1.9402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8010 -2.3527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8010 -2.3527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0987 -1.4252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0987 -1.4252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0987 -1.4252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0987 -1.4252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 8 11 1 0 0 0 0 | + | 8 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 11 2 0 0 0 0 | + | 16 11 2 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 16 18 1 0 0 0 0 | + | 16 18 1 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 19 9 1 0 0 0 0 | + | 19 9 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 6 21 1 0 0 0 0 | + | 6 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 1 1 0 0 0 0 | + | 23 1 1 0 0 0 0 |
| − | 22 24 1 0 0 0 0 | + | 22 24 1 0 0 0 0 |
| − | 24 25 2 0 0 0 0 | + | 24 25 2 0 0 0 0 |
| − | 24 26 1 0 0 0 0 | + | 24 26 1 0 0 0 0 |
| − | 19 27 1 0 0 0 0 | + | 19 27 1 0 0 0 0 |
| − | 3 28 1 0 0 0 0 | + | 3 28 1 0 0 0 0 |
| − | 13 29 1 0 0 0 0 | + | 13 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 14 31 1 0 0 0 0 | + | 14 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 31 32 | + | M SAL 2 2 31 32 |
| − | M SBL 2 1 35 | + | M SBL 2 1 35 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 35 2.3356 -1.6315 | + | M SVB 2 35 2.3356 -1.6315 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 29 30 | + | M SAL 1 2 29 30 |
| − | M SBL 1 1 33 | + | M SBL 1 1 33 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 33 1.0865 -1.9402 | + | M SVB 1 33 1.0865 -1.9402 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FLIFHXNF0011 | + | ID FLIFHXNF0011 |
| − | KNApSAcK_ID C00009594 | + | KNApSAcK_ID C00009594 |
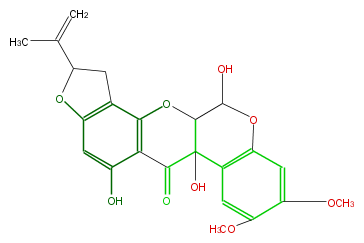
| − | NAME Villol;6,12a-Dihydroxysumatrol | + | NAME Villol;6,12a-Dihydroxysumatrol |
| − | CAS_RN 65206-37-3 | + | CAS_RN 65206-37-3 |
| − | FORMULA C23H22O9 | + | FORMULA C23H22O9 |
| − | EXACTMASS 442.126382302 | + | EXACTMASS 442.126382302 |
| − | AVERAGEMASS 442.41538 | + | AVERAGEMASS 442.41538 |
| − | SMILES C(O3)(C(O)2)C(O)(C(c(c5O)c3c(C4)c(c5)OC(C(C)=C)4)=O)c(c1O2)cc(c(OC)c1)OC | + | SMILES C(O3)(C(O)2)C(O)(C(c(c5O)c3c(C4)c(c5)OC(C(C)=C)4)=O)c(c1O2)cc(c(OC)c1)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
32 36 0 0 0 0 0 0 0 0999 V2000
-1.8722 0.2250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8722 -0.4174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3159 -0.7385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7596 -0.4174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7596 0.2250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3159 0.5462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2033 -0.7385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3530 -0.4174 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.3530 0.2250 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.2033 0.5462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9091 -0.7384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9091 -1.4252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5039 -1.7686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0987 -1.4252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0987 -0.7384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5039 -0.3950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2033 -1.3807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5309 0.2250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9093 0.5462 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.3530 -1.1167 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4495 1.1745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0883 1.2417 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-2.3496 0.6548 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4094 1.7977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0890 2.3527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0501 1.7977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8930 1.2445 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3159 -1.3807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0865 -1.9402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8010 -2.3527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0987 -1.4252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0987 -1.4252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
8 11 1 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 11 2 0 0 0 0
7 17 2 0 0 0 0
16 18 1 0 0 0 0
18 19 1 0 0 0 0
19 9 1 0 0 0 0
8 20 1 0 0 0 0
1 2 1 0 0 0 0
6 21 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 0 0 0 0
23 1 1 0 0 0 0
22 24 1 0 0 0 0
24 25 2 0 0 0 0
24 26 1 0 0 0 0
19 27 1 0 0 0 0
3 28 1 0 0 0 0
13 29 1 0 0 0 0
29 30 1 0 0 0 0
14 31 1 0 0 0 0
31 32 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 31 32
M SBL 2 1 35
M SMT 2 OCH3
M SVB 2 35 2.3356 -1.6315
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 29 30
M SBL 1 1 33
M SMT 1 OCH3
M SVB 1 33 1.0865 -1.9402
S SKP 8
ID FLIFHXNF0011
KNApSAcK_ID C00009594
NAME Villol;6,12a-Dihydroxysumatrol
CAS_RN 65206-37-3
FORMULA C23H22O9
EXACTMASS 442.126382302
AVERAGEMASS 442.41538
SMILES C(O3)(C(O)2)C(O)(C(c(c5O)c3c(C4)c(c5)OC(C(C)=C)4)=O)c(c1O2)cc(c(OC)c1)OC
M END