Mol:BMFYS2ESa001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 51 53 0 0 1 0 0 0 0 0999 V2000 | + | 51 53 0 0 1 0 0 0 0 0999 V2000 |
| − | 2.9781 -3.8348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9781 -3.8348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2872 -4.7859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2872 -4.7859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -3.6269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -3.6269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.3353 -2.8049 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.3353 -2.8049 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.4692 -2.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.4692 -2.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.4692 -1.3049 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.4692 -1.3049 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.3353 -0.8049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.3353 -0.8049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.2013 -1.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.2013 -1.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.2013 -2.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.2013 -2.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.9444 -0.6358 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.9444 -0.6358 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.5377 0.2778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.5377 0.2778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.5432 0.1733 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.5432 0.1733 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.0673 -2.8049 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.0673 -2.8049 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.8740 0.9164 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 19.8740 0.9164 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 20.0820 1.8946 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 20.0820 1.8946 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 19.2159 2.3946 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 19.2159 2.3946 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 18.4728 1.7254 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 18.4728 1.7254 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 17.4946 1.9333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.4946 1.9333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.9955 2.3013 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.9955 2.3013 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.1114 3.3891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.1114 3.3891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.8795 0.8119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.8795 0.8119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.8255 1.1902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.8255 1.1902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.9204 3.9769 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.9204 3.9769 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.5082 3.1678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.5082 3.1678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.7294 4.5646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.7294 4.5646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.3326 4.7859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.3326 4.7859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.8474 1.3981 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.8474 1.3981 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.6394 0.4200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.6394 0.4200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.0553 2.3762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.0553 2.3762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.8692 1.6060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.8692 1.6060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.2001 0.8629 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.2001 0.8629 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.9432 0.1937 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.9432 0.1937 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.4569 1.5320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.4569 1.5320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.5309 0.1197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.5309 0.1197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.5528 0.3276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.5528 0.3276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.8837 -0.4155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.8837 -0.4155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.6268 -1.0846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.6268 -1.0846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.1405 0.2536 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.1405 0.2536 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.2145 -1.1587 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 11.2145 -1.1587 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 10.2364 -0.9507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.2364 -0.9507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.5673 -1.6939 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.5673 -1.6939 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.5891 -1.4860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.5891 -1.4860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.9200 -2.2291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.9200 -2.2291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9418 -2.0212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9418 -2.0212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2727 -2.7644 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2727 -2.7644 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2946 -2.5564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2946 -2.5564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6254 -3.2996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6254 -3.2996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6473 -3.0917 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6473 -3.0917 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.5236 -2.1097 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.5236 -2.1097 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.9274 0.0003 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.9274 0.0003 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.6328 -1.0702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.6328 -1.0702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 31 33 1 0 0 0 0 | + | 31 33 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 39 1 0 0 0 0 | + | 36 39 1 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 8 7 2 0 0 0 0 | + | 8 7 2 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 7 6 1 0 0 0 0 | + | 7 6 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 6 5 2 0 0 0 0 | + | 6 5 2 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 5 4 1 0 0 0 0 | + | 5 4 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 4 9 2 0 0 0 0 | + | 4 9 2 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 9 8 1 0 0 0 0 | + | 9 8 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 1 3 2 0 0 0 0 | + | 1 3 2 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 1 48 1 0 0 0 0 | + | 1 48 1 0 0 0 0 |
| − | 39 49 1 1 0 0 0 | + | 39 49 1 1 0 0 0 |
| − | 40 50 2 0 0 0 0 | + | 40 50 2 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 44 51 2 0 0 0 0 | + | 44 51 2 0 0 0 0 |
| − | 22 18 1 0 0 0 0 | + | 22 18 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 8 10 1 0 0 0 0 | + | 8 10 1 0 0 0 0 |
| − | 15 14 1 0 0 0 0 | + | 15 14 1 0 0 0 0 |
| − | 14 21 1 6 0 0 0 | + | 14 21 1 6 0 0 0 |
| − | 17 21 1 6 0 0 0 | + | 17 21 1 6 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 16 15 1 0 0 0 0 | + | 16 15 1 0 0 0 0 |
| − | 16 20 1 1 0 0 0 | + | 16 20 1 1 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 15 19 1 1 0 0 0 | + | 15 19 1 1 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 14 12 1 0 0 0 0 | + | 14 12 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 23 20 1 0 0 0 0 | + | 23 20 1 0 0 0 0 |
| − | 31 34 1 0 0 0 0 | + | 31 34 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 12 7 1 0 0 0 0 | + | 12 7 1 0 0 0 0 |
| − | 23 26 1 0 0 0 0 | + | 23 26 1 0 0 0 0 |
| − | 31 32 2 0 0 0 0 | + | 31 32 2 0 0 0 0 |
| − | 23 25 2 0 0 0 0 | + | 23 25 2 0 0 0 0 |
| − | S SKP 7 | + | S SKP 7 |
| − | ID BMFYS2ESa001 | + | ID BMFYS2ESa001 |
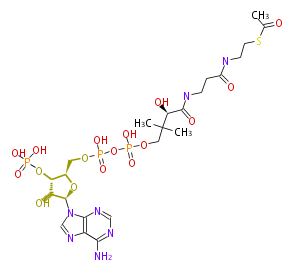
| − | NAME Acetyl-CoA | + | NAME Acetyl-CoA |
| − | FORMULA C23H38N7O17P3S | + | FORMULA C23H38N7O17P3S |
| − | EXACTMASS 809.1257 | + | EXACTMASS 809.1257 |
| − | AVERAGEMASS 809.572 | + | AVERAGEMASS 809.572 |
| − | SMILES C(CNC(CCNC([C@H](O)C(C)(C)COP(OP(OC[C@@H](O1)[C@@H](OP(O)(O)=O)[C@@H](O)[C@@H]1n(c32)cnc2c(N)ncn3)(O)=O)(O)=O)=O)=O)SC(C)=O | + | SMILES C(CNC(CCNC([C@H](O)C(C)(C)COP(OP(OC[C@@H](O1)[C@@H](OP(O)(O)=O)[C@@H](O)[C@@H]1n(c32)cnc2c(N)ncn3)(O)=O)(O)=O)=O)=O)SC(C)=O |
| − | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C00024 | + | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C00024 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
51 53 0 0 1 0 0 0 0 0999 V2000
2.9781 -3.8348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2872 -4.7859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -3.6269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
20.3353 -2.8049 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
19.4692 -2.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
19.4692 -1.3049 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
20.3353 -0.8049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
21.2013 -1.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
21.2013 -2.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
21.9444 -0.6358 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
21.5377 0.2778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
20.5432 0.1733 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
22.0673 -2.8049 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
19.8740 0.9164 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
20.0820 1.8946 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
19.2159 2.3946 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
18.4728 1.7254 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
17.4946 1.9333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
20.9955 2.3013 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
19.1114 3.3891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
18.8795 0.8119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
16.8255 1.1902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
19.9204 3.9769 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
20.5082 3.1678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
20.7294 4.5646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
19.3326 4.7859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
15.8474 1.3981 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
15.6394 0.4200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
16.0553 2.3762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
14.8692 1.6060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
14.2001 0.8629 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
14.9432 0.1937 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
13.4569 1.5320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
13.5309 0.1197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
12.5528 0.3276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.8837 -0.4155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.6268 -1.0846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.1405 0.2536 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.2145 -1.1587 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
10.2364 -0.9507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.5673 -1.6939 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
8.5891 -1.4860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.9200 -2.2291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.9418 -2.0212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.2727 -2.7644 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
5.2946 -2.5564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6254 -3.2996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6473 -3.0917 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
11.5236 -2.1097 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
9.9274 0.0003 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.6328 -1.0702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
31 33 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 39 1 0 0 0 0
1 2 1 0 0 0 0
39 40 1 0 0 0 0
40 41 1 0 0 0 0
8 7 2 0 0 0 0
41 42 1 0 0 0 0
7 6 1 0 0 0 0
42 43 1 0 0 0 0
6 5 2 0 0 0 0
43 44 1 0 0 0 0
5 4 1 0 0 0 0
44 45 1 0 0 0 0
4 9 2 0 0 0 0
45 46 1 0 0 0 0
9 8 1 0 0 0 0
46 47 1 0 0 0 0
47 48 1 0 0 0 0
1 3 2 0 0 0 0
36 37 1 0 0 0 0
9 13 1 0 0 0 0
36 38 1 0 0 0 0
1 48 1 0 0 0 0
39 49 1 1 0 0 0
40 50 2 0 0 0 0
27 29 1 0 0 0 0
44 51 2 0 0 0 0
22 18 1 0 0 0 0
27 28 2 0 0 0 0
27 22 1 0 0 0 0
11 12 1 0 0 0 0
27 30 1 0 0 0 0
8 10 1 0 0 0 0
15 14 1 0 0 0 0
14 21 1 6 0 0 0
17 21 1 6 0 0 0
16 17 1 0 0 0 0
16 15 1 0 0 0 0
16 20 1 1 0 0 0
30 31 1 0 0 0 0
15 19 1 1 0 0 0
17 18 1 0 0 0 0
14 12 1 0 0 0 0
10 11 2 0 0 0 0
23 20 1 0 0 0 0
31 34 1 0 0 0 0
23 24 1 0 0 0 0
12 7 1 0 0 0 0
23 26 1 0 0 0 0
31 32 2 0 0 0 0
23 25 2 0 0 0 0
S SKP 7
ID BMFYS2ESa001
NAME Acetyl-CoA
FORMULA C23H38N7O17P3S
EXACTMASS 809.1257
AVERAGEMASS 809.572
SMILES C(CNC(CCNC([C@H](O)C(C)(C)COP(OP(OC[C@@H](O1)[C@@H](OP(O)(O)=O)[C@@H](O)[C@@H]1n(c32)cnc2c(N)ncn3)(O)=O)(O)=O)=O)=O)SC(C)=O
KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C00024
M END