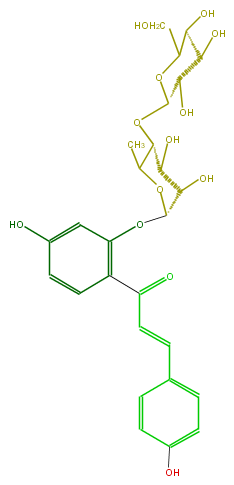
Mol:FL1C1AGS0006
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 40 43 0 0 0 0 0 0 0 0999 V2000 | + | 40 43 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.1305 2.8475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1305 2.8475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1305 1.9985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1305 1.9985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3952 1.5739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3952 1.5739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3400 1.9985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3400 1.9985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3400 2.8475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3400 2.8475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3952 3.2719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3952 3.2719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0750 1.5742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0750 1.5742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8082 1.9976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8082 1.9976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5399 1.5751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5399 1.5751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2705 1.9966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2705 1.9966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9849 1.5840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9849 1.5840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6994 1.9966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6994 1.9966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6994 2.8216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6994 2.8216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9849 3.2342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9849 3.2342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2705 2.8216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2705 2.8216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0750 0.7275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0750 0.7275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2226 3.1568 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2226 3.1568 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3952 0.7254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3952 0.7254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8653 3.2718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8653 3.2718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5820 0.0585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5820 0.0585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9274 -0.5400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9274 -0.5400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2631 -0.3501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2631 -0.3501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5947 -0.5400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5947 -0.5400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2491 0.0585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2491 0.0585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9135 -0.1313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9135 -0.1313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2126 -0.0533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2126 -0.0533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5995 -0.4686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5995 -0.4686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8464 -0.9336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8464 -0.9336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6127 -1.1822 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6127 -1.1822 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9320 -2.6233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9320 -2.6233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1716 -2.5944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1716 -2.5944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0026 -1.9046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0026 -1.9046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6007 -1.3441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6007 -1.3441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2882 -1.4918 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2882 -1.4918 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4774 -2.1846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4774 -2.1846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0933 -3.2254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0933 -3.2254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5268 -3.2096 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5268 -3.2096 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2549 -1.5559 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2549 -1.5559 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2898 -2.3279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2898 -2.3279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2226 -3.2719 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2226 -3.2719 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 10 1 0 0 0 0 | + | 15 10 1 0 0 0 0 |
| − | 7 16 2 0 0 0 0 | + | 7 16 2 0 0 0 0 |
| − | 17 13 1 0 0 0 0 | + | 17 13 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 18 1 0 0 0 0 | + | 24 18 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 27 1 0 0 0 0 | + | 33 27 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 43 | + | M SBL 1 1 43 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 43 0.8125 0.1433 | + | M SBV 1 43 0.8125 0.1433 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL1C1AGS0006 | + | ID FL1C1AGS0006 |
| − | FORMULA C27H32O13 | + | FORMULA C27H32O13 |
| − | EXACTMASS 564.18429111 | + | EXACTMASS 564.18429111 |
| − | AVERAGEMASS 564.53518 | + | AVERAGEMASS 564.53518 |
| − | SMILES OC(C4O)C(O)C(OC4CO)OC(C1O)C(OC(Oc(c(C(C=Cc(c3)ccc(O)c3)=O)2)cc(O)cc2)C1O)C | + | SMILES OC(C4O)C(O)C(OC4CO)OC(C1O)C(OC(Oc(c(C(C=Cc(c3)ccc(O)c3)=O)2)cc(O)cc2)C1O)C |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
40 43 0 0 0 0 0 0 0 0999 V2000
-1.1305 2.8475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1305 1.9985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3952 1.5739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3400 1.9985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3400 2.8475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3952 3.2719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0750 1.5742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8082 1.9976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5399 1.5751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2705 1.9966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9849 1.5840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6994 1.9966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6994 2.8216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9849 3.2342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2705 2.8216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0750 0.7275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2226 3.1568 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3952 0.7254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8653 3.2718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5820 0.0585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9274 -0.5400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2631 -0.3501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5947 -0.5400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2491 0.0585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9135 -0.1313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2126 -0.0533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5995 -0.4686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8464 -0.9336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6127 -1.1822 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9320 -2.6233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1716 -2.5944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0026 -1.9046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6007 -1.3441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2882 -1.4918 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4774 -2.1846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0933 -3.2254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5268 -3.2096 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2549 -1.5559 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2898 -2.3279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2226 -3.2719 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 10 1 0 0 0 0
7 16 2 0 0 0 0
17 13 1 0 0 0 0
3 18 1 0 0 0 0
1 19 1 0 0 0 0
20 21 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 18 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 27 1 0 0 0 0
39 40 1 0 0 0 0
35 39 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 43
M SMT 1 ^ CH2OH
M SBV 1 43 0.8125 0.1433
S SKP 5
ID FL1C1AGS0006
FORMULA C27H32O13
EXACTMASS 564.18429111
AVERAGEMASS 564.53518
SMILES OC(C4O)C(O)C(OC4CO)OC(C1O)C(OC(Oc(c(C(C=Cc(c3)ccc(O)c3)=O)2)cc(O)cc2)C1O)C
M END