Mol:FL1C1AGS0008
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 50 54 0 0 0 0 0 0 0 0999 V2000 | + | 50 54 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.9132 0.6448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9132 0.6448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9132 -0.1802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9132 -0.1802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1988 -0.5927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1988 -0.5927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4843 -0.1802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4843 -0.1802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4843 0.6448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4843 0.6448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1988 1.0573 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1988 1.0573 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2302 -0.5927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2302 -0.5927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9446 -0.1802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9446 -0.1802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9446 0.6448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9446 0.6448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6591 1.0573 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6591 1.0573 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3735 0.6448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3735 0.6448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0879 1.0573 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0879 1.0573 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0879 1.8822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0879 1.8822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3735 2.2947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3735 2.2947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6591 1.8822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6591 1.8822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2302 -1.4177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2302 -1.4177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1988 -1.3040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1988 -1.3040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5845 0.8896 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5845 0.8896 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2158 1.2661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2158 1.2661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8033 0.5514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8033 0.5514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0049 0.7605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0049 0.7605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2114 0.5337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2114 0.5337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6240 1.2484 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6240 1.2484 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4224 1.0394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4224 1.0394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7502 1.4671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7502 1.4671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3614 1.6187 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3614 1.6187 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8919 0.8758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8919 0.8758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3675 0.5514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3675 0.5514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4215 0.1771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4215 0.1771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8460 2.3416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8460 2.3416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2464 0.1071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2464 0.1071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0506 0.2923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0506 0.2923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0844 1.1169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0844 1.1169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5337 1.8091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5337 1.8091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7295 1.6240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7295 1.6240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6955 0.7994 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6955 0.7994 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1078 0.5569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1078 0.5569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8831 -0.0380 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8831 -0.0380 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5669 -0.4964 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5669 -0.4964 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7839 -0.1697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7839 -0.1697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8403 1.4232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8403 1.4232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3401 -1.9269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3401 -1.9269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3401 -2.3416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3401 -2.3416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5241 -2.2896 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5241 -2.2896 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3563 -1.1881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3563 -1.1881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8919 -1.5670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8919 -1.5670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9749 -1.5670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9749 -1.5670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5241 -1.9269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5241 -1.9269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9341 -1.3084 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9341 -1.3084 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8621 -0.9028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8621 -0.9028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 10 1 0 0 0 0 | + | 15 10 1 0 0 0 0 |
| − | 7 16 2 0 0 0 0 | + | 7 16 2 0 0 0 0 |
| − | 3 17 1 0 0 0 0 | + | 3 17 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 19 20 1 1 0 0 0 | + | 19 20 1 1 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 19 27 1 0 0 0 0 | + | 19 27 1 0 0 0 0 |
| − | 20 28 1 0 0 0 0 | + | 20 28 1 0 0 0 0 |
| − | 21 29 1 0 0 0 0 | + | 21 29 1 0 0 0 0 |
| − | 22 18 1 0 0 0 0 | + | 22 18 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 31 39 1 0 0 0 0 | + | 31 39 1 0 0 0 0 |
| − | 32 40 1 0 0 0 0 | + | 32 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 48 42 1 1 0 0 0 | + | 48 42 1 1 0 0 0 |
| − | 47 42 1 1 0 0 0 | + | 47 42 1 1 0 0 0 |
| − | 46 48 1 1 0 0 0 | + | 46 48 1 1 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 48 44 1 0 0 0 0 | + | 48 44 1 0 0 0 0 |
| − | 49 46 1 0 0 0 0 | + | 49 46 1 0 0 0 0 |
| − | 45 50 1 0 0 0 0 | + | 45 50 1 0 0 0 0 |
| − | 47 49 1 0 0 0 0 | + | 47 49 1 0 0 0 0 |
| − | 42 45 1 0 0 0 0 | + | 42 45 1 0 0 0 0 |
| − | 46 41 1 0 0 0 0 | + | 46 41 1 0 0 0 0 |
| − | 34 30 1 0 0 0 0 | + | 34 30 1 0 0 0 0 |
| − | 30 13 1 0 0 0 0 | + | 30 13 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL1C1AGS0008 | + | ID FL1C1AGS0008 |
| − | FORMULA C32H40O18 | + | FORMULA C32H40O18 |
| − | EXACTMASS 712.2214644759999 | + | EXACTMASS 712.2214644759999 |
| − | AVERAGEMASS 712.6492 | + | AVERAGEMASS 712.6492 |
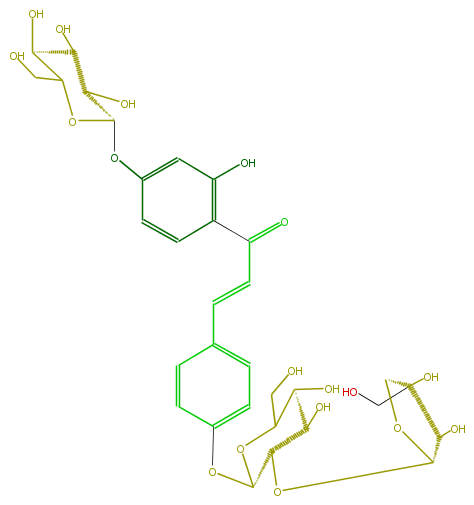
| − | SMILES OC(C(Oc(c5)cc(c(c5)C(=O)C=Cc(c2)ccc(OC(O3)C(OC(O4)C(O)C(C4)(O)CO)C(O)C(C3CO)O)c2)O)1)C(C(C(CO)O1)O)O | + | SMILES OC(C(Oc(c5)cc(c(c5)C(=O)C=Cc(c2)ccc(OC(O3)C(OC(O4)C(O)C(C4)(O)CO)C(O)C(C3CO)O)c2)O)1)C(C(C(CO)O1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
50 54 0 0 0 0 0 0 0 0999 V2000
-1.9132 0.6448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9132 -0.1802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1988 -0.5927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4843 -0.1802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4843 0.6448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1988 1.0573 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2302 -0.5927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9446 -0.1802 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9446 0.6448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6591 1.0573 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3735 0.6448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0879 1.0573 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0879 1.8822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3735 2.2947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6591 1.8822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2302 -1.4177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1988 -1.3040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5845 0.8896 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2158 1.2661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8033 0.5514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0049 0.7605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2114 0.5337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6240 1.2484 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4224 1.0394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7502 1.4671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.3614 1.6187 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.8919 0.8758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.3675 0.5514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4215 0.1771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8460 2.3416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2464 0.1071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0506 0.2923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0844 1.1169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5337 1.8091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7295 1.6240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6955 0.7994 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1078 0.5569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8831 -0.0380 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5669 -0.4964 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7839 -0.1697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8403 1.4232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3401 -1.9269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3401 -2.3416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5241 -2.2896 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3563 -1.1881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.8919 -1.5670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9749 -1.5670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5241 -1.9269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9341 -1.3084 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8621 -0.9028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 10 1 0 0 0 0
7 16 2 0 0 0 0
3 17 1 0 0 0 0
1 18 1 0 0 0 0
19 20 1 1 0 0 0
20 21 1 1 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
19 27 1 0 0 0 0
20 28 1 0 0 0 0
21 29 1 0 0 0 0
22 18 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
31 39 1 0 0 0 0
32 40 1 0 0 0 0
33 41 1 0 0 0 0
48 42 1 1 0 0 0
47 42 1 1 0 0 0
46 48 1 1 0 0 0
42 43 1 0 0 0 0
48 44 1 0 0 0 0
49 46 1 0 0 0 0
45 50 1 0 0 0 0
47 49 1 0 0 0 0
42 45 1 0 0 0 0
46 41 1 0 0 0 0
34 30 1 0 0 0 0
30 13 1 0 0 0 0
S SKP 5
ID FL1C1AGS0008
FORMULA C32H40O18
EXACTMASS 712.2214644759999
AVERAGEMASS 712.6492
SMILES OC(C(Oc(c5)cc(c(c5)C(=O)C=Cc(c2)ccc(OC(O3)C(OC(O4)C(O)C(C4)(O)CO)C(O)C(C3CO)O)c2)O)1)C(C(C(CO)O1)O)O
M END