Mol:FL1C3CGS0004
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 46 0 0 0 0 0 0 0 0999 V2000 | + | 43 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.9718 -0.5479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9718 -0.5479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9718 -1.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9718 -1.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4178 -1.5075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4178 -1.5075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1362 -1.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1362 -1.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1362 -0.5479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1362 -0.5479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4178 -0.2281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4178 -0.2281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6900 -1.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6900 -1.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2425 -1.1883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2425 -1.1883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7938 -1.5066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7938 -1.5066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3440 -1.1890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3440 -1.1890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8823 -1.4998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8823 -1.4998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4207 -1.1890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4207 -1.1890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4207 -0.5674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4207 -0.5674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8823 -0.2565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8823 -0.2565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3440 -0.5674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3440 -0.5674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6900 -2.1453 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6900 -2.1453 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4178 -2.1469 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4178 -2.1469 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5255 -0.2282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5255 -0.2282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9589 -0.2566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9589 -0.2566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4567 -1.5581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4567 -1.5581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9589 -1.4998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9589 -1.4998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5718 -0.4453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5718 -0.4453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2256 -0.9024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2256 -0.9024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7270 -0.7085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7270 -0.7085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2073 -0.7142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2073 -0.7142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5955 -0.3536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5955 -0.3536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1048 -0.5365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1048 -0.5365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9589 -0.6688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9589 -0.6688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7653 -0.9286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7653 -0.9286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4413 -1.1880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4413 -1.1880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5503 -0.0910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5503 -0.0910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5503 0.5893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5503 0.5893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0359 1.1037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0359 1.1037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3638 1.6717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3638 1.6717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4361 1.1037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4361 1.1037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1463 1.6057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1463 1.6057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5674 1.6057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5674 1.6057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2550 1.0646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2550 1.0646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6301 1.0646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6301 1.0646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3177 1.6057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3177 1.6057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6301 2.1469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6301 2.1469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2550 2.1469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2550 2.1469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3065 1.6057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3065 1.6057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 10 1 0 0 0 0 | + | 15 10 1 0 0 0 0 |
| − | 7 16 2 0 0 0 0 | + | 7 16 2 0 0 0 0 |
| − | 3 17 1 0 0 0 0 | + | 3 17 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 13 19 1 0 0 0 0 | + | 13 19 1 0 0 0 0 |
| − | 20 2 1 0 0 0 0 | + | 20 2 1 0 0 0 0 |
| − | 12 21 1 0 0 0 0 | + | 12 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 25 18 1 0 0 0 0 | + | 25 18 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 42 37 1 0 0 0 0 | + | 42 37 1 0 0 0 0 |
| − | 40 43 1 0 0 0 0 | + | 40 43 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL1C3CGS0004 | + | ID FL1C3CGS0004 |
| − | KNApSAcK_ID C00007895 | + | KNApSAcK_ID C00007895 |
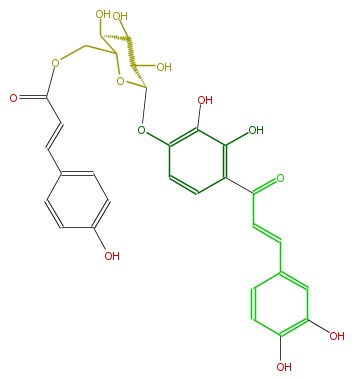
| − | NAME Okanin 4'-(6''-p-coumarylglucoside) | + | NAME Okanin 4'-(6''-p-coumarylglucoside) |
| − | CAS_RN 115070-75-2 | + | CAS_RN 115070-75-2 |
| − | FORMULA C30H28O13 | + | FORMULA C30H28O13 |
| − | EXACTMASS 596.152990982 | + | EXACTMASS 596.152990982 |
| − | AVERAGEMASS 596.53552 | + | AVERAGEMASS 596.53552 |
| − | SMILES C(O)(C3O)C(C(OC(COC(C=Cc(c4)ccc(O)c4)=O)3)Oc(c2)c(O)c(O)c(c2)C(C=Cc(c1)ccc(O)c1O)=O)O | + | SMILES C(O)(C3O)C(C(OC(COC(C=Cc(c4)ccc(O)c4)=O)3)Oc(c2)c(O)c(O)c(c2)C(C=Cc(c1)ccc(O)c1O)=O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 46 0 0 0 0 0 0 0 0999 V2000
-0.9718 -0.5479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9718 -1.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4178 -1.5075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1362 -1.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1362 -0.5479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4178 -0.2281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6900 -1.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2425 -1.1883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7938 -1.5066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3440 -1.1890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8823 -1.4998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4207 -1.1890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4207 -0.5674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8823 -0.2565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3440 -0.5674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6900 -2.1453 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4178 -2.1469 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5255 -0.2282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9589 -0.2566 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4567 -1.5581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9589 -1.4998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5718 -0.4453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2256 -0.9024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7270 -0.7085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2073 -0.7142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5955 -0.3536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1048 -0.5365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9589 -0.6688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7653 -0.9286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4413 -1.1880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5503 -0.0910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5503 0.5893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0359 1.1037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3638 1.6717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4361 1.1037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1463 1.6057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5674 1.6057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2550 1.0646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6301 1.0646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3177 1.6057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6301 2.1469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2550 2.1469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3065 1.6057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 10 1 0 0 0 0
7 16 2 0 0 0 0
3 17 1 0 0 0 0
1 18 1 0 0 0 0
13 19 1 0 0 0 0
20 2 1 0 0 0 0
12 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
27 31 1 0 0 0 0
25 18 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 2 0 0 0 0
33 35 1 0 0 0 0
35 36 2 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 41 1 0 0 0 0
41 42 2 0 0 0 0
42 37 1 0 0 0 0
40 43 1 0 0 0 0
S SKP 8
ID FL1C3CGS0004
KNApSAcK_ID C00007895
NAME Okanin 4'-(6''-p-coumarylglucoside)
CAS_RN 115070-75-2
FORMULA C30H28O13
EXACTMASS 596.152990982
AVERAGEMASS 596.53552
SMILES C(O)(C3O)C(C(OC(COC(C=Cc(c4)ccc(O)c4)=O)3)Oc(c2)c(O)c(O)c(c2)C(C=Cc(c1)ccc(O)c1O)=O)O
M END