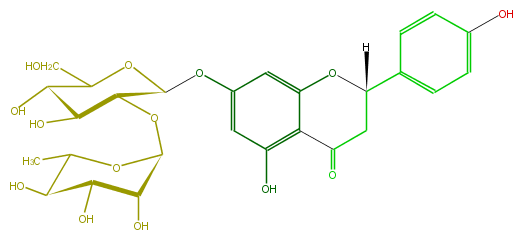
Mol:FL2FAAGS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.4151 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4151 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4151 -0.2283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4151 -0.2283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2802 -0.6296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2802 -0.6296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9754 -0.2283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9754 -0.2283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9754 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9754 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2802 0.9759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2802 0.9759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6707 -0.6296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6707 -0.6296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3659 -0.2283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3659 -0.2283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3659 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3659 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6707 0.9759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6707 0.9759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0608 0.9757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0608 0.9757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7753 0.5632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7753 0.5632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4899 0.9757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4899 0.9757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4899 1.8007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4899 1.8007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7753 2.2133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7753 2.2133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0608 1.8007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0608 1.8007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3659 1.5201 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3659 1.5201 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2802 -1.4319 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2802 -1.4319 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1098 0.9757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1098 0.9757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8204 0.5654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8204 0.5654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2832 0.6892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2832 0.6892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6372 0.0431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6372 0.0431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8432 0.4955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8432 0.4955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5756 1.1417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5756 1.1417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3696 0.6892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3696 0.6892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0467 0.0496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0467 0.0496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9634 0.1918 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9634 0.1918 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4092 -0.0750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4092 -0.0750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6707 -1.1459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6707 -1.1459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7949 -0.7230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7949 -0.7230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2890 -1.5786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2890 -1.5786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3390 -1.3071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3390 -1.3071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3833 -1.5786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3833 -1.5786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8892 -0.7230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8892 -0.7230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8393 -0.9944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8393 -0.9944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3833 -2.2133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3833 -2.2133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5385 -2.0516 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5385 -2.0516 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8386 -1.3728 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8386 -1.3728 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4812 -0.8481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4812 -0.8481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2029 2.2124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2029 2.2124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0958 1.1162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0958 1.1162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2029 1.4128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2029 1.4128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 9 17 1 1 0 0 0 | + | 9 17 1 1 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 20 23 1 1 0 0 0 | + | 20 23 1 1 0 0 0 |
| − | 20 24 1 0 0 0 0 | + | 20 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 21 1 0 0 0 0 | + | 25 21 1 0 0 0 0 |
| − | 23 26 1 0 0 0 0 | + | 23 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 7 29 2 0 0 0 0 | + | 7 29 2 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 33 36 1 0 0 0 0 | + | 33 36 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 31 38 1 0 0 0 0 | + | 31 38 1 0 0 0 0 |
| − | 30 39 1 0 0 0 0 | + | 30 39 1 0 0 0 0 |
| − | 34 26 1 0 0 0 0 | + | 34 26 1 0 0 0 0 |
| − | 14 40 1 0 0 0 0 | + | 14 40 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 25 41 1 0 0 0 0 | + | 25 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 46 0.7262 -0.4270 | + | M SBV 1 46 0.7262 -0.4270 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL2FAAGS0001 | + | ID FL2FAAGS0001 |
| − | FORMULA C27H31HO14 | + | FORMULA C27H31HO14 |
| − | EXACTMASS 580.179205732 | + | EXACTMASS 580.179205732 |
| − | AVERAGEMASS 580.53458 | + | AVERAGEMASS 580.53458 |
| − | SMILES C(C4([H])c(c5)ccc(c5)O)C(=O)c(c3O)c(O4)cc(c3)OC(C1OC(O2)C(C(O)C(O)C(C)2)O)OC(CO)C(C1O)O | + | SMILES C(C4([H])c(c5)ccc(c5)O)C(=O)c(c3O)c(O4)cc(c3)OC(C1OC(O2)C(C(O)C(O)C(C)2)O)OC(CO)C(C1O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-0.4151 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4151 -0.2283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2802 -0.6296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9754 -0.2283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9754 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2802 0.9759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6707 -0.6296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3659 -0.2283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3659 0.5746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6707 0.9759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0608 0.9757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7753 0.5632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4899 0.9757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4899 1.8007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7753 2.2133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0608 1.8007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3659 1.5201 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
0.2802 -1.4319 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1098 0.9757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8204 0.5654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2832 0.6892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6372 0.0431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8432 0.4955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5756 1.1417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3696 0.6892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0467 0.0496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9634 0.1918 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4092 -0.0750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6707 -1.1459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7949 -0.7230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2890 -1.5786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3390 -1.3071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3833 -1.5786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8892 -0.7230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8393 -0.9944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3833 -2.2133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5385 -2.0516 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8386 -1.3728 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4812 -0.8481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2029 2.2124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0958 1.1162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2029 1.4128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
9 17 1 1 0 0 0
3 18 1 0 0 0 0
1 19 1 0 0 0 0
19 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
20 23 1 1 0 0 0
20 24 1 0 0 0 0
24 25 1 0 0 0 0
25 21 1 0 0 0 0
23 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
7 29 2 0 0 0 0
30 31 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
33 36 1 0 0 0 0
32 37 1 0 0 0 0
31 38 1 0 0 0 0
30 39 1 0 0 0 0
34 26 1 0 0 0 0
14 40 1 0 0 0 0
41 42 1 0 0 0 0
25 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 46
M SMT 1 ^CH2OH
M SBV 1 46 0.7262 -0.4270
S SKP 5
ID FL2FAAGS0001
FORMULA C27H31HO14
EXACTMASS 580.179205732
AVERAGEMASS 580.53458
SMILES C(C4([H])c(c5)ccc(c5)O)C(=O)c(c3O)c(O4)cc(c3)OC(C1OC(O2)C(C(O)C(O)C(C)2)O)OC(CO)C(C1O)O
M END