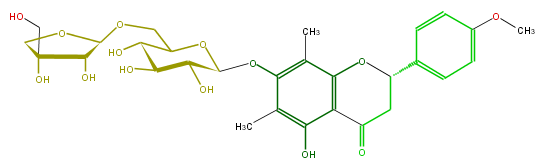
Mol:FL2FABGM0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.1919 0.1544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1919 0.1544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9046 0.5700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9046 0.5700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1951 -0.6706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1951 -0.6706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9119 -1.0800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9119 -1.0800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6245 -0.6643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6245 -0.6643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6208 0.1607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6208 0.1607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3408 -1.0736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3408 -1.0736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0535 -0.6580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0535 -0.6580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0499 0.1670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0499 0.1670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3335 0.5764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3335 0.5764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7036 0.5479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7036 0.5479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4197 0.1381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4197 0.1381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1326 0.5534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1326 0.5534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1294 1.3784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1294 1.3784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4133 1.7881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4133 1.7881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7005 1.3728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7005 1.3728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3419 -1.7169 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3419 -1.7169 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4335 0.5155 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4335 0.5155 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9119 -1.7881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9119 -1.7881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5021 -1.0731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5021 -1.0731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9046 1.3167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9046 1.3167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2786 1.0139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2786 1.0139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8660 0.2992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8660 0.2992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0675 0.5084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0675 0.5084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2741 0.2815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2741 0.2815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6866 0.9963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6866 0.9963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4850 0.7872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4850 0.7872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9289 1.3451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9289 1.3451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7998 1.4782 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7998 1.4782 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8016 0.7768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8016 0.7768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5388 0.3493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5388 0.3493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6839 -0.1561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6839 -0.1561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7163 1.7172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7163 1.7172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8418 0.6409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8418 0.6409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8418 0.0894 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8418 0.0894 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6577 0.1503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6577 0.1503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8256 1.3798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8256 1.3798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2899 1.0009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2899 1.0009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.2070 1.0009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.2070 1.0009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6577 0.6409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6577 0.6409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2478 1.2596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2478 1.2596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.3198 1.6651 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.3198 1.6651 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3198 1.3688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3198 1.3688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 1 3 1 0 0 0 0 | + | 1 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 2 1 0 0 0 0 | + | 6 2 1 0 0 0 0 |
| − | 5 7 1 0 0 0 0 | + | 5 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 6 1 0 0 0 0 | + | 10 6 1 0 0 0 0 |
| − | 9 11 1 6 0 0 0 | + | 9 11 1 6 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 4 19 1 0 0 0 0 | + | 4 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 2 21 1 0 0 0 0 | + | 2 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 14 33 1 0 0 0 0 | + | 14 33 1 0 0 0 0 |
| − | 40 34 1 1 0 0 0 | + | 40 34 1 1 0 0 0 |
| − | 39 34 1 1 0 0 0 | + | 39 34 1 1 0 0 0 |
| − | 38 40 1 1 0 0 0 | + | 38 40 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 40 36 1 0 0 0 0 | + | 40 36 1 0 0 0 0 |
| − | 41 38 1 0 0 0 0 | + | 41 38 1 0 0 0 0 |
| − | 37 42 1 0 0 0 0 | + | 37 42 1 0 0 0 0 |
| − | 39 41 1 0 0 0 0 | + | 39 41 1 0 0 0 0 |
| − | 34 37 1 0 0 0 0 | + | 34 37 1 0 0 0 0 |
| − | 29 38 1 0 0 0 0 | + | 29 38 1 0 0 0 0 |
| − | 33 43 1 0 0 0 0 | + | 33 43 1 0 0 0 0 |
| − | 25 18 1 0 0 0 0 | + | 25 18 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL2FABGM0002 | + | ID FL2FABGM0002 |
| − | FORMULA C29H36O14 | + | FORMULA C29H36O14 |
| − | EXACTMASS 608.21050586 | + | EXACTMASS 608.21050586 |
| − | AVERAGEMASS 608.5877399999999 | + | AVERAGEMASS 608.5877399999999 |
| − | SMILES C(CO)(O)(C1)C(C(OCC(C(O)5)OC(C(O)C5O)Oc(c4C)c(c(O2)c(c4O)C(=O)CC(c(c3)ccc(OC)c3)2)C)O1)O | + | SMILES C(CO)(O)(C1)C(C(OCC(C(O)5)OC(C(O)C5O)Oc(c4C)c(c(O2)c(c4O)C(=O)CC(c(c3)ccc(OC)c3)2)C)O1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
0.1919 0.1544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9046 0.5700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1951 -0.6706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9119 -1.0800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6245 -0.6643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6208 0.1607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3408 -1.0736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0535 -0.6580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0499 0.1670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3335 0.5764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7036 0.5479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4197 0.1381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1326 0.5534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1294 1.3784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4133 1.7881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7005 1.3728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3419 -1.7169 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4335 0.5155 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9119 -1.7881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5021 -1.0731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9046 1.3167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2786 1.0139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8660 0.2992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0675 0.5084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2741 0.2815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6866 0.9963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4850 0.7872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9289 1.3451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7998 1.4782 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8016 0.7768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5388 0.3493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6839 -0.1561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.7163 1.7172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.8418 0.6409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.8418 0.0894 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6577 0.1503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.8256 1.3798 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2899 1.0009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.2070 1.0009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6577 0.6409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2478 1.2596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.3198 1.6651 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.3198 1.3688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
1 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 2 1 0 0 0 0
5 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 6 1 0 0 0 0
9 11 1 6 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
1 18 1 0 0 0 0
4 19 1 0 0 0 0
3 20 1 0 0 0 0
2 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
14 33 1 0 0 0 0
40 34 1 1 0 0 0
39 34 1 1 0 0 0
38 40 1 1 0 0 0
34 35 1 0 0 0 0
40 36 1 0 0 0 0
41 38 1 0 0 0 0
37 42 1 0 0 0 0
39 41 1 0 0 0 0
34 37 1 0 0 0 0
29 38 1 0 0 0 0
33 43 1 0 0 0 0
25 18 1 0 0 0 0
S SKP 5
ID FL2FABGM0002
FORMULA C29H36O14
EXACTMASS 608.21050586
AVERAGEMASS 608.5877399999999
SMILES C(CO)(O)(C1)C(C(OCC(C(O)5)OC(C(O)C5O)Oc(c4C)c(c(O2)c(c4O)C(=O)CC(c(c3)ccc(OC)c3)2)C)O1)O
M END