Mol:FL2FABGS0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.5068 -0.7467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5068 -0.7467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5068 -1.6406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5068 -1.6406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7327 -2.0874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7327 -2.0874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0414 -1.6406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0414 -1.6406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0414 -0.7467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0414 -0.7467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7327 -0.2998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7327 -0.2998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8154 -2.0874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8154 -2.0874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5894 -1.6406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5894 -1.6406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5894 -0.7467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5894 -0.7467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8154 -0.2998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8154 -0.2998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3632 -0.2999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3632 -0.2999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1522 -0.7555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1522 -0.7555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9411 -0.2999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9411 -0.2999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9411 0.6109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9411 0.6109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1522 1.0665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1522 1.0665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3632 0.6109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3632 0.6109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8154 -2.8441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8154 -2.8441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2809 -0.2998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2809 -0.2998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7327 -2.9810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7327 -2.9810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9418 -0.2969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9418 -0.2969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4296 -0.9731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4296 -0.9731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6919 -0.6862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6919 -0.6862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9802 -0.6786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9802 -0.6786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4974 -0.1612 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4974 -0.1612 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1427 -0.5018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1427 -0.5018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5679 -0.0578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5679 -0.0578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5550 -0.5397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5550 -0.5397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1351 -0.9632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1351 -0.9632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0889 -1.1963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0889 -1.1963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2743 1.7475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2743 1.7475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0878 1.0514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0878 1.0514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7377 1.6816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7377 1.6816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1048 2.0345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1048 2.0345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2912 2.7306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2912 2.7306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6413 2.1006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6413 2.1006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4817 0.5768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4817 0.5768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6039 2.9810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6039 2.9810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6008 2.4761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6008 2.4761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7292 2.8929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7292 2.8929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7550 2.2271 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7550 2.2271 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7607 1.0841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7607 1.0841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7550 0.5102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7550 0.5102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 18 1 1 0 0 0 0 | + | 18 1 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 20 27 1 0 0 0 0 | + | 20 27 1 0 0 0 0 |
| − | 21 28 1 0 0 0 0 | + | 21 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 31 30 1 1 0 0 0 | + | 31 30 1 1 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 30 1 1 0 0 0 | + | 35 30 1 1 0 0 0 |
| − | 31 36 1 0 0 0 0 | + | 31 36 1 0 0 0 0 |
| − | 36 26 1 0 0 0 0 | + | 36 26 1 0 0 0 0 |
| − | 34 37 1 0 0 0 0 | + | 34 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 30 40 1 0 0 0 0 | + | 30 40 1 0 0 0 0 |
| − | 23 18 1 0 0 0 0 | + | 23 18 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 14 41 1 0 0 0 0 | + | 14 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 46 -0.8196 -0.4732 | + | M SBV 1 46 -0.8196 -0.4732 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL2FABGS0005 | + | ID FL2FABGS0005 |
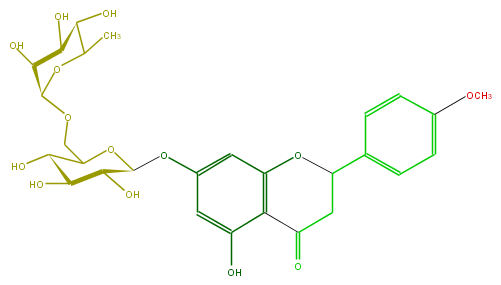
| − | FORMULA C28H34O14 | + | FORMULA C28H34O14 |
| − | EXACTMASS 594.194855796 | + | EXACTMASS 594.194855796 |
| − | AVERAGEMASS 594.56116 | + | AVERAGEMASS 594.56116 |
| − | SMILES OC(C(O)4)C(COC(O5)C(C(C(O)C(C)5)O)O)OC(C(O)4)Oc(c1)cc(c(C(=O)2)c1OC(c(c3)ccc(c3)OC)C2)O | + | SMILES OC(C(O)4)C(COC(O5)C(C(C(O)C(C)5)O)O)OC(C(O)4)Oc(c1)cc(c(C(=O)2)c1OC(c(c3)ccc(c3)OC)C2)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-1.5068 -0.7467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5068 -1.6406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7327 -2.0874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0414 -1.6406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0414 -0.7467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7327 -0.2998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8154 -2.0874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5894 -1.6406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5894 -0.7467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8154 -0.2998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3632 -0.2999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1522 -0.7555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9411 -0.2999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9411 0.6109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1522 1.0665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3632 0.6109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8154 -2.8441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2809 -0.2998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7327 -2.9810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9418 -0.2969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4296 -0.9731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6919 -0.6862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9802 -0.6786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4974 -0.1612 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1427 -0.5018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5679 -0.0578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5550 -0.5397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1351 -0.9632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0889 -1.1963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2743 1.7475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0878 1.0514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7377 1.6816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1048 2.0345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2912 2.7306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6413 2.1006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4817 0.5768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6039 2.9810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6008 2.4761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7292 2.8929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.7550 2.2271 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7607 1.0841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.7550 0.5102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
18 1 1 0 0 0 0
3 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
25 26 1 0 0 0 0
20 27 1 0 0 0 0
21 28 1 0 0 0 0
22 29 1 0 0 0 0
31 30 1 1 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
35 30 1 1 0 0 0
31 36 1 0 0 0 0
36 26 1 0 0 0 0
34 37 1 0 0 0 0
33 38 1 0 0 0 0
35 39 1 0 0 0 0
30 40 1 0 0 0 0
23 18 1 0 0 0 0
41 42 1 0 0 0 0
14 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 46
M SMT 1 OCH3
M SBV 1 46 -0.8196 -0.4732
S SKP 5
ID FL2FABGS0005
FORMULA C28H34O14
EXACTMASS 594.194855796
AVERAGEMASS 594.56116
SMILES OC(C(O)4)C(COC(O5)C(C(C(O)C(C)5)O)O)OC(C(O)4)Oc(c1)cc(c(C(=O)2)c1OC(c(c3)ccc(c3)OC)C2)O
M END