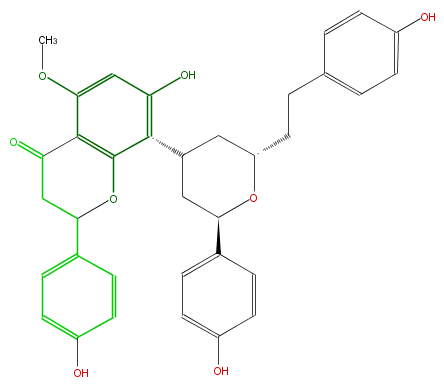
Mol:FL2FBANC0009
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 48 0 0 0 0 0 0 0 0999 V2000 | + | 43 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.6940 -0.8282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6940 -0.8282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0202 -0.4153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0202 -0.4153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6940 -1.6532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6940 -1.6532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0211 -2.0653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0211 -2.0653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7354 -1.6524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7354 -1.6524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7349 -0.8274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7349 -0.8274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4501 -2.0644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4501 -2.0644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1643 -1.6515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1643 -1.6515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1638 -0.8265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1638 -0.8265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4491 -0.4145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4491 -0.4145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7927 -0.4630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7927 -0.4630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5074 -0.8751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5074 -0.8751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2217 -0.4622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2217 -0.4622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2212 0.3628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2212 0.3628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5065 0.7749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5065 0.7749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7923 0.3620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7923 0.3620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4504 -2.7174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4504 -2.7174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0216 -2.8771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0216 -2.8771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4088 -0.4161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4088 -0.4161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8211 0.7092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8211 0.7092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5972 -3.2343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5972 -3.2343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0202 0.3555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0202 0.3555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7347 0.7680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7347 0.7680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7347 1.5930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7347 1.5930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0202 2.0055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0202 2.0055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6943 1.5930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6943 1.5930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6943 0.7680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6943 0.7680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3663 1.9577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3663 1.9577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0808 1.5452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0808 1.5452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7952 1.9577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7952 1.9577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7952 2.7827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7952 2.7827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0808 3.1952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0808 3.1952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3663 2.7827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3663 2.7827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3768 3.1185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3768 3.1185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3862 1.9925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3862 1.9925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0982 1.5814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0982 1.5814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8179 1.9968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8179 1.9968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8179 2.8218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8179 2.8218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5323 3.2343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5323 3.2343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2468 2.8218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2468 2.8218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2468 1.9968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2468 1.9968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5323 1.5843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5323 1.5843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8211 3.1534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8211 3.1534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 1 3 1 0 0 0 0 | + | 1 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 2 1 0 0 0 0 | + | 6 2 1 0 0 0 0 |
| − | 5 7 1 0 0 0 0 | + | 5 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 6 1 0 0 0 0 | + | 10 6 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 4 18 1 0 0 0 0 | + | 4 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 18 21 1 0 0 0 0 | + | 18 21 1 0 0 0 0 |
| − | 2 22 1 6 0 0 0 | + | 2 22 1 6 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 24 28 1 1 0 0 0 | + | 24 28 1 1 0 0 0 |
| − | 28 29 2 0 0 0 0 | + | 28 29 2 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 2 0 0 0 0 | + | 30 31 2 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 33 28 1 0 0 0 0 | + | 33 28 1 0 0 0 0 |
| − | 31 34 1 0 0 0 0 | + | 31 34 1 0 0 0 0 |
| − | 26 35 1 6 0 0 0 | + | 26 35 1 6 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 42 37 1 0 0 0 0 | + | 42 37 1 0 0 0 0 |
| − | 40 43 1 0 0 0 0 | + | 40 43 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL2FBANC0009 | + | ID FL2FBANC0009 |
| − | KNApSAcK_ID C00014293 | + | KNApSAcK_ID C00014293 |
| − | NAME Epicalyxin G | + | NAME Epicalyxin G |
| − | CAS_RN 205313-11-7 | + | CAS_RN 205313-11-7 |
| − | FORMULA C35H34O8 | + | FORMULA C35H34O8 |
| − | EXACTMASS 582.225368064 | + | EXACTMASS 582.225368064 |
| − | AVERAGEMASS 582.63966 | + | AVERAGEMASS 582.63966 |
| − | SMILES c(c6)(ccc(O)c6)C(C2)Oc(c(C(C3)CC(CCc(c5)ccc(c5)O)OC3c(c4)ccc(O)c4)1)c(C2=O)c(cc1O)OC | + | SMILES c(c6)(ccc(O)c6)C(C2)Oc(c(C(C3)CC(CCc(c5)ccc(c5)O)OC3c(c4)ccc(O)c4)1)c(C2=O)c(cc1O)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 48 0 0 0 0 0 0 0 0999 V2000
-0.6940 -0.8282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0202 -0.4153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6940 -1.6532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0211 -2.0653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7354 -1.6524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7349 -0.8274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4501 -2.0644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1643 -1.6515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1638 -0.8265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4491 -0.4145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7927 -0.4630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5074 -0.8751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2217 -0.4622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2212 0.3628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5065 0.7749 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7923 0.3620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4504 -2.7174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0216 -2.8771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4088 -0.4161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8211 0.7092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5972 -3.2343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0202 0.3555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7347 0.7680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7347 1.5930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0202 2.0055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6943 1.5930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6943 0.7680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3663 1.9577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0808 1.5452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7952 1.9577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7952 2.7827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0808 3.1952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3663 2.7827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3768 3.1185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3862 1.9925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0982 1.5814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8179 1.9968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8179 2.8218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5323 3.2343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2468 2.8218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2468 1.9968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5323 1.5843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8211 3.1534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
1 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 2 1 0 0 0 0
5 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 6 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
4 18 1 0 0 0 0
1 19 1 0 0 0 0
14 20 1 0 0 0 0
18 21 1 0 0 0 0
2 22 1 6 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
24 28 1 1 0 0 0
28 29 2 0 0 0 0
29 30 1 0 0 0 0
30 31 2 0 0 0 0
31 32 1 0 0 0 0
32 33 2 0 0 0 0
33 28 1 0 0 0 0
31 34 1 0 0 0 0
26 35 1 6 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 41 1 0 0 0 0
41 42 2 0 0 0 0
42 37 1 0 0 0 0
40 43 1 0 0 0 0
S SKP 8
ID FL2FBANC0009
KNApSAcK_ID C00014293
NAME Epicalyxin G
CAS_RN 205313-11-7
FORMULA C35H34O8
EXACTMASS 582.225368064
AVERAGEMASS 582.63966
SMILES c(c6)(ccc(O)c6)C(C2)Oc(c(C(C3)CC(CCc(c5)ccc(c5)O)OC3c(c4)ccc(O)c4)1)c(C2=O)c(cc1O)OC
M END