Mol:FL3FAACS0048
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 40 44 0 0 0 0 0 0 0 0999 V2000 | + | 40 44 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.5315 0.6048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5315 0.6048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5315 -0.2202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5315 -0.2202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1829 -0.6327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1829 -0.6327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8974 -0.2202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8974 -0.2202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8974 0.6048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8974 0.6048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1829 1.0173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1829 1.0173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6118 -0.6327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6118 -0.6327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3262 -0.2202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3262 -0.2202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3262 0.6048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3262 0.6048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6118 1.0173 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6118 1.0173 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6118 -1.2759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6118 -1.2759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1829 -1.4574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1829 -1.4574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1992 1.1495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1992 1.1495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9522 0.7148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9522 0.7148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7050 1.1495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7050 1.1495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7050 2.0188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7050 2.0188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9522 2.4534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9522 2.4534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1992 2.0188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1992 2.0188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4573 2.4532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4573 2.4532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8107 -0.5342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8107 -0.5342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1485 -1.2759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1485 -1.2759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4862 -0.8786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4862 -0.8786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5857 -0.9845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5857 -0.9845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2214 -0.4018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2214 -0.4018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9366 -0.7461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9366 -0.7461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3650 -0.8543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3650 -0.8543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8778 -1.2759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8778 -1.2759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2457 1.0170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2457 1.0170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8764 -1.4326 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8764 -1.4326 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9499 -1.6899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9499 -1.6899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2877 -2.4315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2877 -2.4315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6254 -2.0343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6254 -2.0343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7249 -2.1403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7249 -2.1403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3606 -1.5575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3606 -1.5575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0758 -1.9018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0758 -1.9018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4573 -1.9829 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4573 -1.9829 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0749 -2.4315 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0749 -2.4315 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2063 -2.4534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2063 -2.4534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2927 -0.1294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2927 -0.1294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5184 0.2541 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5184 0.2541 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 3 12 1 0 0 0 0 | + | 3 12 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 2 23 1 0 0 0 0 | + | 2 23 1 0 0 0 0 |
| − | 1 28 1 0 0 0 0 | + | 1 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 29 1 0 0 0 0 | + | 33 29 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 25 39 1 0 0 0 0 | + | 25 39 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 44 0.3561 -0.6168 | + | M SBV 1 44 0.3561 -0.6168 |
| − | S SKP 5 | + | S SKP 5 |
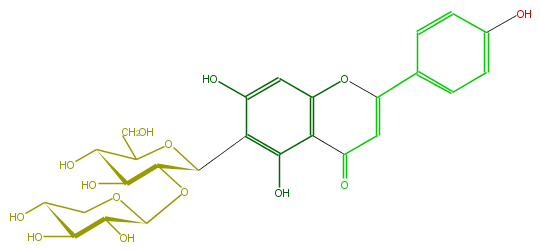
| − | ID FL3FAACS0048 | + | ID FL3FAACS0048 |
| − | FORMULA C26H28O14 | + | FORMULA C26H28O14 |
| − | EXACTMASS 564.147905604 | + | EXACTMASS 564.147905604 |
| − | AVERAGEMASS 564.49212 | + | AVERAGEMASS 564.49212 |
| − | SMILES OC(C1O)C(O)COC1OC(C(O)5)C(OC(CO)C(O)5)c(c4O)c(O)cc(c34)OC(=CC3=O)c(c2)ccc(c2)O | + | SMILES OC(C1O)C(O)COC1OC(C(O)5)C(OC(CO)C(O)5)c(c4O)c(O)cc(c34)OC(=CC3=O)c(c2)ccc(c2)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
40 44 0 0 0 0 0 0 0 0999 V2000
-0.5315 0.6048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5315 -0.2202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1829 -0.6327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8974 -0.2202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8974 0.6048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1829 1.0173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6118 -0.6327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3262 -0.2202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3262 0.6048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6118 1.0173 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6118 -1.2759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1829 -1.4574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1992 1.1495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9522 0.7148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7050 1.1495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7050 2.0188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9522 2.4534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1992 2.0188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4573 2.4532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8107 -0.5342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1485 -1.2759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4862 -0.8786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5857 -0.9845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2214 -0.4018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9366 -0.7461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3650 -0.8543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8778 -1.2759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2457 1.0170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8764 -1.4326 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9499 -1.6899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2877 -2.4315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6254 -2.0343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7249 -2.1403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3606 -1.5575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0758 -1.9018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4573 -1.9829 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0749 -2.4315 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2063 -2.4534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2927 -0.1294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5184 0.2541 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
3 12 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
9 13 1 0 0 0 0
16 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
2 23 1 0 0 0 0
1 28 1 0 0 0 0
22 29 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 29 1 0 0 0 0
39 40 1 0 0 0 0
25 39 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 44
M SMT 1 ^ CH2OH
M SBV 1 44 0.3561 -0.6168
S SKP 5
ID FL3FAACS0048
FORMULA C26H28O14
EXACTMASS 564.147905604
AVERAGEMASS 564.49212
SMILES OC(C1O)C(O)COC1OC(C(O)5)C(OC(CO)C(O)5)c(c4O)c(O)cc(c34)OC(=CC3=O)c(c2)ccc(c2)O
M END