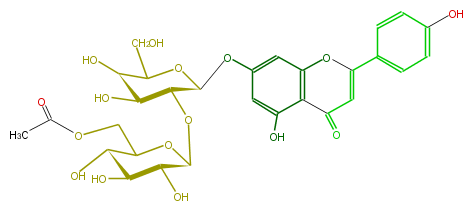
Mol:FL3FAAGS0050
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 45 49 0 0 0 0 0 0 0 0999 V2000 | + | 45 49 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.2809 0.6716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2809 0.6716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2809 0.0986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2809 0.0986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7772 -0.1879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7772 -0.1879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2734 0.0986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2734 0.0986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2734 0.6716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2734 0.6716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7772 0.9581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7772 0.9581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7696 -0.1879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7696 -0.1879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2658 0.0986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2658 0.0986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2658 0.6716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2658 0.6716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7696 0.9581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7696 0.9581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9584 -0.5926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9584 -0.5926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7619 0.9580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7619 0.9580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2676 0.6660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2676 0.6660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7733 0.9580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7733 0.9580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7733 1.5420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7733 1.5420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2676 1.8339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2676 1.8339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7619 1.5420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7619 1.5420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2151 0.9580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2151 0.9580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2789 1.8339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2789 1.8339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6997 -0.6280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6997 -0.6280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4176 0.6282 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -2.4176 0.6282 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -1.9860 0.0586 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.9860 0.0586 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.3645 0.3002 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.3645 0.3002 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.7648 0.3067 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.7648 0.3067 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.2006 0.7426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2006 0.7426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8354 0.5146 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -1.8354 0.5146 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -2.8999 0.9068 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8999 0.9068 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6542 0.1432 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6542 0.1432 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0084 -0.2975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0084 -0.2975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2855 -1.4016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2855 -1.4016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6247 -0.9082 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -2.6247 -0.9082 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.1931 -1.4779 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.1931 -1.4779 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.5717 -1.2362 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.5717 -1.2362 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.9720 -1.2297 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.9720 -1.2297 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.4078 -0.7938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4078 -0.7938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0426 -1.0218 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.0426 -1.0218 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -2.7164 -1.4779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7164 -1.4779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2156 -1.8339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2156 -1.8339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3994 -0.4037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3994 -0.4037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2072 -0.6202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2072 -0.6202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7737 -0.2931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7737 -0.2931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2789 -0.5848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2789 -0.5848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9430 0.0699 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9430 0.0699 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0738 1.2447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0738 1.2447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4058 1.9888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4058 1.9888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 20 3 1 0 0 0 0 | + | 20 3 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 24 18 1 0 0 0 0 | + | 24 18 1 0 0 0 0 |
| − | 36 39 1 0 0 0 0 | + | 36 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 41 43 2 0 0 0 0 | + | 41 43 2 0 0 0 0 |
| − | 26 44 1 0 0 0 0 | + | 26 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 44 45 | + | M SAL 1 2 44 45 |
| − | M SBL 1 1 48 | + | M SBL 1 1 48 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 48 -2.0738 1.2447 | + | M SVB 1 48 -2.0738 1.2447 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FAAGS0050 | + | ID FL3FAAGS0050 |
| − | KNApSAcK_ID C00004190 | + | KNApSAcK_ID C00004190 |
| − | NAME Apigenin 7-(6'''-acetylallosyl-(1->2)glucoside) | + | NAME Apigenin 7-(6'''-acetylallosyl-(1->2)glucoside) |
| − | CAS_RN 135626-72-1 | + | CAS_RN 135626-72-1 |
| − | FORMULA C29H32O16 | + | FORMULA C29H32O16 |
| − | EXACTMASS 636.1690349759999 | + | EXACTMASS 636.1690349759999 |
| − | AVERAGEMASS 636.5547799999999 | + | AVERAGEMASS 636.5547799999999 |
| − | SMILES c(c1)(O[C@H](O5)[C@H]([C@H]([C@H](C5CO)O)O)O[C@H](O4)[C@H]([C@H]([C@@H](O)C4COC(C)=O)O)O)cc(O)c(C(=O)2)c1OC(c(c3)ccc(O)c3)=C2 | + | SMILES c(c1)(O[C@H](O5)[C@H]([C@H]([C@H](C5CO)O)O)O[C@H](O4)[C@H]([C@H]([C@@H](O)C4COC(C)=O)O)O)cc(O)c(C(=O)2)c1OC(c(c3)ccc(O)c3)=C2 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
45 49 0 0 0 0 0 0 0 0999 V2000
0.2809 0.6716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2809 0.0986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7772 -0.1879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2734 0.0986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2734 0.6716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7772 0.9581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7696 -0.1879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2658 0.0986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2658 0.6716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7696 0.9581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9584 -0.5926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7619 0.9580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2676 0.6660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7733 0.9580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7733 1.5420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2676 1.8339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7619 1.5420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2151 0.9580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2789 1.8339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6997 -0.6280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4176 0.6282 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-1.9860 0.0586 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.3645 0.3002 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.7648 0.3067 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.2006 0.7426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8354 0.5146 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-2.8999 0.9068 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6542 0.1432 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0084 -0.2975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2855 -1.4016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6247 -0.9082 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.1931 -1.4779 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.5717 -1.2362 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.9720 -1.2297 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.4078 -0.7938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0426 -1.0218 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-2.7164 -1.4779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2156 -1.8339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3994 -0.4037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2072 -0.6202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7737 -0.2931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2789 -0.5848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9430 0.0699 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0738 1.2447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4058 1.9888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
15 19 1 0 0 0 0
20 3 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
32 37 1 0 0 0 0
33 38 1 0 0 0 0
30 31 1 0 0 0 0
34 29 1 0 0 0 0
24 18 1 0 0 0 0
36 39 1 0 0 0 0
39 40 1 0 0 0 0
40 41 1 0 0 0 0
41 42 1 0 0 0 0
41 43 2 0 0 0 0
26 44 1 0 0 0 0
44 45 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 44 45
M SBL 1 1 48
M SMT 1 CH2OH
M SVB 1 48 -2.0738 1.2447
S SKP 8
ID FL3FAAGS0050
KNApSAcK_ID C00004190
NAME Apigenin 7-(6'''-acetylallosyl-(1->2)glucoside)
CAS_RN 135626-72-1
FORMULA C29H32O16
EXACTMASS 636.1690349759999
AVERAGEMASS 636.5547799999999
SMILES c(c1)(O[C@H](O5)[C@H]([C@H]([C@H](C5CO)O)O)O[C@H](O4)[C@H]([C@H]([C@@H](O)C4COC(C)=O)O)O)cc(O)c(C(=O)2)c1OC(c(c3)ccc(O)c3)=C2
M END