Mol:FL3FAAGS0062
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 55 60 0 0 0 0 0 0 0 0999 V2000 | + | 55 60 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.9152 0.1136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9152 0.1136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9152 -0.7114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9152 -0.7114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2007 -1.1239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2007 -1.1239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5138 -0.7114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5138 -0.7114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5138 0.1136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5138 0.1136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2007 0.5261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2007 0.5261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2281 -1.1239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2281 -1.1239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9426 -0.7114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9426 -0.7114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9426 0.1136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9426 0.1136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2281 0.5261 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2281 0.5261 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6571 0.5261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6571 0.5261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3715 0.1136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3715 0.1136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0860 0.5261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0860 0.5261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0860 1.3511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0860 1.3511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3715 1.7636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3715 1.7636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6571 1.3511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6571 1.3511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2281 -1.9488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2281 -1.9488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2007 -1.9488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2007 -1.9488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6296 0.5261 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6296 0.5261 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8322 1.7818 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8322 1.7818 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2447 -1.2698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2447 -1.2698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0334 -1.0270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0334 -1.0270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0076 -0.2021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0076 -0.2021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4058 0.5208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4058 0.5208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6171 0.2781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6171 0.2781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6428 -0.5469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6428 -0.5469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1123 -0.5515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1123 -0.5515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2447 -1.8957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2447 -1.8957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7149 -1.5787 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7149 -1.5787 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8721 0.2970 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8721 0.2970 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6706 -0.1099 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6706 -0.1099 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7590 -1.1635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7590 -1.1635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3769 1.0917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3769 1.0917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9643 0.3771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9643 0.3771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1660 0.5861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1660 0.5861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3725 0.3594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3725 0.3594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7851 1.0740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7851 1.0740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5835 0.8649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5835 0.8649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8709 1.4235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8709 1.4235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9589 0.7557 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9589 0.7557 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5983 0.5469 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5983 0.5469 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5715 -0.0202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5715 -0.0202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5676 1.9488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5676 1.9488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4312 1.4235 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4312 1.4235 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3338 -0.5828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3338 -0.5828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7077 -1.1204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7077 -1.1204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0238 -0.6586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0238 -0.6586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1999 -0.6098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1999 -0.6098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8260 -0.0721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8260 -0.0721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5100 -0.5338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5100 -0.5338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0052 -0.1440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0052 -0.1440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6271 -0.1440 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6271 -0.1440 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8721 -0.8936 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8721 -0.8936 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4610 -1.1204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4610 -1.1204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2540 -1.1030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2540 -1.1030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 21 28 1 0 0 0 0 | + | 21 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 27 31 2 0 0 0 0 | + | 27 31 2 0 0 0 0 |
| − | 27 32 1 0 0 0 0 | + | 27 32 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 33 40 1 0 0 0 0 | + | 33 40 1 0 0 0 0 |
| − | 34 41 1 0 0 0 0 | + | 34 41 1 0 0 0 0 |
| − | 35 42 1 0 0 0 0 | + | 35 42 1 0 0 0 0 |
| − | 39 43 2 0 0 0 0 | + | 39 43 2 0 0 0 0 |
| − | 39 44 1 0 0 0 0 | + | 39 44 1 0 0 0 0 |
| − | 24 20 1 0 0 0 0 | + | 24 20 1 0 0 0 0 |
| − | 19 36 1 0 0 0 0 | + | 19 36 1 0 0 0 0 |
| − | 45 46 1 1 0 0 0 | + | 45 46 1 1 0 0 0 |
| − | 46 47 1 1 0 0 0 | + | 46 47 1 1 0 0 0 |
| − | 48 47 1 1 0 0 0 | + | 48 47 1 1 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 45 53 1 0 0 0 0 | + | 45 53 1 0 0 0 0 |
| − | 46 54 1 0 0 0 0 | + | 46 54 1 0 0 0 0 |
| − | 47 55 1 0 0 0 0 | + | 47 55 1 0 0 0 0 |
| − | 48 42 1 0 0 0 0 | + | 48 42 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FAAGS0062 | + | ID FL3FAAGS0062 |
| − | FORMULA C33H36O22 | + | FORMULA C33H36O22 |
| − | EXACTMASS 784.169822836 | + | EXACTMASS 784.169822836 |
| − | AVERAGEMASS 784.62574 | + | AVERAGEMASS 784.62574 |
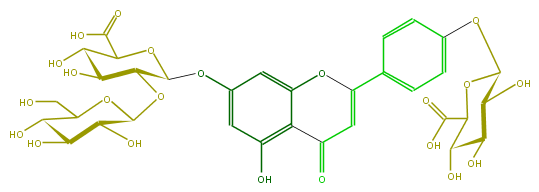
| − | SMILES C(C(C(O)1)OC(Oc(c2)ccc(C(=C6)Oc(c(C(=O)6)5)cc(cc5O)OC(O4)C(C(O)C(C4C(O)=O)O)OC(C3O)OC(CO)C(O)C(O)3)c2)C(O)C(O)1)(O)=O | + | SMILES C(C(C(O)1)OC(Oc(c2)ccc(C(=C6)Oc(c(C(=O)6)5)cc(cc5O)OC(O4)C(C(O)C(C4C(O)=O)O)OC(C3O)OC(CO)C(O)C(O)3)c2)C(O)C(O)1)(O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
55 60 0 0 0 0 0 0 0 0999 V2000
-0.9152 0.1136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9152 -0.7114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2007 -1.1239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5138 -0.7114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5138 0.1136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2007 0.5261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2281 -1.1239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9426 -0.7114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9426 0.1136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2281 0.5261 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6571 0.5261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3715 0.1136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0860 0.5261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0860 1.3511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3715 1.7636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6571 1.3511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2281 -1.9488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2007 -1.9488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6296 0.5261 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8322 1.7818 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2447 -1.2698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0334 -1.0270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0076 -0.2021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4058 0.5208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6171 0.2781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6428 -0.5469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1123 -0.5515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2447 -1.8957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7149 -1.5787 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.8721 0.2970 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6706 -0.1099 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7590 -1.1635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3769 1.0917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9643 0.3771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1660 0.5861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3725 0.3594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7851 1.0740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5835 0.8649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8709 1.4235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9589 0.7557 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5983 0.5469 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5715 -0.0202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5676 1.9488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4312 1.4235 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.3338 -0.5828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7077 -1.1204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0238 -0.6586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1999 -0.6098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8260 -0.0721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5100 -0.5338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0052 -0.1440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.6271 -0.1440 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.8721 -0.8936 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.4610 -1.1204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2540 -1.1030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
3 18 1 0 0 0 0
1 19 1 0 0 0 0
14 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
26 27 1 0 0 0 0
21 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
27 31 2 0 0 0 0
27 32 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
38 39 1 0 0 0 0
33 40 1 0 0 0 0
34 41 1 0 0 0 0
35 42 1 0 0 0 0
39 43 2 0 0 0 0
39 44 1 0 0 0 0
24 20 1 0 0 0 0
19 36 1 0 0 0 0
45 46 1 1 0 0 0
46 47 1 1 0 0 0
48 47 1 1 0 0 0
48 49 1 0 0 0 0
49 50 1 0 0 0 0
50 45 1 0 0 0 0
50 51 1 0 0 0 0
51 52 1 0 0 0 0
45 53 1 0 0 0 0
46 54 1 0 0 0 0
47 55 1 0 0 0 0
48 42 1 0 0 0 0
S SKP 5
ID FL3FAAGS0062
FORMULA C33H36O22
EXACTMASS 784.169822836
AVERAGEMASS 784.62574
SMILES C(C(C(O)1)OC(Oc(c2)ccc(C(=C6)Oc(c(C(=O)6)5)cc(cc5O)OC(O4)C(C(O)C(C4C(O)=O)O)OC(C3O)OC(CO)C(O)C(O)3)c2)C(O)C(O)1)(O)=O
M END