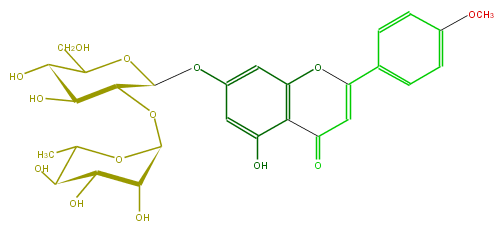
Mol:FL3FABGS0006
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.9932 0.7036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9932 0.7036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9932 -0.1057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9932 -0.1057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2922 -0.5105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2922 -0.5105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4087 -0.1057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4087 -0.1057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4087 0.7036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4087 0.7036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2922 1.1083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2922 1.1083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1096 -0.5105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1096 -0.5105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8106 -0.1057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8106 -0.1057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8106 0.7036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8106 0.7036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1096 1.1083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1096 1.1083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1096 -1.1414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1096 -1.1414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5113 1.1081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5113 1.1081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2256 0.6957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2256 0.6957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9400 1.1081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9400 1.1081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9400 1.9331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9400 1.9331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2256 2.3455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2256 2.3455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5113 1.9331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5113 1.9331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6939 1.1081 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6939 1.1081 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2922 -1.1419 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2922 -1.1419 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1110 1.1489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1110 1.1489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4701 0.3026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4701 0.3026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5470 0.6617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5470 0.6617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6563 0.6712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6563 0.6712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3036 1.3186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3036 1.3186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2464 0.9801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2464 0.9801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7645 0.9193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7645 0.9193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3030 0.3970 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3030 0.3970 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6882 0.0213 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6882 0.0213 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4529 -0.7374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4529 -0.7374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9567 -1.6102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9567 -1.6102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9878 -1.3332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9878 -1.3332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0129 -1.6102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0129 -1.6102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5089 -0.7374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5089 -0.7374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4780 -1.0142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4780 -1.0142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5441 -2.0092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5441 -2.0092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3425 -1.2244 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3425 -1.2244 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0445 -0.8959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0445 -0.8959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0129 -2.3455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0129 -2.3455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8247 1.5806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8247 1.5806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9365 1.0678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9365 1.0678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6542 2.3454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6542 2.3454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7645 1.7044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7645 1.7044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 19 3 1 0 0 0 0 | + | 19 3 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 33 28 1 0 0 0 0 | + | 33 28 1 0 0 0 0 |
| − | 31 35 1 0 0 0 0 | + | 31 35 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 29 37 1 0 0 0 0 | + | 29 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 23 18 1 0 0 0 0 | + | 23 18 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 25 39 1 0 0 0 0 | + | 25 39 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 15 41 1 0 0 0 0 | + | 15 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 44 0.5783 -0.6006 | + | M SBV 1 44 0.5783 -0.6006 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 41 42 | + | M SAL 2 2 41 42 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 46 -0.7142 -0.4123 | + | M SBV 2 46 -0.7142 -0.4123 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FABGS0006 | + | ID FL3FABGS0006 |
| − | FORMULA C28H32O14 | + | FORMULA C28H32O14 |
| − | EXACTMASS 592.179205732 | + | EXACTMASS 592.179205732 |
| − | AVERAGEMASS 592.54528 | + | AVERAGEMASS 592.54528 |
| − | SMILES O(C)c(c5)ccc(c5)C(=C4)Oc(c3C4=O)cc(cc3O)OC(O2)C(C(C(O)C2CO)O)OC(O1)C(O)C(C(C1C)O)O | + | SMILES O(C)c(c5)ccc(c5)C(=C4)Oc(c3C4=O)cc(cc3O)OC(O2)C(C(C(O)C2CO)O)OC(O1)C(O)C(C(C1C)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-0.9932 0.7036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9932 -0.1057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2922 -0.5105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4087 -0.1057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4087 0.7036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2922 1.1083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1096 -0.5105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8106 -0.1057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8106 0.7036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1096 1.1083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1096 -1.1414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5113 1.1081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2256 0.6957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9400 1.1081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9400 1.9331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2256 2.3455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5113 1.9331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6939 1.1081 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2922 -1.1419 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1110 1.1489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4701 0.3026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5470 0.6617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6563 0.6712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3036 1.3186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2464 0.9801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7645 0.9193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.3030 0.3970 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6882 0.0213 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4529 -0.7374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9567 -1.6102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9878 -1.3332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0129 -1.6102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5089 -0.7374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4780 -1.0142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5441 -2.0092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.3425 -1.2244 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0445 -0.8959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0129 -2.3455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8247 1.5806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9365 1.0678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6542 2.3454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.7645 1.7044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
19 3 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 29 1 0 0 0 0
33 28 1 0 0 0 0
31 35 1 0 0 0 0
30 36 1 0 0 0 0
29 37 1 0 0 0 0
32 38 1 0 0 0 0
23 18 1 0 0 0 0
39 40 1 0 0 0 0
25 39 1 0 0 0 0
41 42 1 0 0 0 0
15 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 44
M SMT 1 ^CH2OH
M SBV 1 44 0.5783 -0.6006
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 41 42
M SBL 2 1 46
M SMT 2 OCH3
M SBV 2 46 -0.7142 -0.4123
S SKP 5
ID FL3FABGS0006
FORMULA C28H32O14
EXACTMASS 592.179205732
AVERAGEMASS 592.54528
SMILES O(C)c(c5)ccc(c5)C(=C4)Oc(c3C4=O)cc(cc3O)OC(O2)C(C(C(O)C2CO)O)OC(O1)C(O)C(C(C1C)O)O
M END