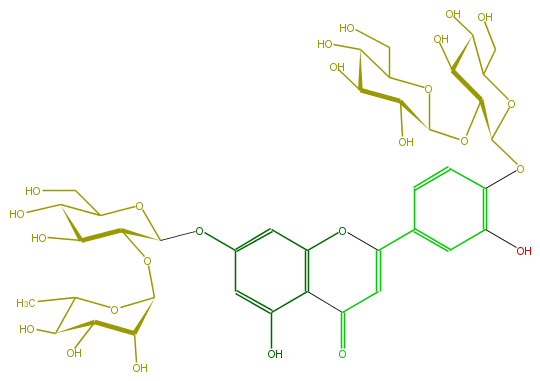
Mol:FL3FACGS0018
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 64 70 0 0 0 0 0 0 0 0999 V2000 | + | 64 70 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.7264 -1.2106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7264 -1.2106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7264 -2.0356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7264 -2.0356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4409 -2.4481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4409 -2.4481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1553 -2.0356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1553 -2.0356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1553 -1.2106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1553 -1.2106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4409 -0.7981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4409 -0.7981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0119 -0.7981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0119 -0.7981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7025 -1.2106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7025 -1.2106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7025 -2.0356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7025 -2.0356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0119 -2.4481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0119 -2.4481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8698 -0.7981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8698 -0.7981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4409 -3.2731 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4409 -3.2731 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0119 -3.2731 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0119 -3.2731 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5843 -1.2106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5843 -1.2106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2988 -0.7981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2988 -0.7981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2988 0.0269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2988 0.0269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5843 0.4394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5843 0.4394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8698 0.0269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8698 0.0269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0132 -1.2106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0132 -1.2106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7918 2.8308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7918 2.8308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7918 2.0058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7918 2.0058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5875 1.7878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5875 1.7878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1699 1.2101 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1699 1.2101 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1700 2.0351 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1700 2.0351 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3743 2.2532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3743 2.2532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6507 0.9821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6507 0.9821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1613 2.9398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1613 2.9398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2718 2.4847 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2718 2.4847 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3757 2.8706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3757 2.8706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6067 3.2683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6067 3.2683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9178 -2.1564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9178 -2.1564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3303 -2.8709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3303 -2.8709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5371 -2.6441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5371 -2.6441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7390 -2.8709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7390 -2.8709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3264 -2.1564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3264 -2.1564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1197 -2.3830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1197 -2.3830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6964 -3.5529 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6964 -3.5529 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0101 -3.2568 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0101 -3.2568 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8097 -2.7663 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8097 -2.7663 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7891 -2.2506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7891 -2.2506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2177 -0.2971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2177 -0.2971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8052 -1.0116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8052 -1.0116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0071 -0.8025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0071 -0.8025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2139 -1.0116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2139 -1.0116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6263 -0.2971 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6263 -0.2971 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4245 -0.5062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4245 -0.5062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4694 -1.4153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4694 -1.4153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4170 -0.7981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4170 -0.7981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0132 -0.4709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0132 -0.4709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5801 -0.9439 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5801 -0.9439 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9181 0.0006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9181 0.0006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6944 -0.0127 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6944 -0.0127 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0467 3.1153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0467 3.1153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6342 2.4008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6342 2.4008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2143 1.8143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2143 1.8143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4299 1.0228 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4299 1.0228 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8424 1.7372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8424 1.7372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2623 2.3238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2623 2.3238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8857 0.9953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8857 0.9953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0132 0.4394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0132 0.4394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5884 3.5529 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5884 3.5529 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3396 3.0520 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3396 3.0520 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4919 2.8293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4919 2.8293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2385 3.4805 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2385 3.4805 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 1 7 1 0 0 0 0 | + | 1 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 2 1 0 0 0 0 | + | 10 2 1 0 0 0 0 |
| − | 5 11 1 0 0 0 0 | + | 5 11 1 0 0 0 0 |
| − | 3 12 2 0 0 0 0 | + | 3 12 2 0 0 0 0 |
| − | 10 13 1 0 0 0 0 | + | 10 13 1 0 0 0 0 |
| − | 8 48 1 0 0 0 0 | + | 8 48 1 0 0 0 0 |
| − | 11 14 2 0 0 0 0 | + | 11 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 11 1 0 0 0 0 | + | 18 11 1 0 0 0 0 |
| − | 16 60 1 0 0 0 0 | + | 16 60 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 22 26 1 0 0 0 0 | + | 22 26 1 0 0 0 0 |
| − | 23 59 1 0 0 0 0 | + | 23 59 1 0 0 0 0 |
| − | 20 27 1 0 0 0 0 | + | 20 27 1 0 0 0 0 |
| − | 21 28 1 0 0 0 0 | + | 21 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 35 47 1 0 0 0 0 | + | 35 47 1 0 0 0 0 |
| − | 34 37 1 0 0 0 0 | + | 34 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 32 39 1 0 0 0 0 | + | 32 39 1 0 0 0 0 |
| − | 31 40 1 0 0 0 0 | + | 31 40 1 0 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 44 43 1 1 0 0 0 | + | 44 43 1 1 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 41 1 0 0 0 0 | + | 46 41 1 0 0 0 0 |
| − | 43 47 1 0 0 0 0 | + | 43 47 1 0 0 0 0 |
| − | 44 48 1 0 0 0 0 | + | 44 48 1 0 0 0 0 |
| − | 41 49 1 0 0 0 0 | + | 41 49 1 0 0 0 0 |
| − | 42 50 1 0 0 0 0 | + | 42 50 1 0 0 0 0 |
| − | 46 51 1 0 0 0 0 | + | 46 51 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 53 54 1 1 0 0 0 | + | 53 54 1 1 0 0 0 |
| − | 54 55 1 1 0 0 0 | + | 54 55 1 1 0 0 0 |
| − | 56 55 1 1 0 0 0 | + | 56 55 1 1 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 58 53 1 0 0 0 0 | + | 58 53 1 0 0 0 0 |
| − | 55 59 1 0 0 0 0 | + | 55 59 1 0 0 0 0 |
| − | 56 60 1 0 0 0 0 | + | 56 60 1 0 0 0 0 |
| − | 53 61 1 0 0 0 0 | + | 53 61 1 0 0 0 0 |
| − | 54 62 1 0 0 0 0 | + | 54 62 1 0 0 0 0 |
| − | 58 63 1 0 0 0 0 | + | 58 63 1 0 0 0 0 |
| − | 63 64 1 0 0 0 0 | + | 63 64 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FACGS0018 | + | ID FL3FACGS0018 |
| − | FORMULA C39H50O25 | + | FORMULA C39H50O25 |
| − | EXACTMASS 918.26411715 | + | EXACTMASS 918.26411715 |
| − | AVERAGEMASS 918.7993 | + | AVERAGEMASS 918.7993 |
| − | SMILES C(C(=O)4)=C(c(c7)cc(c(c7)OC(O5)C(OC(C6O)OC(CO)C(C6O)O)C(O)C(C5CO)O)O)Oc(c43)cc(cc3O)OC(O1)C(OC(O2)C(O)C(O)C(O)C2C)C(C(O)C1CO)O | + | SMILES C(C(=O)4)=C(c(c7)cc(c(c7)OC(O5)C(OC(C6O)OC(CO)C(C6O)O)C(O)C(C5CO)O)O)Oc(c43)cc(cc3O)OC(O1)C(OC(O2)C(O)C(O)C(O)C2C)C(C(O)C1CO)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
64 70 0 0 0 0 0 0 0 0999 V2000
0.7264 -1.2106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7264 -2.0356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4409 -2.4481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1553 -2.0356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1553 -1.2106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4409 -0.7981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0119 -0.7981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7025 -1.2106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7025 -2.0356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0119 -2.4481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8698 -0.7981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4409 -3.2731 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0119 -3.2731 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5843 -1.2106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2988 -0.7981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2988 0.0269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5843 0.4394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8698 0.0269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0132 -1.2106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7918 2.8308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7918 2.0058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5875 1.7878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1699 1.2101 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1700 2.0351 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3743 2.2532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6507 0.9821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1613 2.9398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2718 2.4847 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3757 2.8706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6067 3.2683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9178 -2.1564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3303 -2.8709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5371 -2.6441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7390 -2.8709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3264 -2.1564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1197 -2.3830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6964 -3.5529 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0101 -3.2568 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8097 -2.7663 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7891 -2.2506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2177 -0.2971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8052 -1.0116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0071 -0.8025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2139 -1.0116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6263 -0.2971 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4245 -0.5062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4694 -1.4153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4170 -0.7981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0132 -0.4709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5801 -0.9439 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9181 0.0006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6944 -0.0127 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0467 3.1153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6342 2.4008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2143 1.8143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4299 1.0228 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8424 1.7372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2623 2.3238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8857 0.9953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0132 0.4394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5884 3.5529 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3396 3.0520 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4919 2.8293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2385 3.4805 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 1 0 0 0 0
1 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 2 1 0 0 0 0
5 11 1 0 0 0 0
3 12 2 0 0 0 0
10 13 1 0 0 0 0
8 48 1 0 0 0 0
11 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 11 1 0 0 0 0
16 60 1 0 0 0 0
15 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
22 26 1 0 0 0 0
23 59 1 0 0 0 0
20 27 1 0 0 0 0
21 28 1 0 0 0 0
25 29 1 0 0 0 0
29 30 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
35 47 1 0 0 0 0
34 37 1 0 0 0 0
33 38 1 0 0 0 0
32 39 1 0 0 0 0
31 40 1 0 0 0 0
41 42 1 1 0 0 0
42 43 1 1 0 0 0
44 43 1 1 0 0 0
44 45 1 0 0 0 0
45 46 1 0 0 0 0
46 41 1 0 0 0 0
43 47 1 0 0 0 0
44 48 1 0 0 0 0
41 49 1 0 0 0 0
42 50 1 0 0 0 0
46 51 1 0 0 0 0
51 52 1 0 0 0 0
53 54 1 1 0 0 0
54 55 1 1 0 0 0
56 55 1 1 0 0 0
56 57 1 0 0 0 0
57 58 1 0 0 0 0
58 53 1 0 0 0 0
55 59 1 0 0 0 0
56 60 1 0 0 0 0
53 61 1 0 0 0 0
54 62 1 0 0 0 0
58 63 1 0 0 0 0
63 64 1 0 0 0 0
S SKP 5
ID FL3FACGS0018
FORMULA C39H50O25
EXACTMASS 918.26411715
AVERAGEMASS 918.7993
SMILES C(C(=O)4)=C(c(c7)cc(c(c7)OC(O5)C(OC(C6O)OC(CO)C(C6O)O)C(O)C(C5CO)O)O)Oc(c43)cc(cc3O)OC(O1)C(OC(O2)C(O)C(O)C(O)C2C)C(C(O)C1CO)O
M END