Mol:FL3FACGS0034
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.8703 -0.9349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8703 -0.9349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8703 -1.4557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8703 -1.4557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4192 -1.7162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4192 -1.7162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9681 -1.4557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9681 -1.4557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9681 -0.9349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9681 -0.9349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4192 -0.6745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4192 -0.6745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5171 -1.7162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5171 -1.7162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0660 -1.4557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0660 -1.4557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0660 -0.9349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0660 -0.9349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5171 -0.6745 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5171 -0.6745 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3455 -2.0840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3455 -2.0840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6151 -0.6746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6151 -0.6746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1554 -0.9400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1554 -0.9400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3043 -0.6746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3043 -0.6746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3043 -0.1437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3043 -0.1437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1554 0.1217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1554 0.1217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6151 -0.1437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6151 -0.1437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3898 -0.6350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3898 -0.6350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4192 -2.2359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4192 -2.2359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8987 0.1994 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8987 0.1994 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1554 0.6525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1554 0.6525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9336 0.4358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9336 0.4358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3054 0.0731 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.3054 0.0731 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.5047 0.7706 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.5047 0.7706 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.3054 1.4723 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.3054 1.4723 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.9336 1.8351 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.9336 1.8351 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.7343 1.1376 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.7343 1.1376 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -4.2170 2.3260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2170 2.3260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9134 1.6987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9134 1.6987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0428 0.5039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0428 0.5039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9808 0.7665 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.9808 0.7665 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.5932 0.0951 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.5932 0.0951 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.3386 0.3081 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3386 0.3081 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0886 0.0951 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.0886 0.0951 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.4763 0.7665 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.4763 0.7665 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.7308 0.5535 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.7308 0.5535 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.2608 0.5736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2608 0.5736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0378 1.0852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0378 1.0852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0357 0.4436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0357 0.4436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5034 1.4125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5034 1.4125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2657 0.7653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2657 0.7653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6008 0.1218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6008 0.1218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3152 -0.2907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3152 -0.2907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 18 1 1 0 0 0 0 | + | 18 1 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 22 1 1 0 0 0 | + | 27 22 1 1 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 18 23 1 0 0 0 0 | + | 18 23 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 32 20 1 0 0 0 0 | + | 32 20 1 0 0 0 0 |
| − | 27 40 1 0 0 0 0 | + | 27 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 46 3.6008 0.1218 | + | M SVB 2 46 3.6008 0.1218 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 44 -4.3152 1.7118 | + | M SVB 1 44 -4.3152 1.7118 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FACGS0034 | + | ID FL3FACGS0034 |
| − | KNApSAcK_ID C00004294 | + | KNApSAcK_ID C00004294 |
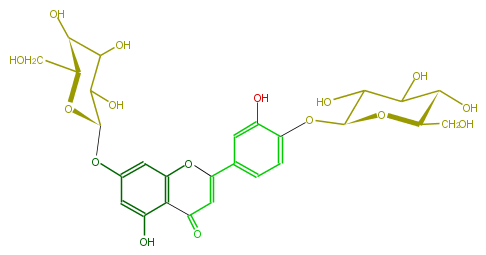
| − | NAME Luteolin 7,4'-diglucoside | + | NAME Luteolin 7,4'-diglucoside |
| − | CAS_RN 70404-47-6 | + | CAS_RN 70404-47-6 |
| − | FORMULA C27H30O16 | + | FORMULA C27H30O16 |
| − | EXACTMASS 610.153384912 | + | EXACTMASS 610.153384912 |
| − | AVERAGEMASS 610.5175 | + | AVERAGEMASS 610.5175 |
| − | SMILES c(O)(c4)c(c(cc(O[C@H](C(O)5)O[C@@H](CO)[C@@H](O)C5O)4)1)C(=O)C=C(c(c2)cc(O)c(O[C@H](O3)C(C([C@@H](O)[C@@H](CO)3)O)O)c2)O1 | + | SMILES c(O)(c4)c(c(cc(O[C@H](C(O)5)O[C@@H](CO)[C@@H](O)C5O)4)1)C(=O)C=C(c(c2)cc(O)c(O[C@H](O3)C(C([C@@H](O)[C@@H](CO)3)O)O)c2)O1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-2.8703 -0.9349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8703 -1.4557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4192 -1.7162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9681 -1.4557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9681 -0.9349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4192 -0.6745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5171 -1.7162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0660 -1.4557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0660 -0.9349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5171 -0.6745 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3455 -2.0840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6151 -0.6746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1554 -0.9400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3043 -0.6746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3043 -0.1437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1554 0.1217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6151 -0.1437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3898 -0.6350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4192 -2.2359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8987 0.1994 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1554 0.6525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9336 0.4358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3054 0.0731 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.5047 0.7706 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.3054 1.4723 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.9336 1.8351 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.7343 1.1376 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-4.2170 2.3260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9134 1.6987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0428 0.5039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9808 0.7665 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.5932 0.0951 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.3386 0.3081 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0886 0.0951 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.4763 0.7665 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.7308 0.5535 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.2608 0.5736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0378 1.0852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0357 0.4436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5034 1.4125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2657 0.7653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6008 0.1218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3152 -0.2907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
18 1 1 0 0 0 0
3 19 1 0 0 0 0
15 20 1 0 0 0 0
16 21 1 0 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 1 0 0 0
27 22 1 1 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
18 23 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
36 38 1 0 0 0 0
35 39 1 0 0 0 0
32 20 1 0 0 0 0
27 40 1 0 0 0 0
40 41 1 0 0 0 0
34 42 1 0 0 0 0
42 43 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 46
M SMT 2 CH2OH
M SVB 2 46 3.6008 0.1218
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 44
M SMT 1 CH2OH
M SVB 1 44 -4.3152 1.7118
S SKP 8
ID FL3FACGS0034
KNApSAcK_ID C00004294
NAME Luteolin 7,4'-diglucoside
CAS_RN 70404-47-6
FORMULA C27H30O16
EXACTMASS 610.153384912
AVERAGEMASS 610.5175
SMILES c(O)(c4)c(c(cc(O[C@H](C(O)5)O[C@@H](CO)[C@@H](O)C5O)4)1)C(=O)C=C(c(c2)cc(O)c(O[C@H](O3)C(C([C@@H](O)[C@@H](CO)3)O)O)c2)O1
M END