Mol:FL3FADCS0020
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.4365 0.4902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4365 0.4902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4365 -0.3194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4365 -0.3194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7354 -0.7241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7354 -0.7241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0343 -0.3194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0343 -0.3194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0343 0.4902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0343 0.4902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7354 0.8950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7354 0.8950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6668 -0.7241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6668 -0.7241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3679 -0.3194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3679 -0.3194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3679 0.4902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3679 0.4902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6668 0.8950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6668 0.8950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6668 -1.3554 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6668 -1.3554 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1374 0.8949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1374 0.8949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7354 -1.4017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7354 -1.4017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1208 0.9208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1208 0.9208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8354 0.5082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8354 0.5082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5499 0.9208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5499 0.9208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5499 1.7460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5499 1.7460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8354 2.1585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8354 2.1585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1208 1.7460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1208 1.7460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6497 -2.7482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6497 -2.7482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1320 -2.0551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1320 -2.0551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3517 -1.9055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3517 -1.9055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4181 -0.7852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4181 -0.7852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9408 -1.5853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9408 -1.5853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7611 -1.8153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7611 -1.8153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9793 -3.3597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9793 -3.3597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2375 -2.8962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2375 -2.8962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7205 -1.1982 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7205 -1.1982 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3329 -3.5516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3329 -3.5516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9085 -2.9963 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9085 -2.9963 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1152 -2.6099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1152 -2.6099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2127 -1.6219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2127 -1.6219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6431 -2.4727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6431 -2.4727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4443 -2.6189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4443 -2.6189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8659 -3.6552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8659 -3.6552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9891 -3.9988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9891 -3.9988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6516 -3.2853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6516 -3.2853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1843 -2.4752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1843 -2.4752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9996 -3.1972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9996 -3.1972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1843 2.1123 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1843 2.1123 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8354 2.8679 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8354 2.8679 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3917 3.9988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3917 3.9988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 3 13 1 0 0 0 0 | + | 3 13 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 19 2 0 0 0 0 | + | 18 19 2 0 0 0 0 |
| − | 19 14 1 0 0 0 0 | + | 19 14 1 0 0 0 0 |
| − | 14 9 1 0 0 0 0 | + | 14 9 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 2 0 0 0 0 | + | 20 26 2 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 28 1 0 0 0 0 | + | 32 28 1 0 0 0 0 |
| − | 25 38 1 0 0 0 0 | + | 25 38 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 23 2 1 0 0 0 0 | + | 23 2 1 0 0 0 0 |
| − | 17 40 1 0 0 0 0 | + | 17 40 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 18 41 1 0 0 0 0 | + | 18 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 46 0.0000 -0.7094 | + | M SBV 1 46 0.0000 -0.7094 |
| − | S SKP 5 | + | S SKP 5 |
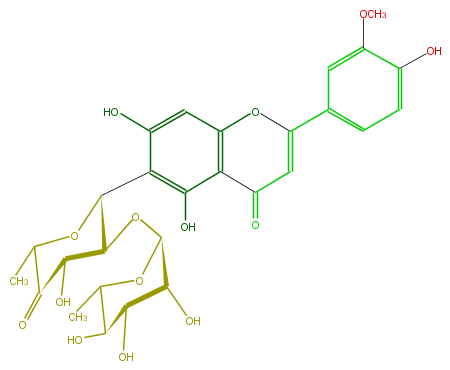
| − | ID FL3FADCS0020 | + | ID FL3FADCS0020 |
| − | FORMULA C28H30O14 | + | FORMULA C28H30O14 |
| − | EXACTMASS 590.163555668 | + | EXACTMASS 590.163555668 |
| − | AVERAGEMASS 590.5294 | + | AVERAGEMASS 590.5294 |
| − | SMILES C(C1=O)=C(c(c5)cc(c(O)c5)OC)Oc(c2)c1c(O)c(C(C3OC(C4O)OC(C)C(C(O)4)O)OC(C)C(C3O)=O)c2O | + | SMILES C(C1=O)=C(c(c5)cc(c(O)c5)OC)Oc(c2)c1c(O)c(C(C3OC(C4O)OC(C)C(C(O)4)O)OC(C)C(C3O)=O)c2O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-1.4365 0.4902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4365 -0.3194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7354 -0.7241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0343 -0.3194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0343 0.4902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7354 0.8950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6668 -0.7241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3679 -0.3194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3679 0.4902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6668 0.8950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6668 -1.3554 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1374 0.8949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7354 -1.4017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1208 0.9208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8354 0.5082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5499 0.9208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5499 1.7460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8354 2.1585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1208 1.7460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6497 -2.7482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1320 -2.0551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3517 -1.9055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4181 -0.7852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9408 -1.5853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7611 -1.8153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9793 -3.3597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2375 -2.8962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7205 -1.1982 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3329 -3.5516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9085 -2.9963 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1152 -2.6099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2127 -1.6219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6431 -2.4727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4443 -2.6189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8659 -3.6552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9891 -3.9988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6516 -3.2853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1843 -2.4752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9996 -3.1972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1843 2.1123 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8354 2.8679 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3917 3.9988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
3 13 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 18 1 0 0 0 0
18 19 2 0 0 0 0
19 14 1 0 0 0 0
14 9 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 2 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
29 30 1 1 0 0 0
30 31 1 1 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 29 1 0 0 0 0
29 35 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 28 1 0 0 0 0
25 38 1 0 0 0 0
34 39 1 0 0 0 0
23 2 1 0 0 0 0
17 40 1 0 0 0 0
41 42 1 0 0 0 0
18 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 46
M SMT 1 OCH3
M SBV 1 46 0.0000 -0.7094
S SKP 5
ID FL3FADCS0020
FORMULA C28H30O14
EXACTMASS 590.163555668
AVERAGEMASS 590.5294
SMILES C(C1=O)=C(c(c5)cc(c(O)c5)OC)Oc(c2)c1c(O)c(C(C3OC(C4O)OC(C)C(C(O)4)O)OC(C)C(C3O)=O)c2O
M END