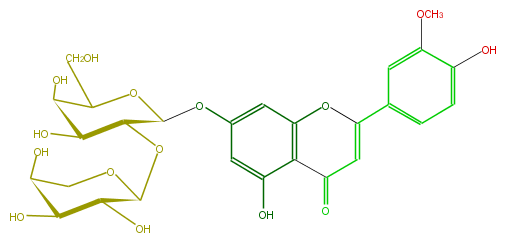
Mol:FL3FADGS0007
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.4387 -0.6920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4387 -0.6920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4387 -1.4182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4387 -1.4182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1903 -1.7813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1903 -1.7813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8193 -1.4182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8193 -1.4182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8193 -0.6920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8193 -0.6920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1903 -0.3289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1903 -0.3289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4482 -1.7813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4482 -1.7813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0772 -1.4182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0772 -1.4182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0772 -0.6920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0772 -0.6920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4482 -0.3289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4482 -0.3289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4482 -2.4379 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4482 -2.4379 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7059 -0.3289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7059 -0.3289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3469 -0.6991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3469 -0.6991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9879 -0.3289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9879 -0.3289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9879 0.4113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9879 0.4113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3469 0.7813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3469 0.7813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7059 0.4113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7059 0.4113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1903 -2.5061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1903 -2.5061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6532 0.7953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6532 0.7953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0663 -0.3296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0663 -0.3296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0088 -0.2290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0088 -0.2290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4335 -0.9883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4335 -0.9883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6052 -0.6662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6052 -0.6662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8061 -0.6576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8061 -0.6576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3868 -0.0766 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3868 -0.0766 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2330 -0.3805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2330 -0.3805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9424 0.1823 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9424 0.1823 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1639 -0.8890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1639 -0.8890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8818 -1.1962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8818 -1.1962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3206 -1.2430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3206 -1.2430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4705 -1.8023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4705 -1.8023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8952 -2.5616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8952 -2.5616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0670 -2.2395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0670 -2.2395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2678 -2.2309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2678 -2.2309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8485 -1.6499 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8485 -1.6499 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6947 -1.9538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6947 -1.9538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6532 -2.5424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6532 -2.5424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2817 -2.7901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2817 -2.7901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3469 1.5051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3469 1.5051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8885 2.7901 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8885 2.7901 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7747 0.6297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7747 0.6297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7883 1.7451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7883 1.7451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 24 20 1 0 0 0 0 | + | 24 20 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 16 39 1 0 0 0 0 | + | 16 39 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 26 41 1 0 0 0 0 | + | 26 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 44 0.0000 -0.7238 | + | M SBV 1 44 0.0000 -0.7238 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 41 42 | + | M SAL 2 2 41 42 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 ^ CH2OH | + | M SMT 2 ^ CH2OH |
| − | M SBV 2 46 0.5418 -1.0102 | + | M SBV 2 46 0.5418 -1.0102 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FADGS0007 | + | ID FL3FADGS0007 |
| − | FORMULA C27H30O15 | + | FORMULA C27H30O15 |
| − | EXACTMASS 594.15847029 | + | EXACTMASS 594.15847029 |
| − | AVERAGEMASS 594.5181 | + | AVERAGEMASS 594.5181 |
| − | SMILES O(C(c(c5)cc(OC)c(c5)O)=4)c(c3C(=O)C4)cc(cc3O)OC(O2)C(C(C(O)C(CO)2)O)OC(O1)C(C(C(C1)O)O)O | + | SMILES O(C(c(c5)cc(OC)c(c5)O)=4)c(c3C(=O)C4)cc(cc3O)OC(O2)C(C(C(O)C(CO)2)O)OC(O1)C(C(C(C1)O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-0.4387 -0.6920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4387 -1.4182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1903 -1.7813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8193 -1.4182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8193 -0.6920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1903 -0.3289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4482 -1.7813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0772 -1.4182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0772 -0.6920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4482 -0.3289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4482 -2.4379 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7059 -0.3289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3469 -0.6991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9879 -0.3289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9879 0.4113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3469 0.7813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7059 0.4113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1903 -2.5061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6532 0.7953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0663 -0.3296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0088 -0.2290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4335 -0.9883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6052 -0.6662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8061 -0.6576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3868 -0.0766 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2330 -0.3805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9424 0.1823 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1639 -0.8890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8818 -1.1962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3206 -1.2430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4705 -1.8023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8952 -2.5616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0670 -2.2395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2678 -2.2309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8485 -1.6499 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6947 -1.9538 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6532 -2.5424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2817 -2.7901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3469 1.5051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8885 2.7901 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7747 0.6297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7883 1.7451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
1 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
32 37 1 0 0 0 0
33 38 1 0 0 0 0
30 31 1 0 0 0 0
34 29 1 0 0 0 0
24 20 1 0 0 0 0
39 40 1 0 0 0 0
16 39 1 0 0 0 0
41 42 1 0 0 0 0
26 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 44
M SMT 1 OCH3
M SBV 1 44 0.0000 -0.7238
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 41 42
M SBL 2 1 46
M SMT 2 ^ CH2OH
M SBV 2 46 0.5418 -1.0102
S SKP 5
ID FL3FADGS0007
FORMULA C27H30O15
EXACTMASS 594.15847029
AVERAGEMASS 594.5181
SMILES O(C(c(c5)cc(OC)c(c5)O)=4)c(c3C(=O)C4)cc(cc3O)OC(O2)C(C(C(O)C(CO)2)O)OC(O1)C(C(C(C1)O)O)O
M END