Mol:FL3FAGGS0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.5004 -1.0970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5004 -1.0970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5004 -1.6591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5004 -1.6591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0493 -1.9195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0493 -1.9195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5983 -1.6591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5983 -1.6591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5983 -1.1382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5983 -1.1382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0493 -0.8778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0493 -0.8778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1472 -1.9195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1472 -1.9195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6961 -1.6591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6961 -1.6591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6961 -1.1382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6961 -1.1382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1472 -0.8778 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1472 -0.8778 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1472 -2.3256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1472 -2.3256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2452 -0.8779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2452 -0.8779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7855 -1.1433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7855 -1.1433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3258 -0.8779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3258 -0.8779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3258 -0.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3258 -0.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7855 -0.0816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7855 -0.0816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2452 -0.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2452 -0.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0493 -2.4392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0493 -2.4392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9532 -0.7981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9532 -0.7981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1338 -0.0817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1338 -0.0817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1338 -1.1433 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1338 -1.1433 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7770 0.7230 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7770 0.7230 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0990 1.9854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0990 1.9854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9928 1.1381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9928 1.1381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2054 1.0169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2054 1.0169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5810 0.6654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5810 0.6654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7715 1.3767 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7715 1.3767 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5646 1.5201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5646 1.5201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7647 1.9854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7647 1.9854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7084 1.5061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7084 1.5061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1940 0.1857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1940 0.1857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6188 -0.7568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6188 -0.7568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2312 -1.4283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2312 -1.4283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9766 -1.2152 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9766 -1.2152 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7266 -1.4283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7266 -1.4283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1143 -0.7568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1143 -0.7568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3688 -0.9698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3688 -0.9698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8988 -0.9497 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8988 -0.9497 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6758 -0.4382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6758 -0.4382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6737 -1.0798 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6737 -1.0798 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2388 -1.4015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2388 -1.4015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9532 -1.8140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9532 -1.8140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5759 2.4392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5759 2.4392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8615 2.0267 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8615 2.0267 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 14 21 1 0 0 0 0 | + | 14 21 1 0 0 0 0 |
| − | 22 16 1 0 0 0 0 | + | 22 16 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 26 22 1 0 0 0 0 | + | 26 22 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 37 39 1 0 0 0 0 | + | 37 39 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 33 21 1 0 0 0 0 | + | 33 21 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 28 43 1 0 0 0 0 | + | 28 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 45 -6.3830 4.1993 | + | M SBV 1 45 -6.3830 4.1993 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 43 44 | + | M SAL 2 2 43 44 |
| − | M SBL 2 1 47 | + | M SBL 2 1 47 |
| − | M SMT 2 ^CH2OH | + | M SMT 2 ^CH2OH |
| − | M SBV 2 47 -6.9066 5.0917 | + | M SBV 2 47 -6.9066 5.0917 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FAGGS0005 | + | ID FL3FAGGS0005 |
| − | KNApSAcK_ID C00004456 | + | KNApSAcK_ID C00004456 |
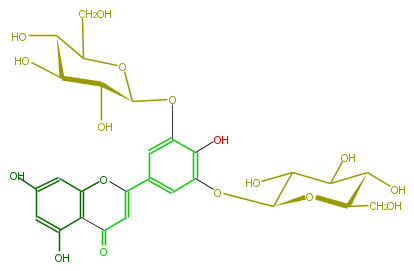
| − | NAME Tricetin 3',5'-diglucoside | + | NAME Tricetin 3',5'-diglucoside |
| − | CAS_RN 93373-19-4 | + | CAS_RN 93373-19-4 |
| − | FORMULA C27H30O17 | + | FORMULA C27H30O17 |
| − | EXACTMASS 626.148299534 | + | EXACTMASS 626.148299534 |
| − | AVERAGEMASS 626.5169000000001 | + | AVERAGEMASS 626.5169000000001 |
| − | SMILES Oc(c1)cc(O2)c(C(=O)C=C2c(c4)cc(c(O)c4OC(O5)C(C(C(O)C5CO)O)O)OC(O3)C(C(C(O)C3CO)O)O)c1O | + | SMILES Oc(c1)cc(O2)c(C(=O)C=C2c(c4)cc(c(O)c4OC(O5)C(C(C(O)C5CO)O)O)OC(O3)C(C(C(O)C3CO)O)O)c1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-3.5004 -1.0970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5004 -1.6591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0493 -1.9195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5983 -1.6591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5983 -1.1382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0493 -0.8778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1472 -1.9195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6961 -1.6591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6961 -1.1382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1472 -0.8778 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1472 -2.3256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2452 -0.8779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7855 -1.1433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3258 -0.8779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3258 -0.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7855 -0.0816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2452 -0.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0493 -2.4392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9532 -0.7981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1338 -0.0817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1338 -1.1433 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7770 0.7230 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0990 1.9854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9928 1.1381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2054 1.0169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5810 0.6654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7715 1.3767 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5646 1.5201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7647 1.9854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7084 1.5061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1940 0.1857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6188 -0.7568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2312 -1.4283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9766 -1.2152 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7266 -1.4283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1143 -0.7568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3688 -0.9698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8988 -0.9497 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6758 -0.4382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6737 -1.0798 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2388 -1.4015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9532 -1.8140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5759 2.4392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8615 2.0267 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
1 19 1 0 0 0 0
15 20 1 0 0 0 0
14 21 1 0 0 0 0
22 16 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 31 1 0 0 0 0
26 22 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
32 38 1 0 0 0 0
37 39 1 0 0 0 0
36 40 1 0 0 0 0
33 21 1 0 0 0 0
35 41 1 0 0 0 0
41 42 1 0 0 0 0
28 43 1 0 0 0 0
43 44 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 45
M SMT 1 CH2OH
M SBV 1 45 -6.3830 4.1993
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 43 44
M SBL 2 1 47
M SMT 2 ^CH2OH
M SBV 2 47 -6.9066 5.0917
S SKP 8
ID FL3FAGGS0005
KNApSAcK_ID C00004456
NAME Tricetin 3',5'-diglucoside
CAS_RN 93373-19-4
FORMULA C27H30O17
EXACTMASS 626.148299534
AVERAGEMASS 626.5169000000001
SMILES Oc(c1)cc(O2)c(C(=O)C=C2c(c4)cc(c(O)c4OC(O5)C(C(C(O)C5CO)O)O)OC(O3)C(C(C(O)C3CO)O)O)c1O
M END