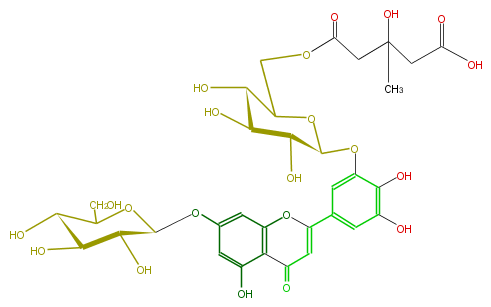
Mol:FL3FAGGS0006
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 54 58 0 0 0 0 0 0 0 0999 V2000 | + | 54 58 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.5426 -1.4528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5426 -1.4528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5426 -2.0149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5426 -2.0149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0915 -2.2753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0915 -2.2753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3595 -2.0149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3595 -2.0149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3595 -1.4940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3595 -1.4940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0915 -1.2336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0915 -1.2336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8106 -2.2753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8106 -2.2753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2617 -2.0149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2617 -2.0149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2617 -1.4940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2617 -1.4940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8106 -1.2336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8106 -1.2336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8106 -2.6814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8106 -2.6814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7126 -1.2337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7126 -1.2337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1723 -1.4991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1723 -1.4991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6320 -1.2337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6320 -1.2337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6320 -0.7028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6320 -0.7028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1723 -0.4374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1723 -0.4374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7126 -0.7028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7126 -0.7028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0915 -2.7950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0915 -2.7950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0038 -1.1865 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0038 -1.1865 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0916 -0.4375 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0916 -0.4375 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7971 -1.2158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7971 -1.2158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2815 -1.8964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2815 -1.8964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5390 -1.6076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5390 -1.6076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8226 -1.5999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8226 -1.5999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3432 -1.0792 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3432 -1.0792 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9928 -1.4220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9928 -1.4220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5109 -1.6279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5109 -1.6279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0853 -1.9355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0853 -1.9355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1136 -2.3218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1136 -2.3218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1723 0.0933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1723 0.0933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9072 2.1300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9072 2.1300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3029 1.7811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3029 1.7811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8368 2.2161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8368 2.2161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2387 1.8654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2387 1.8654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7643 2.2986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7643 2.2986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2037 1.9749 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2037 1.9749 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8368 2.7950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8368 2.7950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8368 1.2859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8368 1.2859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9072 2.6907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9072 2.6907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7643 2.7317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7643 2.7317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0098 1.3104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0098 1.3104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0964 0.4631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0964 0.4631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8838 0.3419 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8838 0.3419 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5082 -0.0096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5082 -0.0096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3177 0.7017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3177 0.7017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5837 0.7296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5837 0.7296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8340 1.3104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8340 1.3104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6192 0.8311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6192 0.8311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8952 -0.4893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8952 -0.4893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2873 1.8359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2873 1.8359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5109 1.7814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5109 1.7814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0916 -1.4991 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0916 -1.4991 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0627 -1.0236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0627 -1.0236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8596 -0.8100 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8596 -0.8100 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 1 1 0 0 0 0 | + | 19 1 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 16 30 1 0 0 0 0 | + | 16 30 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 33 37 1 0 0 0 0 | + | 33 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 31 39 2 0 0 0 0 | + | 31 39 2 0 0 0 0 |
| − | 35 40 2 0 0 0 0 | + | 35 40 2 0 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 44 43 1 1 0 0 0 | + | 44 43 1 1 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 41 1 0 0 0 0 | + | 46 41 1 0 0 0 0 |
| − | 41 47 1 0 0 0 0 | + | 41 47 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 46 50 1 0 0 0 0 | + | 46 50 1 0 0 0 0 |
| − | 44 30 1 0 0 0 0 | + | 44 30 1 0 0 0 0 |
| − | 50 36 1 0 0 0 0 | + | 50 36 1 0 0 0 0 |
| − | 31 51 1 0 0 0 0 | + | 31 51 1 0 0 0 0 |
| − | 14 52 1 0 0 0 0 | + | 14 52 1 0 0 0 0 |
| − | 26 53 1 0 0 0 0 | + | 26 53 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 53 54 | + | M SAL 1 2 53 54 |
| − | M SBL 1 1 57 | + | M SBL 1 1 57 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 57 -8.6067 5.6589 | + | M SBV 1 57 -8.6067 5.6589 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FAGGS0006 | + | ID FL3FAGGS0006 |
| − | KNApSAcK_ID C00004527 | + | KNApSAcK_ID C00004527 |
| − | NAME Tricetin 7-glucoside-3'-[6''-(3-hydroxy-3-methylglutaryl)glucoside | + | NAME Tricetin 7-glucoside-3'-[6''-(3-hydroxy-3-methylglutaryl)glucoside |
| − | CAS_RN 151750-87-7 | + | CAS_RN 151750-87-7 |
| − | FORMULA C33H38O21 | + | FORMULA C33H38O21 |
| − | EXACTMASS 770.190558278 | + | EXACTMASS 770.190558278 |
| − | AVERAGEMASS 770.64222 | + | AVERAGEMASS 770.64222 |
| − | SMILES OC(C5O)C(OC(C(O)5)COC(CC(C)(O)CC(O)=O)=O)Oc(c(O)1)cc(C(O4)=CC(c(c34)c(O)cc(c3)OC(C2O)OC(C(O)C2O)CO)=O)cc1O | + | SMILES OC(C5O)C(OC(C(O)5)COC(CC(C)(O)CC(O)=O)=O)Oc(c(O)1)cc(C(O4)=CC(c(c34)c(O)cc(c3)OC(C2O)OC(C(O)C2O)CO)=O)cc1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
54 58 0 0 0 0 0 0 0 0999 V2000
-0.5426 -1.4528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5426 -2.0149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0915 -2.2753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3595 -2.0149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3595 -1.4940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0915 -1.2336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8106 -2.2753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2617 -2.0149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2617 -1.4940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8106 -1.2336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8106 -2.6814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7126 -1.2337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1723 -1.4991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6320 -1.2337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6320 -0.7028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1723 -0.4374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7126 -0.7028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0915 -2.7950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0038 -1.1865 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0916 -0.4375 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7971 -1.2158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2815 -1.8964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5390 -1.6076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8226 -1.5999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3432 -1.0792 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9928 -1.4220 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5109 -1.6279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0853 -1.9355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1136 -2.3218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1723 0.0933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9072 2.1300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3029 1.7811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8368 2.2161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2387 1.8654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7643 2.2986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2037 1.9749 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8368 2.7950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8368 1.2859 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9072 2.6907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7643 2.7317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0098 1.3104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0964 0.4631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8838 0.3419 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5082 -0.0096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3177 0.7017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5837 0.7296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8340 1.3104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6192 0.8311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8952 -0.4893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2873 1.8359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5109 1.7814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0916 -1.4991 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0627 -1.0236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8596 -0.8100 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 1 1 0 0 0 0
15 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 19 1 0 0 0 0
16 30 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
33 37 1 0 0 0 0
33 38 1 0 0 0 0
31 39 2 0 0 0 0
35 40 2 0 0 0 0
41 42 1 1 0 0 0
42 43 1 1 0 0 0
44 43 1 1 0 0 0
44 45 1 0 0 0 0
45 46 1 0 0 0 0
46 41 1 0 0 0 0
41 47 1 0 0 0 0
42 48 1 0 0 0 0
43 49 1 0 0 0 0
46 50 1 0 0 0 0
44 30 1 0 0 0 0
50 36 1 0 0 0 0
31 51 1 0 0 0 0
14 52 1 0 0 0 0
26 53 1 0 0 0 0
53 54 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 53 54
M SBL 1 1 57
M SMT 1 ^CH2OH
M SBV 1 57 -8.6067 5.6589
S SKP 8
ID FL3FAGGS0006
KNApSAcK_ID C00004527
NAME Tricetin 7-glucoside-3'-[6''-(3-hydroxy-3-methylglutaryl)glucoside
CAS_RN 151750-87-7
FORMULA C33H38O21
EXACTMASS 770.190558278
AVERAGEMASS 770.64222
SMILES OC(C5O)C(OC(C(O)5)COC(CC(C)(O)CC(O)=O)=O)Oc(c(O)1)cc(C(O4)=CC(c(c34)c(O)cc(c3)OC(C2O)OC(C(O)C2O)CO)=O)cc1O
M END