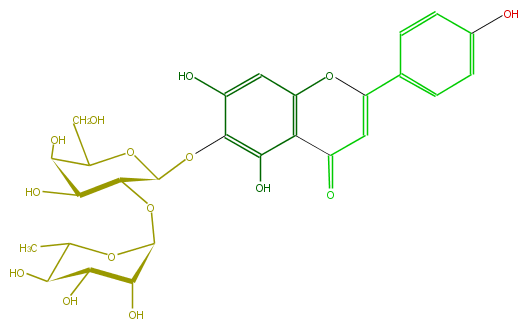
Mol:FL3FEAGS0011
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.7815 1.3675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7815 1.3675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7815 0.5580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7815 0.5580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0805 0.1533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0805 0.1533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6203 0.5580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6203 0.5580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6203 1.3675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6203 1.3675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0805 1.7721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0805 1.7721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3213 0.1533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3213 0.1533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0223 0.5580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0223 0.5580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0223 1.3675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0223 1.3675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3213 1.7721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3213 1.7721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3420 -0.6054 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3420 -0.6054 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7230 1.7720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7230 1.7720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4374 1.3595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4374 1.3595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1517 1.7720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1517 1.7720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1517 2.5970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1517 2.5970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4374 3.0094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4374 3.0094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7230 2.5970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7230 2.5970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4822 1.7720 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4822 1.7720 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8659 3.0092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8659 3.0092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0805 -0.4780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0805 -0.4780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4809 0.1543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4809 0.1543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2566 0.0972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2566 0.0972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6957 -0.6431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6957 -0.6431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8880 -0.3291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8880 -0.3291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1087 -0.3207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1087 -0.3207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6750 0.2458 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6750 0.2458 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5001 -0.0505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5001 -0.0505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1937 0.4920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1937 0.4920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5481 -0.5428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5481 -0.5428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2589 -0.8805 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2589 -0.8805 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8922 -1.6022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8922 -1.6022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3330 -2.3658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3330 -2.3658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4851 -2.1236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4851 -2.1236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6322 -2.3658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6322 -2.3658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1911 -1.6022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1911 -1.6022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0391 -1.8445 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0391 -1.8445 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9485 -2.7307 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9485 -2.7307 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8659 -2.1674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8659 -2.1674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5884 -1.6625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5884 -1.6625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6322 -3.0094 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6322 -3.0094 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8537 0.8975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8537 0.8975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4990 1.7731 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4990 1.7731 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 20 3 1 0 0 0 0 | + | 20 3 1 0 0 0 0 |
| − | 2 21 1 0 0 0 0 | + | 2 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 33 37 1 0 0 0 0 | + | 33 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 31 39 1 0 0 0 0 | + | 31 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 25 21 1 0 0 0 0 | + | 25 21 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 27 41 1 0 0 0 0 | + | 27 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 46 0.3536 -0.9480 | + | M SBV 1 46 0.3536 -0.9480 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FEAGS0011 | + | ID FL3FEAGS0011 |
| − | FORMULA C27H30O15 | + | FORMULA C27H30O15 |
| − | EXACTMASS 594.15847029 | + | EXACTMASS 594.15847029 |
| − | AVERAGEMASS 594.5181 | + | AVERAGEMASS 594.5181 |
| − | SMILES c(c5)(ccc(c5)C(O4)=CC(c(c34)c(O)c(c(c3)O)OC(C1OC(C2O)OC(C)C(C2O)O)OC(CO)C(C(O)1)O)=O)O | + | SMILES c(c5)(ccc(c5)C(O4)=CC(c(c34)c(O)c(c(c3)O)OC(C1OC(C2O)OC(C)C(C2O)O)OC(CO)C(C(O)1)O)=O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-0.7815 1.3675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7815 0.5580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0805 0.1533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6203 0.5580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6203 1.3675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0805 1.7721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3213 0.1533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0223 0.5580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0223 1.3675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3213 1.7721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3420 -0.6054 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7230 1.7720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4374 1.3595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1517 1.7720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1517 2.5970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4374 3.0094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7230 2.5970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4822 1.7720 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8659 3.0092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0805 -0.4780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4809 0.1543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2566 0.0972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6957 -0.6431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8880 -0.3291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1087 -0.3207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6750 0.2458 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5001 -0.0505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1937 0.4920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5481 -0.5428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2589 -0.8805 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8922 -1.6022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3330 -2.3658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4851 -2.1236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6322 -2.3658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1911 -1.6022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0391 -1.8445 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9485 -2.7307 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8659 -2.1674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5884 -1.6625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6322 -3.0094 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8537 0.8975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4990 1.7731 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
15 19 1 0 0 0 0
20 3 1 0 0 0 0
2 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
35 30 1 0 0 0 0
33 37 1 0 0 0 0
32 38 1 0 0 0 0
31 39 1 0 0 0 0
34 40 1 0 0 0 0
25 21 1 0 0 0 0
41 42 1 0 0 0 0
27 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 46
M SMT 1 ^ CH2OH
M SBV 1 46 0.3536 -0.9480
S SKP 5
ID FL3FEAGS0011
FORMULA C27H30O15
EXACTMASS 594.15847029
AVERAGEMASS 594.5181
SMILES c(c5)(ccc(c5)C(O4)=CC(c(c34)c(O)c(c(c3)O)OC(C1OC(C2O)OC(C)C(C2O)O)OC(CO)C(C(O)1)O)=O)O
M END