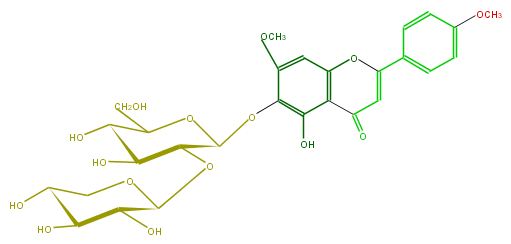
Mol:FL3FEAGS0036
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.2096 0.9398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2096 0.9398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2096 0.1980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2096 0.1980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8049 -0.1456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8049 -0.1456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4000 0.1980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4000 0.1980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4000 0.8854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4000 0.8854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8049 1.2290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8049 1.2290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9953 -0.1456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9953 -0.1456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5905 0.1980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5905 0.1980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5905 0.8854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5905 0.8854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9953 1.2290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9953 1.2290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2218 -0.6311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2218 -0.6311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1855 1.2289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1855 1.2289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7921 0.8787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7921 0.8787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3987 1.2289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3987 1.2289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3987 1.9294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3987 1.9294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7921 2.2797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7921 2.2797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1855 1.9294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1855 1.9294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8049 -0.8314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8049 -0.8314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4315 -0.1721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4315 -0.1721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8179 -0.3801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8179 -0.3801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1375 -1.2782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1375 -1.2782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1577 -0.8972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1577 -0.8972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2123 -0.8870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2123 -0.8870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8994 -0.1998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8994 -0.1998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9001 -0.5592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9001 -0.5592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5154 -0.6556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5154 -0.6556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9699 -1.2487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9699 -1.2487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4300 -1.3461 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4300 -1.3461 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9480 -2.2616 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9480 -2.2616 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2582 -1.8742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2582 -1.8742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5779 -2.7723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5779 -2.7723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5981 -2.3913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5981 -2.3913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6526 -2.3812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6526 -2.3812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3397 -1.6939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3397 -1.6939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3405 -2.0534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3405 -2.0534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2777 -2.8355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2777 -2.8355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8221 -2.8992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8221 -2.8992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0052 2.2795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0052 2.2795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9480 1.7352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9480 1.7352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7438 0.0752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7438 0.0752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2882 1.0180 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2882 1.0180 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2618 1.7563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2618 1.7563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9214 2.8992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9214 2.8992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 2 19 1 0 0 0 0 | + | 2 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 31 36 1 0 0 0 0 | + | 31 36 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 33 28 1 0 0 0 0 | + | 33 28 1 0 0 0 0 |
| − | 23 19 1 0 0 0 0 | + | 23 19 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 15 38 1 0 0 0 0 | + | 15 38 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 25 40 1 0 0 0 0 | + | 25 40 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 1 42 1 0 0 0 0 | + | 1 42 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 38 39 | + | M SAL 1 2 38 39 |
| − | M SBL 1 1 43 | + | M SBL 1 1 43 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 43 -0.6065 -0.3501 | + | M SBV 1 43 -0.6065 -0.3501 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 40 41 | + | M SAL 2 2 40 41 |
| − | M SBL 2 1 45 | + | M SBL 2 1 45 |
| − | M SMT 2 ^ CH2OH | + | M SMT 2 ^ CH2OH |
| − | M SBV 2 45 0.8436 -0.6343 | + | M SBV 2 45 0.8436 -0.6343 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 42 43 | + | M SAL 3 2 42 43 |
| − | M SBL 3 1 47 | + | M SBL 3 1 47 |
| − | M SMT 3 ^ OCH3 | + | M SMT 3 ^ OCH3 |
| − | M SBV 3 47 0.4714 -0.8166 | + | M SBV 3 47 0.4714 -0.8166 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FEAGS0036 | + | ID FL3FEAGS0036 |
| − | FORMULA C28H32O15 | + | FORMULA C28H32O15 |
| − | EXACTMASS 608.174120354 | + | EXACTMASS 608.174120354 |
| − | AVERAGEMASS 608.54468 | + | AVERAGEMASS 608.54468 |
| − | SMILES C(C(O2)C(O)C(C(C(Oc(c5O)c(OC)cc(c35)OC(c(c4)ccc(OC)c4)=CC3=O)2)OC(O1)C(C(O)C(C1)O)O)O)O | + | SMILES C(C(O2)C(O)C(C(C(Oc(c5O)c(OC)cc(c35)OC(c(c4)ccc(OC)c4)=CC3=O)2)OC(O1)C(C(O)C(C1)O)O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
0.2096 0.9398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2096 0.1980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8049 -0.1456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4000 0.1980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4000 0.8854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8049 1.2290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9953 -0.1456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5905 0.1980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5905 0.8854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9953 1.2290 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2218 -0.6311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1855 1.2289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7921 0.8787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3987 1.2289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3987 1.9294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7921 2.2797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1855 1.9294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8049 -0.8314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4315 -0.1721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8179 -0.3801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1375 -1.2782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1577 -0.8972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2123 -0.8870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8994 -0.1998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9001 -0.5592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5154 -0.6556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9699 -1.2487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4300 -1.3461 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.9480 -2.2616 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2582 -1.8742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5779 -2.7723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5981 -2.3913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6526 -2.3812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3397 -1.6939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3405 -2.0534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2777 -2.8355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8221 -2.8992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0052 2.2795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.9480 1.7352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7438 0.0752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2882 1.0180 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2618 1.7563 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9214 2.8992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
2 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
31 36 1 0 0 0 0
29 30 1 0 0 0 0
33 28 1 0 0 0 0
23 19 1 0 0 0 0
32 37 1 0 0 0 0
38 39 1 0 0 0 0
15 38 1 0 0 0 0
40 41 1 0 0 0 0
25 40 1 0 0 0 0
42 43 1 0 0 0 0
1 42 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 38 39
M SBL 1 1 43
M SMT 1 OCH3
M SBV 1 43 -0.6065 -0.3501
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 40 41
M SBL 2 1 45
M SMT 2 ^ CH2OH
M SBV 2 45 0.8436 -0.6343
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 42 43
M SBL 3 1 47
M SMT 3 ^ OCH3
M SBV 3 47 0.4714 -0.8166
S SKP 5
ID FL3FEAGS0036
FORMULA C28H32O15
EXACTMASS 608.174120354
AVERAGEMASS 608.54468
SMILES C(C(O2)C(O)C(C(C(Oc(c5O)c(OC)cc(c35)OC(c(c4)ccc(OC)c4)=CC3=O)2)OC(O1)C(C(O)C(C1)O)O)O)O
M END