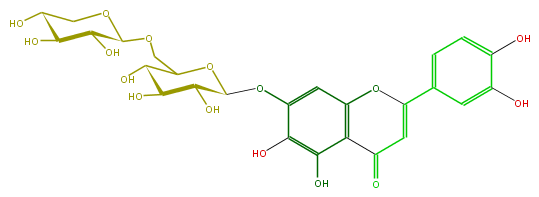
Mol:FL3FECGS0044
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | 4.0411 1.0751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0411 1.0751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0411 0.2501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0411 0.2501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7556 -0.1624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7556 -0.1624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4700 0.2501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4700 0.2501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4700 1.0751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4700 1.0751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7556 1.4876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7556 1.4876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3266 -0.1624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3266 -0.1624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6121 0.2501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6121 0.2501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8976 -0.1624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8976 -0.1624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8976 -0.9875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8976 -0.9875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6121 -1.4000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6121 -1.4000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3266 -0.9875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3266 -0.9875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1831 0.2501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1831 0.2501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4686 -0.1624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4686 -0.1624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4686 -0.9875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4686 -0.9875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1831 -1.4000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1831 -1.4000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6121 -2.1183 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6121 -2.1183 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2458 0.2501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2458 0.2501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1831 -2.0859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1831 -2.0859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1757 1.4767 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1757 1.4767 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1346 -0.1335 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1346 -0.1335 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1845 -1.3646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1845 -1.3646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6359 2.1183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6359 2.1183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2234 1.4039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2234 1.4039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4253 1.6130 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4253 1.6130 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6320 1.3862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6320 1.3862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0444 2.1007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0444 2.1007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8427 1.8917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8427 1.8917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9721 1.1435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9721 1.1435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7987 1.4220 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7987 1.4220 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.1757 1.8450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.1757 1.8450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0783 0.8024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0783 0.8024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6658 0.0880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6658 0.0880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8677 0.2971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8677 0.2971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0744 0.0703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0744 0.0703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4869 0.7848 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4869 0.7848 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2851 0.5758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2851 0.5758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8531 1.0156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8531 1.0156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9795 1.4755 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9795 1.4755 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6013 0.4830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6013 0.4830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2368 0.0567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2368 0.0567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5002 -0.2630 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5002 -0.2630 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 4 21 1 0 0 0 0 | + | 4 21 1 0 0 0 0 |
| − | 15 22 1 0 0 0 0 | + | 15 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 32 40 1 0 0 0 0 | + | 32 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 18 1 0 0 0 0 | + | 35 18 1 0 0 0 0 |
| − | 26 39 1 0 0 0 0 | + | 26 39 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FECGS0044 | + | ID FL3FECGS0044 |
| − | FORMULA C26H28O16 | + | FORMULA C26H28O16 |
| − | EXACTMASS 596.137734848 | + | EXACTMASS 596.137734848 |
| − | AVERAGEMASS 596.49092 | + | AVERAGEMASS 596.49092 |
| − | SMILES C(C5O)(C(OC(C5O)COC(O4)C(O)C(C(O)C4)O)Oc(c1)c(O)c(O)c(C2=O)c(OC(c(c3)ccc(c3O)O)=C2)1)O | + | SMILES C(C5O)(C(OC(C5O)COC(O4)C(O)C(C(O)C4)O)Oc(c1)c(O)c(O)c(C2=O)c(OC(c(c3)ccc(c3O)O)=C2)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
4.0411 1.0751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0411 0.2501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7556 -0.1624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4700 0.2501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4700 1.0751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7556 1.4876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3266 -0.1624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6121 0.2501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8976 -0.1624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8976 -0.9875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6121 -1.4000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3266 -0.9875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1831 0.2501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4686 -0.1624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4686 -0.9875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1831 -1.4000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6121 -2.1183 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2458 0.2501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1831 -2.0859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.1757 1.4767 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.1346 -0.1335 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1845 -1.3646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.6359 2.1183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2234 1.4039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4253 1.6130 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6320 1.3862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0444 2.1007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8427 1.8917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9721 1.1435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.7987 1.4220 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.1757 1.8450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0783 0.8024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6658 0.0880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8677 0.2971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0744 0.0703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4869 0.7848 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2851 0.5758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8531 1.0156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9795 1.4755 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6013 0.4830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2368 0.0567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5002 -0.2630 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
16 19 1 0 0 0 0
5 20 1 0 0 0 0
4 21 1 0 0 0 0
15 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
23 31 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
32 40 1 0 0 0 0
33 41 1 0 0 0 0
34 42 1 0 0 0 0
35 18 1 0 0 0 0
26 39 1 0 0 0 0
S SKP 5
ID FL3FECGS0044
FORMULA C26H28O16
EXACTMASS 596.137734848
AVERAGEMASS 596.49092
SMILES C(C5O)(C(OC(C5O)COC(O4)C(O)C(C(O)C4)O)Oc(c1)c(O)c(O)c(C2=O)c(OC(c(c3)ccc(c3O)O)=C2)1)O
M END