Mol:FL3FFAGS0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.0314 0.8515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0314 0.8515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0314 0.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0314 0.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5978 -0.2382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5978 -0.2382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2270 0.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2270 0.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2270 0.8515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2270 0.8515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5978 1.2147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5978 1.2147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8560 -0.2382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8560 -0.2382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4852 0.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4852 0.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4852 0.8515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4852 0.8515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8560 1.2147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8560 1.2147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8560 -0.8046 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8560 -0.8046 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1141 1.2146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1141 1.2146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7552 0.8444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7552 0.8444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3964 1.2146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3964 1.2146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3964 1.9549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3964 1.9549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7552 2.3252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7552 2.3252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1141 1.9549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1141 1.9549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5978 -0.8049 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5978 -0.8049 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0712 2.3858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0712 2.3858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7558 1.2698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7558 1.2698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5978 2.0457 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5978 2.0457 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3317 1.5250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3317 1.5250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6125 0.5757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6125 0.5757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5769 0.9784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5769 0.9784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5776 0.9893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5776 0.9893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3038 1.7155 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3038 1.7155 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3617 1.3357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3617 1.3357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0712 1.2231 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0712 1.2231 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6291 0.7467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6291 0.7467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7140 0.3316 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7140 0.3316 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7925 -0.5817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7925 -0.5817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3578 -1.5608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3578 -1.5608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2706 -1.2500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2706 -1.2500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1768 -1.5608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1768 -1.5608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6114 -0.5817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6114 -0.5817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6986 -0.8921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6986 -0.8921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0011 -1.9721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0011 -1.9721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9541 -1.1914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9541 -1.1914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6612 -0.6577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6612 -0.6577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1768 -2.3858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1768 -2.3858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1025 1.9861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1025 1.9861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1059 1.4108 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1059 1.4108 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 18 3 1 0 0 0 0 | + | 18 3 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 1 1 0 0 0 0 | + | 20 1 1 0 0 0 0 |
| − | 6 21 1 0 0 0 0 | + | 6 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 33 37 1 0 0 0 0 | + | 33 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 31 39 1 0 0 0 0 | + | 31 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 27 41 1 0 0 0 0 | + | 27 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 46 0.7408 -0.6504 | + | M SBV 1 46 0.7408 -0.6504 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FFAGS0003 | + | ID FL3FFAGS0003 |
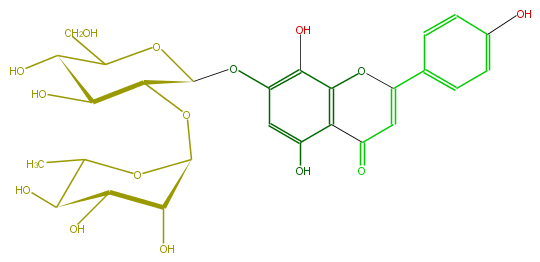
| − | FORMULA C27H30O15 | + | FORMULA C27H30O15 |
| − | EXACTMASS 594.15847029 | + | EXACTMASS 594.15847029 |
| − | AVERAGEMASS 594.5181 | + | AVERAGEMASS 594.5181 |
| − | SMILES C(C(O)1)(C(OC(C2O)C(Oc(c3)c(c(O4)c(C(C=C4c(c5)ccc(O)c5)=O)c(O)3)O)OC(C(O)2)CO)OC(C)C1O)O | + | SMILES C(C(O)1)(C(OC(C2O)C(Oc(c3)c(c(O4)c(C(C=C4c(c5)ccc(O)c5)=O)c(O)3)O)OC(C(O)2)CO)OC(C)C1O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-0.0314 0.8515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0314 0.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5978 -0.2382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2270 0.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2270 0.8515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5978 1.2147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8560 -0.2382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4852 0.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4852 0.8515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8560 1.2147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8560 -0.8046 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1141 1.2146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7552 0.8444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3964 1.2146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3964 1.9549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7552 2.3252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1141 1.9549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5978 -0.8049 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0712 2.3858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7558 1.2698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5978 2.0457 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3317 1.5250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6125 0.5757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5769 0.9784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5776 0.9893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3038 1.7155 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3617 1.3357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0712 1.2231 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6291 0.7467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7140 0.3316 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7925 -0.5817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3578 -1.5608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2706 -1.2500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1768 -1.5608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6114 -0.5817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6986 -0.8921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0011 -1.9721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9541 -1.1914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6612 -0.6577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1768 -2.3858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1025 1.9861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1059 1.4108 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
18 3 1 0 0 0 0
19 15 1 0 0 0 0
20 1 1 0 0 0 0
6 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
35 30 1 0 0 0 0
33 37 1 0 0 0 0
32 38 1 0 0 0 0
31 39 1 0 0 0 0
34 40 1 0 0 0 0
25 20 1 0 0 0 0
41 42 1 0 0 0 0
27 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 46
M SMT 1 ^CH2OH
M SBV 1 46 0.7408 -0.6504
S SKP 5
ID FL3FFAGS0003
FORMULA C27H30O15
EXACTMASS 594.15847029
AVERAGEMASS 594.5181
SMILES C(C(O)1)(C(OC(C2O)C(Oc(c3)c(c(O4)c(C(C=C4c(c5)ccc(O)c5)=O)c(O)3)O)OC(C(O)2)CO)OC(C)C1O)O
M END