Mol:FL3FFCGS0014
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 48 52 0 0 0 0 0 0 0 0999 V2000 | + | 48 52 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.2984 0.5700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2984 0.5700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2984 0.0079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2984 0.0079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7494 -0.2525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7494 -0.2525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2005 0.0079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2005 0.0079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2005 0.5287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2005 0.5287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7494 0.7892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7494 0.7892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6516 -0.2525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6516 -0.2525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1026 0.0079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1026 0.0079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1026 0.5287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1026 0.5287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6516 0.7892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6516 0.7892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6516 -0.6586 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6516 -0.6586 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5535 0.7891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5535 0.7891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0132 0.5236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0132 0.5236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4730 0.7891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4730 0.7891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4730 1.3199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4730 1.3199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0132 1.5853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0132 1.5853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5535 1.3199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5535 1.3199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7494 -0.7723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7494 -0.7723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1545 0.8689 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1545 0.8689 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7494 1.3676 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7494 1.3676 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0011 2.2456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0011 2.2456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8712 0.7159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8712 0.7159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3556 0.0352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3556 0.0352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6131 0.3240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6131 0.3240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8966 0.3317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8966 0.3317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4173 0.8525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4173 0.8525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1757 0.5801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1757 0.5801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4477 0.3830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4477 0.3830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1594 -0.0039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1594 -0.0039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1877 -0.3902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1877 -0.3902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9082 -1.7093 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9082 -1.7093 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1187 -1.1198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1187 -1.1198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6031 -1.8004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6031 -1.8004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8606 -1.5116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8606 -1.5116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1441 -1.5039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1441 -1.5039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6648 -0.9832 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6648 -0.9832 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4232 -1.2555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4232 -1.2555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6031 -2.2456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6031 -2.2456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4352 -2.2258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4352 -2.2258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0040 -0.6747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0040 -0.6747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7017 -0.8616 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7017 -0.8616 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2816 -0.7063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2816 -0.7063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2816 -0.2021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2816 -0.2021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7803 -0.9942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7803 -0.9942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4713 1.3875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4713 1.3875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7568 0.9750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7568 0.9750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0658 1.7495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0658 1.7495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7803 1.3370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7803 1.3370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 6 20 1 0 0 0 0 | + | 6 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 37 40 1 0 0 0 0 | + | 37 40 1 0 0 0 0 |
| − | 25 19 1 0 0 0 0 | + | 25 19 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 2 0 0 0 0 | + | 42 43 2 0 0 0 0 |
| − | 42 44 1 0 0 0 0 | + | 42 44 1 0 0 0 0 |
| − | 27 45 1 0 0 0 0 | + | 27 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 15 47 1 0 0 0 0 | + | 15 47 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 45 46 | + | M SAL 1 2 45 46 |
| − | M SBL 1 1 49 | + | M SBL 1 1 49 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 49 -7.7439 6.2756 | + | M SBV 1 49 -7.7439 6.2756 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 47 48 | + | M SAL 2 2 47 48 |
| − | M SBL 2 1 51 | + | M SBL 2 1 51 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 51 -6.8555 5.8979 | + | M SBV 2 51 -6.8555 5.8979 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FFCGS0014 | + | ID FL3FFCGS0014 |
| − | KNApSAcK_ID C00004432 | + | KNApSAcK_ID C00004432 |
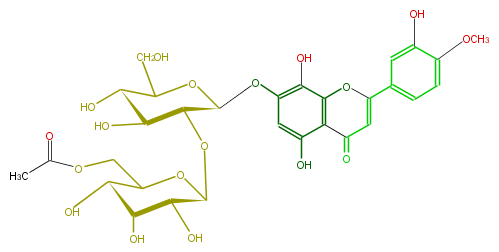
| − | NAME 8-Hydroxyluteolin 4'-methyl ether 7-(6'''-acetylallosyl-(1->2)-glucoside | + | NAME 8-Hydroxyluteolin 4'-methyl ether 7-(6'''-acetylallosyl-(1->2)-glucoside |
| − | CAS_RN 93753-29-8 | + | CAS_RN 93753-29-8 |
| − | FORMULA C30H34O18 | + | FORMULA C30H34O18 |
| − | EXACTMASS 682.174514284 | + | EXACTMASS 682.174514284 |
| − | AVERAGEMASS 682.58016 | + | AVERAGEMASS 682.58016 |
| − | SMILES O(CC(O1)C(C(C(O)C1OC(C2Oc(c5)c(c(c4c5O)OC(=CC(=O)4)c(c3)cc(O)c(c3)OC)O)C(O)C(O)C(CO)O2)O)O)C(C)=O | + | SMILES O(CC(O1)C(C(C(O)C1OC(C2Oc(c5)c(c(c4c5O)OC(=CC(=O)4)c(c3)cc(O)c(c3)OC)O)C(O)C(O)C(CO)O2)O)O)C(C)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
48 52 0 0 0 0 0 0 0 0999 V2000
0.2984 0.5700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2984 0.0079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7494 -0.2525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2005 0.0079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2005 0.5287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7494 0.7892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6516 -0.2525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1026 0.0079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1026 0.5287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6516 0.7892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6516 -0.6586 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5535 0.7891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0132 0.5236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4730 0.7891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4730 1.3199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0132 1.5853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5535 1.3199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7494 -0.7723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1545 0.8689 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7494 1.3676 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0011 2.2456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8712 0.7159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3556 0.0352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6131 0.3240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8966 0.3317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4173 0.8525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1757 0.5801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4477 0.3830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1594 -0.0039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1877 -0.3902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9082 -1.7093 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1187 -1.1198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6031 -1.8004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8606 -1.5116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1441 -1.5039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6648 -0.9832 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4232 -1.2555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6031 -2.2456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4352 -2.2258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0040 -0.6747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7017 -0.8616 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2816 -0.7063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2816 -0.2021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7803 -0.9942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4713 1.3875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7568 0.9750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0658 1.7495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7803 1.3370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
1 19 1 0 0 0 0
6 20 1 0 0 0 0
16 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
33 38 1 0 0 0 0
34 39 1 0 0 0 0
31 32 1 0 0 0 0
35 30 1 0 0 0 0
37 40 1 0 0 0 0
25 19 1 0 0 0 0
40 41 1 0 0 0 0
41 42 1 0 0 0 0
42 43 2 0 0 0 0
42 44 1 0 0 0 0
27 45 1 0 0 0 0
45 46 1 0 0 0 0
15 47 1 0 0 0 0
47 48 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 45 46
M SBL 1 1 49
M SMT 1 ^CH2OH
M SBV 1 49 -7.7439 6.2756
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 47 48
M SBL 2 1 51
M SMT 2 OCH3
M SBV 2 51 -6.8555 5.8979
S SKP 8
ID FL3FFCGS0014
KNApSAcK_ID C00004432
NAME 8-Hydroxyluteolin 4'-methyl ether 7-(6'''-acetylallosyl-(1->2)-glucoside
CAS_RN 93753-29-8
FORMULA C30H34O18
EXACTMASS 682.174514284
AVERAGEMASS 682.58016
SMILES O(CC(O1)C(C(C(O)C1OC(C2Oc(c5)c(c(c4c5O)OC(=CC(=O)4)c(c3)cc(O)c(c3)OC)O)C(O)C(O)C(CO)O2)O)O)C(C)=O
M END