Mol:FL3FGLNS0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 29 31 0 0 0 0 0 0 0 0999 V2000 | + | 29 31 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.2502 -0.9188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2502 -0.9188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6175 -0.8072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6175 -0.8072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3978 -0.2036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3978 -0.2036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8107 0.2885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8107 0.2885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4434 0.1769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4434 0.1769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6631 -0.4267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6631 -0.4267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5910 0.8921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5910 0.8921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0039 1.3842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0039 1.3842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6366 1.2726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6366 1.2726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8563 0.6690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8563 0.6690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0978 0.9790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0978 0.9790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0493 1.7645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0493 1.7645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8254 2.3796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8254 2.3796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2462 2.8812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2462 2.8812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8909 2.7675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8909 2.7675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1149 2.1523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1149 2.1523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6940 1.6508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6940 1.6508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7655 -0.0921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7655 -0.0921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9179 1.0358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9179 1.0358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3117 3.2689 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3117 3.2689 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0295 4.0441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0295 4.0441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9041 3.8209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9041 3.8209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5620 4.7605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5620 4.7605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2757 -0.2524 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2757 -0.2524 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2375 0.0212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2375 0.0212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7974 -1.3781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7974 -1.3781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5634 -2.0210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5634 -2.0210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7090 -1.5217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7090 -1.5217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2966 -0.8071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2966 -0.8071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 17 19 1 0 0 0 0 | + | 17 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 14 22 1 0 0 0 0 | + | 14 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 6 24 1 0 0 0 0 | + | 6 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 1 26 1 0 0 0 0 | + | 1 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 2 28 1 0 0 0 0 | + | 2 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | M STY 1 5 SUP | + | M STY 1 5 SUP |
| − | M SLB 1 5 5 | + | M SLB 1 5 5 |
| − | M SAL 5 2 28 29 | + | M SAL 5 2 28 29 |
| − | M SBL 5 1 30 | + | M SBL 5 1 30 |
| − | M SMT 5 OCH3 | + | M SMT 5 OCH3 |
| − | M SVB 5 30 -2.9555 -0.3996 | + | M SVB 5 30 -2.9555 -0.3996 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 26 27 | + | M SAL 4 2 26 27 |
| − | M SBL 4 1 28 | + | M SBL 4 1 28 |
| − | M SMT 4 OCH3 | + | M SMT 4 OCH3 |
| − | M SVB 4 28 -2.5983 0.77 | + | M SVB 4 28 -2.5983 0.77 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 24 25 | + | M SAL 3 2 24 25 |
| − | M SBL 3 1 26 | + | M SBL 3 1 26 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 26 -1.4067 1.0455 | + | M SVB 3 26 -1.4067 1.0455 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 22 23 | + | M SAL 2 2 22 23 |
| − | M SBL 2 1 24 | + | M SBL 2 1 24 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 24 1.8838 0.2661 | + | M SVB 2 24 1.8838 0.2661 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 20 21 | + | M SAL 1 2 20 21 |
| − | M SBL 1 1 22 | + | M SBL 1 1 22 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 22 2.241 1.4543 | + | M SVB 1 22 2.241 1.4543 |
| − | S SKP 8 | + | S SKP 8 |
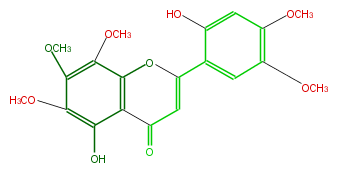
| − | ID FL3FGLNS0003 | + | ID FL3FGLNS0003 |
| − | KNApSAcK_ID C00003967 | + | KNApSAcK_ID C00003967 |
| − | NAME Agehoustin F | + | NAME Agehoustin F |
| − | CAS_RN 111316-38-2 | + | CAS_RN 111316-38-2 |
| − | FORMULA C20H20O9 | + | FORMULA C20H20O9 |
| − | EXACTMASS 404.11073223799997 | + | EXACTMASS 404.11073223799997 |
| − | AVERAGEMASS 404.3674 | + | AVERAGEMASS 404.3674 |
| − | SMILES O(C)c(c31)c(c(c(c(C(=O)C=C(O3)c(c2)c(cc(OC)c2OC)O)1)O)OC)OC | + | SMILES O(C)c(c31)c(c(c(c(C(=O)C=C(O3)c(c2)c(cc(OC)c2OC)O)1)O)OC)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/ 29 31 0 0 0 0 0 0 0 0999 V2000 -3.2502 -0.9188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -2.6175 -0.8072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -2.3978 -0.2036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -2.8107 0.2885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -3.4434 0.1769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -3.6631 -0.4267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -2.5910 0.8921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -3.0039 1.3842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -3.6366 1.2726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -3.8563 0.6690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 -2.0978 0.9790 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 -4.0493 1.7645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -3.8254 2.3796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -4.2462 2.8812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -4.8909 2.7675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -5.1149 2.1523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -4.6940 1.6508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -1.7655 -0.0921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 -4.9179 1.0358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 -5.3117 3.2689 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 -5.0295 4.0441 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -3.9041 3.8209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 -3.5620 4.7605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -4.2757 -0.2524 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 -5.2375 0.0212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -3.7974 -1.3781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 -4.5634 -2.0210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -2.7090 -1.5217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 -2.2966 -0.8071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 1 2 1 0 0 0 0 2 3 2 0 0 0 0 3 4 1 0 0 0 0 4 5 2 0 0 0 0 5 6 1 0 0 0 0 6 1 2 0 0 0 0 4 7 1 0 0 0 0 7 8 1 0 0 0 0 8 9 2 0 0 0 0 9 10 1 0 0 0 0 10 5 1 0 0 0 0 7 11 2 0 0 0 0 9 12 1 0 0 0 0 12 13 2 0 0 0 0 13 14 1 0 0 0 0 14 15 2 0 0 0 0 15 16 1 0 0 0 0 16 17 2 0 0 0 0 17 12 1 0 0 0 0 3 18 1 0 0 0 0 17 19 1 0 0 0 0 15 20 1 0 0 0 0 20 21 1 0 0 0 0 14 22 1 0 0 0 0 22 23 1 0 0 0 0 6 24 1 0 0 0 0 24 25 1 0 0 0 0 1 26 1 0 0 0 0 26 27 1 0 0 0 0 2 28 1 0 0 0 0 28 29 1 0 0 0 0 M STY 1 5 SUP M SLB 1 5 5 M SAL 5 2 28 29 M SBL 5 1 30 M SMT 5 OCH3 M SVB 5 30 -2.9555 -0.3996 M STY 1 4 SUP M SLB 1 4 4 M SAL 4 2 26 27 M SBL 4 1 28 M SMT 4 OCH3 M SVB 4 28 -2.5983 0.77 M STY 1 3 SUP M SLB 1 3 3 M SAL 3 2 24 25 M SBL 3 1 26 M SMT 3 OCH3 M SVB 3 26 -1.4067 1.0455 M STY 1 2 SUP M SLB 1 2 2 M SAL 2 2 22 23 M SBL 2 1 24 M SMT 2 OCH3 M SVB 2 24 1.8838 0.2661 M STY 1 1 SUP M SLB 1 1 1 M SAL 1 2 20 21 M SBL 1 1 22 M SMT 1 OCH3 M SVB 1 22 2.241 1.4543 S SKP 8 ID FL3FGLNS0003 KNApSAcK_ID C00003967 NAME Agehoustin F CAS_RN 111316-38-2 FORMULA C20H20O9 EXACTMASS 404.11073223799997 AVERAGEMASS 404.3674 SMILES O(C)c(c31)c(c(c(c(C(=O)C=C(O3)c(c2)c(cc(OC)c2OC)O)1)O)OC)OC M END