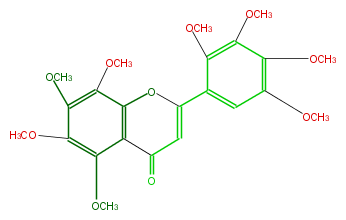
Mol:FL3FGLNS0008
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 33 35 0 0 0 0 0 0 0 0999 V2000 | + | 33 35 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.2410 -0.1713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2410 -0.1713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2410 -0.8137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2410 -0.8137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6847 -1.1349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6847 -1.1349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1284 -0.8137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1284 -0.8137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1284 -0.1713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1284 -0.1713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6847 0.1499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6847 0.1499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5721 -1.1349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5721 -1.1349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0158 -0.8137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0158 -0.8137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0158 -0.1713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0158 -0.1713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5721 0.1499 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5721 0.1499 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5721 -1.6357 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5721 -1.6357 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5403 0.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5403 0.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1072 -0.1776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1072 -0.1776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6742 0.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6742 0.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6742 0.8044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6742 0.8044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1072 1.1318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1072 1.1318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5403 0.8044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5403 0.8044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3907 1.7079 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3907 1.7079 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8322 2.6051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8322 2.6051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4067 0.7229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4067 0.7229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9702 1.6226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9702 1.6226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5402 -0.3503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5402 -0.3503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4062 -0.8504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4062 -0.8504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6847 -2.1349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6847 -2.1349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6847 -3.1349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6847 -3.1349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1830 1.4232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1830 1.4232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3170 2.2892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3170 2.2892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9555 -0.7222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9555 -0.7222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9474 -0.5952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9474 -0.5952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6742 0.8044 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6742 0.8044 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6742 0.8044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6742 0.8044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5983 0.4474 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5983 0.4474 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0984 1.3134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0984 1.3134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 16 18 1 0 0 0 0 | + | 16 18 1 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 6 20 1 0 0 0 0 | + | 6 20 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 14 22 1 0 0 0 0 | + | 14 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 3 24 1 0 0 0 0 | + | 3 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 17 26 1 0 0 0 0 | + | 17 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 2 28 1 0 0 0 0 | + | 2 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 15 30 1 0 0 0 0 | + | 15 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 1 32 1 0 0 0 0 | + | 1 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | M STY 1 8 SUP | + | M STY 1 8 SUP |
| − | M SLB 1 8 8 | + | M SLB 1 8 8 |
| − | M SAL 8 2 32 33 | + | M SAL 8 2 32 33 |
| − | M SBL 8 1 34 | + | M SBL 8 1 34 |
| − | M SMT 8 OCH3 | + | M SMT 8 OCH3 |
| − | M SVB 8 34 -2.5983 0.4474 | + | M SVB 8 34 -2.5983 0.4474 |
| − | M STY 1 7 SUP | + | M STY 1 7 SUP |
| − | M SLB 1 7 7 | + | M SLB 1 7 7 |
| − | M SAL 7 2 30 31 | + | M SAL 7 2 30 31 |
| − | M SBL 7 1 32 | + | M SBL 7 1 32 |
| − | M SMT 7 OCH3 | + | M SMT 7 OCH3 |
| − | M SVB 7 32 2.241 1.1317 | + | M SVB 7 32 2.241 1.1317 |
| − | M STY 1 6 SUP | + | M STY 1 6 SUP |
| − | M SLB 1 6 6 | + | M SLB 1 6 6 |
| − | M SAL 6 2 28 29 | + | M SAL 6 2 28 29 |
| − | M SBL 6 1 30 | + | M SBL 6 1 30 |
| − | M SMT 6 OCH3 | + | M SMT 6 OCH3 |
| − | M SVB 6 30 -2.9555 -0.7222 | + | M SVB 6 30 -2.9555 -0.7222 |
| − | M STY 1 5 SUP | + | M STY 1 5 SUP |
| − | M SLB 1 5 5 | + | M SLB 1 5 5 |
| − | M SAL 5 2 26 27 | + | M SAL 5 2 26 27 |
| − | M SBL 5 1 28 | + | M SBL 5 1 28 |
| − | M SMT 5 OCH3 | + | M SMT 5 OCH3 |
| − | M SVB 5 28 0.183 1.4232 | + | M SVB 5 28 0.183 1.4232 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 24 25 | + | M SAL 4 2 24 25 |
| − | M SBL 4 1 26 | + | M SBL 4 1 26 |
| − | M SMT 4 OCH3 | + | M SMT 4 OCH3 |
| − | M SVB 4 26 -2.1212 -1.2954 | + | M SVB 4 26 -2.1212 -1.2954 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 22 23 | + | M SAL 3 2 22 23 |
| − | M SBL 3 1 24 | + | M SBL 3 1 24 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 24 1.8838 -0.0565 | + | M SVB 3 24 1.8838 -0.0565 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 20 21 | + | M SAL 2 2 20 21 |
| − | M SBL 2 1 22 | + | M SBL 2 1 22 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 22 -1.4067 0.7229 | + | M SVB 2 22 -1.4067 0.7229 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 18 19 | + | M SAL 1 2 18 19 |
| − | M SBL 1 1 20 | + | M SBL 1 1 20 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 20 1.3907 1.7079 | + | M SVB 1 20 1.3907 1.7079 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FGLNS0008 | + | ID FL3FGLNS0008 |
| − | KNApSAcK_ID C00003982 | + | KNApSAcK_ID C00003982 |
| − | NAME Agehoustin A | + | NAME Agehoustin A |
| − | CAS_RN 87402-97-8 | + | CAS_RN 87402-97-8 |
| − | FORMULA C23H26O10 | + | FORMULA C23H26O10 |
| − | EXACTMASS 462.152597052 | + | EXACTMASS 462.152597052 |
| − | AVERAGEMASS 462.44654 | + | AVERAGEMASS 462.44654 |
| − | SMILES c(C2=O)(c(OC)3)c(c(OC)c(OC)c(OC)3)OC(=C2)c(c1)c(OC)c(OC)c(OC)c(OC)1 | + | SMILES c(C2=O)(c(OC)3)c(c(OC)c(OC)c(OC)3)OC(=C2)c(c1)c(OC)c(OC)c(OC)c(OC)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
33 35 0 0 0 0 0 0 0 0999 V2000
-2.2410 -0.1713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2410 -0.8137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6847 -1.1349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1284 -0.8137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1284 -0.1713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6847 0.1499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5721 -1.1349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0158 -0.8137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0158 -0.1713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5721 0.1499 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5721 -1.6357 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5403 0.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1072 -0.1776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6742 0.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6742 0.8044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1072 1.1318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5403 0.8044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3907 1.7079 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8322 2.6051 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4067 0.7229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9702 1.6226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5402 -0.3503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4062 -0.8504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6847 -2.1349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6847 -3.1349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1830 1.4232 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3170 2.2892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9555 -0.7222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9474 -0.5952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6742 0.8044 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6742 0.8044 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5983 0.4474 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0984 1.3134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
16 18 1 0 0 0 0
18 19 1 0 0 0 0
6 20 1 0 0 0 0
20 21 1 0 0 0 0
14 22 1 0 0 0 0
22 23 1 0 0 0 0
3 24 1 0 0 0 0
24 25 1 0 0 0 0
17 26 1 0 0 0 0
26 27 1 0 0 0 0
2 28 1 0 0 0 0
28 29 1 0 0 0 0
15 30 1 0 0 0 0
30 31 1 0 0 0 0
1 32 1 0 0 0 0
32 33 1 0 0 0 0
M STY 1 8 SUP
M SLB 1 8 8
M SAL 8 2 32 33
M SBL 8 1 34
M SMT 8 OCH3
M SVB 8 34 -2.5983 0.4474
M STY 1 7 SUP
M SLB 1 7 7
M SAL 7 2 30 31
M SBL 7 1 32
M SMT 7 OCH3
M SVB 7 32 2.241 1.1317
M STY 1 6 SUP
M SLB 1 6 6
M SAL 6 2 28 29
M SBL 6 1 30
M SMT 6 OCH3
M SVB 6 30 -2.9555 -0.7222
M STY 1 5 SUP
M SLB 1 5 5
M SAL 5 2 26 27
M SBL 5 1 28
M SMT 5 OCH3
M SVB 5 28 0.183 1.4232
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 24 25
M SBL 4 1 26
M SMT 4 OCH3
M SVB 4 26 -2.1212 -1.2954
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 22 23
M SBL 3 1 24
M SMT 3 OCH3
M SVB 3 24 1.8838 -0.0565
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 20 21
M SBL 2 1 22
M SMT 2 OCH3
M SVB 2 22 -1.4067 0.7229
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 18 19
M SBL 1 1 20
M SMT 1 OCH3
M SVB 1 20 1.3907 1.7079
S SKP 8
ID FL3FGLNS0008
KNApSAcK_ID C00003982
NAME Agehoustin A
CAS_RN 87402-97-8
FORMULA C23H26O10
EXACTMASS 462.152597052
AVERAGEMASS 462.44654
SMILES c(C2=O)(c(OC)3)c(c(OC)c(OC)c(OC)3)OC(=C2)c(c1)c(OC)c(OC)c(OC)c(OC)1
M END