Mol:FL4DACGS0016
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 33 36 0 0 0 0 0 0 0 0999 V2000 | + | 33 36 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.7407 -0.7628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7407 -0.7628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2198 -1.0635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2198 -1.0635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3011 -0.7628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3011 -0.7628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3011 -0.1613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3011 -0.1613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2198 0.1394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2198 0.1394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7407 -0.1613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7407 -0.1613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8220 -1.0635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8220 -1.0635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3429 -0.7628 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.3429 -0.7628 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.3429 -0.1613 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 1.3429 -0.1613 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 0.8220 0.1394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8220 0.1394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8220 -1.5874 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8220 -1.5874 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9226 0.1381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9226 0.1381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4513 -0.1671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4513 -0.1671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9800 0.1381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9800 0.1381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9800 0.7486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9800 0.7486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4513 1.0539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4513 1.0539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9226 0.7486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9226 0.7486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2739 0.1465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2739 0.1465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8111 -1.3395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8111 -1.3395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5087 1.0539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5087 1.0539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3164 -0.0186 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.3164 -0.0186 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.9723 -0.4728 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.9723 -0.4728 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.4768 -0.2801 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.4768 -0.2801 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.9986 -0.2750 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.9986 -0.2750 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.3461 0.0726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3461 0.0726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8522 -0.1092 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.8522 -0.1092 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -4.2873 0.2210 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2873 0.2210 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5087 -0.4989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5087 -0.4989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1928 -0.7567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1928 -0.7567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2198 -1.6644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2198 -1.6644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4513 1.6644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4513 1.6644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8684 0.2971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8684 0.2971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7537 0.7621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7537 0.7621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 6 0 0 0 | + | 9 12 1 6 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 6 18 1 0 0 0 0 | + | 6 18 1 0 0 0 0 |
| − | 8 19 1 1 0 0 0 | + | 8 19 1 1 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 18 1 0 0 0 0 | + | 24 18 1 0 0 0 0 |
| − | 2 30 1 0 0 0 0 | + | 2 30 1 0 0 0 0 |
| − | 16 31 1 0 0 0 0 | + | 16 31 1 0 0 0 0 |
| − | 26 32 1 0 0 0 0 | + | 26 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 32 33 | + | M SAL 1 2 32 33 |
| − | M SBL 1 1 35 | + | M SBL 1 1 35 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 35 -2.6952 0.2659 | + | M SVB 1 35 -2.6952 0.2659 |
| − | S SKP 8 | + | S SKP 8 |
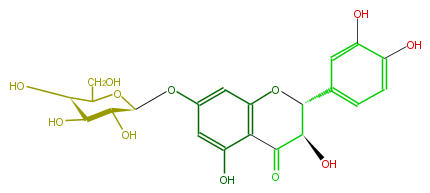
| − | ID FL4DACGS0016 | + | ID FL4DACGS0016 |
| − | KNApSAcK_ID C00008708 | + | KNApSAcK_ID C00008708 |
| − | NAME Taxifolin 7-galactoside | + | NAME Taxifolin 7-galactoside |
| − | CAS_RN 72442-45-6 | + | CAS_RN 72442-45-6 |
| − | FORMULA C21H22O12 | + | FORMULA C21H22O12 |
| − | EXACTMASS 466.111126168 | + | EXACTMASS 466.111126168 |
| − | AVERAGEMASS 466.39218 | + | AVERAGEMASS 466.39218 |
| − | SMILES c(O)(c(O)4)ccc(c4)[C@H]([C@H]3O)Oc(c1)c(C3=O)c(cc(O[C@H](O2)[C@@H](O)[C@@H](O)[C@@H](O)C2CO)1)O | + | SMILES c(O)(c(O)4)ccc(c4)[C@H]([C@H]3O)Oc(c1)c(C3=O)c(cc(O[C@H](O2)[C@@H](O)[C@@H](O)[C@@H](O)C2CO)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
33 36 0 0 0 0 0 0 0 0999 V2000
-0.7407 -0.7628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2198 -1.0635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3011 -0.7628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3011 -0.1613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2198 0.1394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7407 -0.1613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8220 -1.0635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3429 -0.7628 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.3429 -0.1613 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
0.8220 0.1394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8220 -1.5874 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9226 0.1381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4513 -0.1671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9800 0.1381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9800 0.7486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4513 1.0539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9226 0.7486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2739 0.1465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8111 -1.3395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5087 1.0539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3164 -0.0186 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.9723 -0.4728 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.4768 -0.2801 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.9986 -0.2750 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.3461 0.0726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8522 -0.1092 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-4.2873 0.2210 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5087 -0.4989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1928 -0.7567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2198 -1.6644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4513 1.6644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8684 0.2971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7537 0.7621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
3 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 4 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 6 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
6 18 1 0 0 0 0
8 19 1 1 0 0 0
15 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 18 1 0 0 0 0
2 30 1 0 0 0 0
16 31 1 0 0 0 0
26 32 1 0 0 0 0
32 33 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 32 33
M SBL 1 1 35
M SMT 1 CH2OH
M SVB 1 35 -2.6952 0.2659
S SKP 8
ID FL4DACGS0016
KNApSAcK_ID C00008708
NAME Taxifolin 7-galactoside
CAS_RN 72442-45-6
FORMULA C21H22O12
EXACTMASS 466.111126168
AVERAGEMASS 466.39218
SMILES c(O)(c(O)4)ccc(c4)[C@H]([C@H]3O)Oc(c1)c(C3=O)c(cc(O[C@H](O2)[C@@H](O)[C@@H](O)[C@@H](O)C2CO)1)O
M END