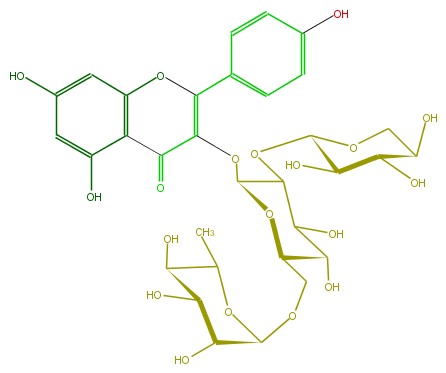
Mol:FL5FAAGL0027
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 51 56 0 0 0 0 0 0 0 0999 V2000 | + | 51 56 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.3618 1.8158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3618 1.8158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3618 1.0064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3618 1.0064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6608 0.6017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6608 0.6017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9598 1.0064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9598 1.0064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9598 1.8158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9598 1.8158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6608 2.2206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6608 2.2206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2588 0.6017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2588 0.6017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5578 1.0064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5578 1.0064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5578 1.8158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5578 1.8158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2588 2.2206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2588 2.2206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2588 -0.0294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2588 -0.0294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1430 2.2204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1430 2.2204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8575 1.8080 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8575 1.8080 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5720 2.2204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5720 2.2204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5720 3.0455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5720 3.0455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8575 3.4579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8575 3.4579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1430 3.0455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1430 3.0455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6608 -0.2075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6608 -0.2075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0626 2.2204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0626 2.2204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2863 3.4578 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2863 3.4578 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2291 0.5562 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2291 0.5562 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1502 -1.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1502 -1.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0643 -1.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0643 -1.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4073 -0.8021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4073 -0.8021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1827 0.0893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1827 0.0893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2685 0.0894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2685 0.0894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9255 -0.5463 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9255 -0.5463 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1840 -0.9479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1840 -0.9479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0905 -1.9976 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0905 -1.9976 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6167 0.6552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6167 0.6552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6422 -1.9160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6422 -1.9160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7190 1.0102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7190 1.0102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2765 0.2744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2765 0.2744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0792 0.5866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0792 0.5866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8539 0.5949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8539 0.5949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2910 1.1579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2910 1.1579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5886 0.7873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5886 0.7873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4513 0.4356 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4513 0.4356 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8088 0.0811 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8088 0.0811 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0626 1.3739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0626 1.3739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1653 -3.1320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1653 -3.1320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7490 -3.1320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7490 -3.1320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0919 -2.4963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0919 -2.4963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1328 -1.6050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1328 -1.6050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1508 -1.6568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1508 -1.6568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4939 -2.2926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4939 -2.2926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1288 -1.0418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1288 -1.0418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4999 -0.9098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4999 -0.9098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3215 -2.1712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3215 -2.1712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7566 -3.4579 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7566 -3.4579 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3833 -2.5880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3833 -2.5880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 8 21 1 0 0 0 0 | + | 8 21 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 22 1 1 0 0 0 | + | 27 22 1 1 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 25 30 1 0 0 0 0 | + | 25 30 1 0 0 0 0 |
| − | 22 31 1 0 0 0 0 | + | 22 31 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 30 32 1 0 0 0 0 | + | 30 32 1 0 0 0 0 |
| − | 35 40 1 0 0 0 0 | + | 35 40 1 0 0 0 0 |
| − | 42 41 1 1 0 0 0 | + | 42 41 1 1 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 1 0 0 0 | + | 45 46 1 1 0 0 0 |
| − | 46 41 1 1 0 0 0 | + | 46 41 1 1 0 0 0 |
| − | 45 47 1 0 0 0 0 | + | 45 47 1 0 0 0 0 |
| − | 44 48 1 0 0 0 0 | + | 44 48 1 0 0 0 0 |
| − | 46 49 1 0 0 0 0 | + | 46 49 1 0 0 0 0 |
| − | 41 50 1 0 0 0 0 | + | 41 50 1 0 0 0 0 |
| − | 42 51 1 0 0 0 0 | + | 42 51 1 0 0 0 0 |
| − | 51 31 1 0 0 0 0 | + | 51 31 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGL0027 | + | ID FL5FAAGL0027 |
| − | FORMULA C32H38O19 | + | FORMULA C32H38O19 |
| − | EXACTMASS 726.200729034 | + | EXACTMASS 726.200729034 |
| − | AVERAGEMASS 726.6327200000001 | + | AVERAGEMASS 726.6327200000001 |
| − | SMILES c(c(O)1)(C(=O)3)c(OC(=C(OC(O5)C(C(O)C(C(COC(C(O)6)OC(C)C(C(O)6)O)5)O)OC(O4)C(O)C(C(O)C4)O)3)c(c2)ccc(c2)O)cc(c1)O | + | SMILES c(c(O)1)(C(=O)3)c(OC(=C(OC(O5)C(C(O)C(C(COC(C(O)6)OC(C)C(C(O)6)O)5)O)OC(O4)C(O)C(C(O)C4)O)3)c(c2)ccc(c2)O)cc(c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
51 56 0 0 0 0 0 0 0 0999 V2000
-3.3618 1.8158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3618 1.0064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6608 0.6017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9598 1.0064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9598 1.8158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6608 2.2206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2588 0.6017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5578 1.0064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5578 1.8158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2588 2.2206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2588 -0.0294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1430 2.2204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8575 1.8080 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5720 2.2204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5720 3.0455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8575 3.4579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1430 3.0455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6608 -0.2075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0626 2.2204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2863 3.4578 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2291 0.5562 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1502 -1.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0643 -1.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4073 -0.8021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1827 0.0893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2685 0.0894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9255 -0.5463 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1840 -0.9479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0905 -1.9976 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6167 0.6552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6422 -1.9160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7190 1.0102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2765 0.2744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0792 0.5866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8539 0.5949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2910 1.1579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5886 0.7873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4513 0.4356 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8088 0.0811 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0626 1.3739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1653 -3.1320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7490 -3.1320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0919 -2.4963 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1328 -1.6050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1508 -1.6568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4939 -2.2926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1288 -1.0418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4999 -0.9098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3215 -2.1712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7566 -3.4579 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3833 -2.5880 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
1 19 1 0 0 0 0
15 20 1 0 0 0 0
8 21 1 0 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 1 0 0 0
27 22 1 1 0 0 0
24 28 1 0 0 0 0
23 29 1 0 0 0 0
26 21 1 0 0 0 0
25 30 1 0 0 0 0
22 31 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
33 38 1 0 0 0 0
34 39 1 0 0 0 0
30 32 1 0 0 0 0
35 40 1 0 0 0 0
42 41 1 1 0 0 0
42 43 1 0 0 0 0
43 44 1 0 0 0 0
44 45 1 0 0 0 0
45 46 1 1 0 0 0
46 41 1 1 0 0 0
45 47 1 0 0 0 0
44 48 1 0 0 0 0
46 49 1 0 0 0 0
41 50 1 0 0 0 0
42 51 1 0 0 0 0
51 31 1 0 0 0 0
S SKP 5
ID FL5FAAGL0027
FORMULA C32H38O19
EXACTMASS 726.200729034
AVERAGEMASS 726.6327200000001
SMILES c(c(O)1)(C(=O)3)c(OC(=C(OC(O5)C(C(O)C(C(COC(C(O)6)OC(C)C(C(O)6)O)5)O)OC(O4)C(O)C(C(O)C4)O)3)c(c2)ccc(c2)O)cc(c1)O
M END