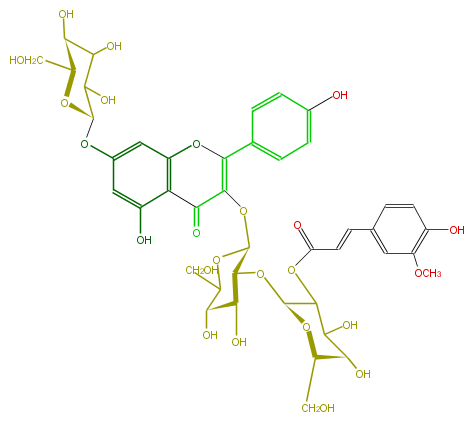
Mol:FL5FAAGL0095
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 67 73 0 0 0 0 0 0 0 0999 V2000 | + | 67 73 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.7577 1.0937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7577 1.0937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7577 0.4513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7577 0.4513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2014 0.1301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2014 0.1301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6451 0.4513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6451 0.4513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6451 1.0937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6451 1.0937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2014 1.4149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2014 1.4149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0888 0.1301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0888 0.1301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5325 0.4513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5325 0.4513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5325 1.0937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5325 1.0937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0888 1.4149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0888 1.4149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0888 -0.3707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0888 -0.3707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0236 1.4147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0236 1.4147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5905 1.0874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5905 1.0874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1575 1.4147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1575 1.4147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1575 2.0694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1575 2.0694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5905 2.3968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5905 2.3968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0236 2.0694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0236 2.0694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3138 1.4147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3138 1.4147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7243 2.3967 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7243 2.3967 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1384 0.0624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1384 0.0624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2014 -0.5120 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2014 -0.5120 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8802 -1.9352 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -0.8802 -1.9352 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -0.2981 -1.7850 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.2981 -1.7850 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.3259 -1.1903 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.3259 -1.1903 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.0352 -0.6771 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.0352 -0.6771 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.6737 -0.9024 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6737 -0.9024 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6173 -1.5221 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.6173 -1.5221 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.8802 -2.4081 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8802 -2.4081 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2981 -2.5414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2981 -2.5414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2075 -1.1830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2075 -1.1830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3074 -1.8040 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.3074 -1.8040 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.6686 -1.8040 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.6686 -1.8040 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.1278 -2.2482 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1278 -2.2482 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2847 -2.8712 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.2847 -2.8712 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.9236 -2.8712 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.9236 -2.8712 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.4644 -2.4270 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.4644 -2.4270 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.9229 -2.2132 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9229 -2.2132 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1827 -3.1799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1827 -3.1799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2202 -0.7118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2202 -0.7118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8171 -1.1149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8171 -1.1149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9342 -0.2164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9342 -0.2164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7453 -0.7118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7453 -0.7118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9723 -0.3186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9723 -0.3186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4260 -0.3186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4260 -0.3186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6988 -0.7911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6988 -0.7911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2444 -0.7911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2444 -0.7911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5172 -0.3186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5172 -0.3186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2444 0.1539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2444 0.1539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6988 0.1539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6988 0.1539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0617 -0.3186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0617 -0.3186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7212 2.2352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7212 2.2352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1533 1.9073 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.1533 1.9073 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.3335 2.5378 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.3335 2.5378 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.1533 3.1722 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.1533 3.1722 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.7212 3.5002 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.7212 3.5002 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.5411 2.8696 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.5411 2.8696 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -3.7658 4.0105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7658 4.0105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7989 3.3768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7989 3.3768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9159 2.2967 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9159 2.2967 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3576 -1.1649 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3576 -1.1649 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1794 -1.2369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1794 -1.2369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2250 -1.1093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2250 -1.1093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1252 -1.5447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1252 -1.5447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2173 3.0879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2173 3.0879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9592 2.4173 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9592 2.4173 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0954 -3.8624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0954 -3.8624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5120 -3.2791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5120 -3.2791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 3 21 1 0 0 0 0 | + | 3 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 35 38 1 0 0 0 0 | + | 35 38 1 0 0 0 0 |
| − | 32 30 1 0 0 0 0 | + | 32 30 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 31 1 0 0 0 0 | + | 40 31 1 0 0 0 0 |
| − | 39 41 2 0 0 0 0 | + | 39 41 2 0 0 0 0 |
| − | 39 42 1 0 0 0 0 | + | 39 42 1 0 0 0 0 |
| − | 42 43 2 0 0 0 0 | + | 42 43 2 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 2 0 0 0 0 | + | 48 49 2 0 0 0 0 |
| − | 49 44 1 0 0 0 0 | + | 49 44 1 0 0 0 0 |
| − | 47 50 1 0 0 0 0 | + | 47 50 1 0 0 0 0 |
| − | 52 51 1 1 0 0 0 | + | 52 51 1 1 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 55 56 1 1 0 0 0 | + | 55 56 1 1 0 0 0 |
| − | 56 51 1 1 0 0 0 | + | 56 51 1 1 0 0 0 |
| − | 55 57 1 0 0 0 0 | + | 55 57 1 0 0 0 0 |
| − | 54 58 1 0 0 0 0 | + | 54 58 1 0 0 0 0 |
| − | 53 59 1 0 0 0 0 | + | 53 59 1 0 0 0 0 |
| − | 18 52 1 0 0 0 0 | + | 18 52 1 0 0 0 0 |
| − | 46 60 1 0 0 0 0 | + | 46 60 1 0 0 0 0 |
| − | 60 61 1 0 0 0 0 | + | 60 61 1 0 0 0 0 |
| − | 27 62 1 0 0 0 0 | + | 27 62 1 0 0 0 0 |
| − | 62 63 1 0 0 0 0 | + | 62 63 1 0 0 0 0 |
| − | 56 64 1 0 0 0 0 | + | 56 64 1 0 0 0 0 |
| − | 64 65 1 0 0 0 0 | + | 64 65 1 0 0 0 0 |
| − | 34 66 1 0 0 0 0 | + | 34 66 1 0 0 0 0 |
| − | 66 67 1 0 0 0 0 | + | 66 67 1 0 0 0 0 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 66 67 | + | M SAL 4 2 66 67 |
| − | M SBL 4 1 72 | + | M SBL 4 1 72 |
| − | M SMT 4 CH2OH | + | M SMT 4 CH2OH |
| − | M SVB 4 72 1.0954 -3.8624 | + | M SVB 4 72 1.0954 -3.8624 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 64 65 | + | M SAL 3 2 64 65 |
| − | M SBL 3 1 70 | + | M SBL 3 1 70 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 70 -4.0617 3.3532 | + | M SVB 3 70 -4.0617 3.3532 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 62 63 | + | M SAL 2 2 62 63 |
| − | M SBL 2 1 68 | + | M SBL 2 1 68 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 68 -1.225 -1.1093 | + | M SVB 2 68 -1.225 -1.1093 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 60 61 | + | M SAL 1 2 60 61 |
| − | M SBL 1 1 66 | + | M SBL 1 1 66 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 66 3.1889 -1.1778 | + | M SVB 1 66 3.1889 -1.1778 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FAAGL0095 | + | ID FL5FAAGL0095 |
| − | KNApSAcK_ID C00005914 | + | KNApSAcK_ID C00005914 |
| − | NAME Kaempferol 3-[2'''-(E)-ferulylsophoroside]-7-glucoside | + | NAME Kaempferol 3-[2'''-(E)-ferulylsophoroside]-7-glucoside |
| − | CAS_RN 151649-63-7 | + | CAS_RN 151649-63-7 |
| − | FORMULA C43H48O24 | + | FORMULA C43H48O24 |
| − | EXACTMASS 948.253552464 | + | EXACTMASS 948.253552464 |
| − | AVERAGEMASS 948.82682 | + | AVERAGEMASS 948.82682 |
| − | SMILES [C@H](O)([C@H](CO)7)C(C([C@H](O7)Oc(c6)cc(O)c(c65)C(=O)C(=C(O5)c(c4)ccc(O)c4)O[C@H](O3)[C@H]([C@@H](O)[C@H](C3CO)O)O[C@@H](C1OC(C=Cc(c2)cc(OC)c(O)c2)=O)O[C@@H]([C@@H](C1O)O)CO)O)O | + | SMILES [C@H](O)([C@H](CO)7)C(C([C@H](O7)Oc(c6)cc(O)c(c65)C(=O)C(=C(O5)c(c4)ccc(O)c4)O[C@H](O3)[C@H]([C@@H](O)[C@H](C3CO)O)O[C@@H](C1OC(C=Cc(c2)cc(OC)c(O)c2)=O)O[C@@H]([C@@H](C1O)O)CO)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
67 73 0 0 0 0 0 0 0 0999 V2000
-2.7577 1.0937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7577 0.4513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2014 0.1301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6451 0.4513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6451 1.0937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2014 1.4149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0888 0.1301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5325 0.4513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5325 1.0937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0888 1.4149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0888 -0.3707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0236 1.4147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5905 1.0874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1575 1.4147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1575 2.0694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5905 2.3968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0236 2.0694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3138 1.4147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7243 2.3967 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1384 0.0624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2014 -0.5120 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8802 -1.9352 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-0.2981 -1.7850 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.3259 -1.1903 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.0352 -0.6771 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.6737 -0.9024 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6173 -1.5221 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.8802 -2.4081 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2981 -2.5414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2075 -1.1830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3074 -1.8040 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.6686 -1.8040 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.1278 -2.2482 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2847 -2.8712 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.9236 -2.8712 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.4644 -2.4270 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.9229 -2.2132 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1827 -3.1799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2202 -0.7118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8171 -1.1149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9342 -0.2164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7453 -0.7118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9723 -0.3186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4260 -0.3186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6988 -0.7911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2444 -0.7911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5172 -0.3186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2444 0.1539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6988 0.1539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0617 -0.3186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7212 2.2352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1533 1.9073 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.3335 2.5378 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.1533 3.1722 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.7212 3.5002 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.5411 2.8696 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-3.7658 4.0105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7989 3.3768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9159 2.2967 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3576 -1.1649 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1794 -1.2369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2250 -1.1093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1252 -1.5447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2173 3.0879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9592 2.4173 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0954 -3.8624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5120 -3.2791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
15 19 1 0 0 0 0
8 20 1 0 0 0 0
3 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 20 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
36 37 1 0 0 0 0
35 38 1 0 0 0 0
32 30 1 0 0 0 0
39 40 1 0 0 0 0
40 31 1 0 0 0 0
39 41 2 0 0 0 0
39 42 1 0 0 0 0
42 43 2 0 0 0 0
43 44 1 0 0 0 0
44 45 2 0 0 0 0
45 46 1 0 0 0 0
46 47 2 0 0 0 0
47 48 1 0 0 0 0
48 49 2 0 0 0 0
49 44 1 0 0 0 0
47 50 1 0 0 0 0
52 51 1 1 0 0 0
52 53 1 0 0 0 0
53 54 1 0 0 0 0
54 55 1 0 0 0 0
55 56 1 1 0 0 0
56 51 1 1 0 0 0
55 57 1 0 0 0 0
54 58 1 0 0 0 0
53 59 1 0 0 0 0
18 52 1 0 0 0 0
46 60 1 0 0 0 0
60 61 1 0 0 0 0
27 62 1 0 0 0 0
62 63 1 0 0 0 0
56 64 1 0 0 0 0
64 65 1 0 0 0 0
34 66 1 0 0 0 0
66 67 1 0 0 0 0
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 66 67
M SBL 4 1 72
M SMT 4 CH2OH
M SVB 4 72 1.0954 -3.8624
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 64 65
M SBL 3 1 70
M SMT 3 CH2OH
M SVB 3 70 -4.0617 3.3532
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 62 63
M SBL 2 1 68
M SMT 2 CH2OH
M SVB 2 68 -1.225 -1.1093
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 60 61
M SBL 1 1 66
M SMT 1 OCH3
M SVB 1 66 3.1889 -1.1778
S SKP 8
ID FL5FAAGL0095
KNApSAcK_ID C00005914
NAME Kaempferol 3-[2'''-(E)-ferulylsophoroside]-7-glucoside
CAS_RN 151649-63-7
FORMULA C43H48O24
EXACTMASS 948.253552464
AVERAGEMASS 948.82682
SMILES [C@H](O)([C@H](CO)7)C(C([C@H](O7)Oc(c6)cc(O)c(c65)C(=O)C(=C(O5)c(c4)ccc(O)c4)O[C@H](O3)[C@H]([C@@H](O)[C@H](C3CO)O)O[C@@H](C1OC(C=Cc(c2)cc(OC)c(O)c2)=O)O[C@@H]([C@@H](C1O)O)CO)O)O
M END