Mol:FL5FAAGL0100
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 64 70 0 0 0 0 0 0 0 0999 V2000 | + | 64 70 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.4460 0.2818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4460 0.2818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1744 0.6400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1744 0.6400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1744 1.3563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1744 1.3563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4460 1.7145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4460 1.7145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0664 1.3563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0664 1.3563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0664 0.6400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0664 0.6400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8178 0.2685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8178 0.2685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4613 0.6400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4613 0.6400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4613 1.3829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4613 1.3829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8178 1.7544 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8178 1.7544 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8178 -0.2807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8178 -0.2807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4460 -0.1979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4460 -0.1979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7715 1.3873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7715 1.3873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3794 1.7383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3794 1.7383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3794 2.4402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3794 2.4402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7715 2.7912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7715 2.7912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1636 2.4402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1636 2.4402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1636 1.7383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1636 1.7383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7831 2.6733 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7831 2.6733 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9593 0.3524 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9593 0.3524 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5119 1.6135 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5119 1.6135 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3270 1.5193 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -2.3270 1.5193 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.6908 2.1495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6908 2.1495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3948 1.9650 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.3948 1.9650 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -4.0945 2.1495 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -4.0945 2.1495 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.7307 1.5193 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.7307 1.5193 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.0267 1.7036 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.0267 1.7036 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -4.5265 2.3989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5265 2.3989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1364 1.2353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1364 1.2353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6935 1.1266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6935 1.1266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6935 0.4138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6935 0.4138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1993 0.1285 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1993 0.1285 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1319 0.1607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1319 0.1607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1319 -0.4774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1319 -0.4774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5759 -0.7337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5759 -0.7337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1823 -0.3836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1823 -0.3836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7888 -0.7337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7888 -0.7337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7888 -1.4340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7888 -1.4340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1823 -1.7841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1823 -1.7841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5759 -1.4340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5759 -1.4340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2069 -1.6754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2069 -1.6754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7220 -0.5102 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.7220 -0.5102 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.9962 -0.3157 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.9962 -0.3157 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.3826 -0.9602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3826 -0.9602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3826 -1.7116 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.3826 -1.7116 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.1085 -1.9062 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.1085 -1.9062 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.7220 -1.2617 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.7220 -1.2617 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.9912 -2.3437 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9912 -2.3437 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5829 0.0089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5829 0.0089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6460 0.7933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6460 0.7933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0324 0.4390 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.0324 0.4390 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.7198 0.2670 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 3.7198 0.2670 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 4.2235 -0.2313 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.2235 -0.2313 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.8372 0.1229 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.8372 0.1229 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.1498 0.2950 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 4.1498 0.2950 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 4.0922 0.9120 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0922 0.9120 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5006 0.5406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5006 0.5406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2383 0.2689 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2383 0.2689 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4731 -0.6636 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4731 -0.6636 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1352 -1.2617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1352 -1.2617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5795 2.7467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5795 2.7467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8517 3.4324 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8517 3.4324 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1523 -2.7912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1523 -2.7912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5690 -2.2079 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5690 -2.2079 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 3 1 0 0 0 0 | + | 10 3 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 13 2 0 0 0 0 | + | 18 13 2 0 0 0 0 |
| − | 9 18 1 0 0 0 0 | + | 9 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 22 27 1 1 0 0 0 | + | 22 27 1 1 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 25 28 1 0 0 0 0 | + | 25 28 1 0 0 0 0 |
| − | 26 29 1 0 0 0 0 | + | 26 29 1 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 2 0 0 0 0 | + | 31 32 2 0 0 0 0 |
| − | 31 33 1 0 0 0 0 | + | 31 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 35 2 0 0 0 0 | + | 40 35 2 0 0 0 0 |
| − | 38 41 1 0 0 0 0 | + | 38 41 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 45 44 1 1 0 0 0 | + | 45 44 1 1 0 0 0 |
| − | 46 45 1 1 0 0 0 | + | 46 45 1 1 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 42 1 0 0 0 0 | + | 47 42 1 0 0 0 0 |
| − | 20 43 1 0 0 0 0 | + | 20 43 1 0 0 0 0 |
| − | 46 48 1 0 0 0 0 | + | 46 48 1 0 0 0 0 |
| − | 42 49 1 0 0 0 0 | + | 42 49 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 1 1 0 0 0 | + | 51 52 1 1 0 0 0 |
| − | 53 52 1 1 0 0 0 | + | 53 52 1 1 0 0 0 |
| − | 54 53 1 1 0 0 0 | + | 54 53 1 1 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 55 50 1 0 0 0 0 | + | 55 50 1 0 0 0 0 |
| − | 49 51 1 0 0 0 0 | + | 49 51 1 0 0 0 0 |
| − | 52 56 1 0 0 0 0 | + | 52 56 1 0 0 0 0 |
| − | 55 57 1 0 0 0 0 | + | 55 57 1 0 0 0 0 |
| − | 54 58 1 0 0 0 0 | + | 54 58 1 0 0 0 0 |
| − | 53 59 1 0 0 0 0 | + | 53 59 1 0 0 0 0 |
| − | 47 60 1 0 0 0 0 | + | 47 60 1 0 0 0 0 |
| − | 24 61 1 0 0 0 0 | + | 24 61 1 0 0 0 0 |
| − | 61 62 1 0 0 0 0 | + | 61 62 1 0 0 0 0 |
| − | 45 63 1 0 0 0 0 | + | 45 63 1 0 0 0 0 |
| − | 63 64 1 0 0 0 0 | + | 63 64 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 63 64 | + | M SAL 2 2 63 64 |
| − | M SBL 2 1 69 | + | M SBL 2 1 69 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 69 2.1523 -2.7912 | + | M SVB 2 69 2.1523 -2.7912 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 61 62 | + | M SAL 1 2 61 62 |
| − | M SBL 1 1 67 | + | M SBL 1 1 67 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 67 -3.5795 2.7467 | + | M SVB 1 67 -3.5795 2.7467 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FAAGL0100 | + | ID FL5FAAGL0100 |
| − | KNApSAcK_ID C00005919 | + | KNApSAcK_ID C00005919 |
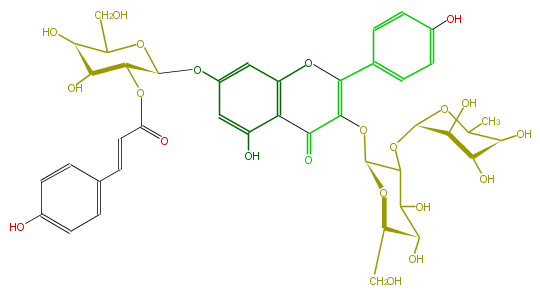
| − | NAME Kaempferol 3-neohesperidoside-7-(2''-p-coumarylglucoside) | + | NAME Kaempferol 3-neohesperidoside-7-(2''-p-coumarylglucoside) |
| − | CAS_RN 173268-23-0 | + | CAS_RN 173268-23-0 |
| − | FORMULA C42H46O22 | + | FORMULA C42H46O22 |
| − | EXACTMASS 902.248073156 | + | EXACTMASS 902.248073156 |
| − | AVERAGEMASS 902.80144 | + | AVERAGEMASS 902.80144 |
| − | SMILES [C@H](O)([C@@H](OC([C@H](OC(=C6c(c7)ccc(O)c7)C(=O)c(c3O6)c(cc(O[C@@H]([C@H]4OC(C=Cc(c5)ccc(O)c5)=O)OC(CO)[C@H](O)[C@@H]4O)c3)O)2)C([C@@H](O)[C@H](O2)CO)O)1)[C@H](O)[C@H](O)C(C)O1 | + | SMILES [C@H](O)([C@@H](OC([C@H](OC(=C6c(c7)ccc(O)c7)C(=O)c(c3O6)c(cc(O[C@@H]([C@H]4OC(C=Cc(c5)ccc(O)c5)=O)OC(CO)[C@H](O)[C@@H]4O)c3)O)2)C([C@@H](O)[C@H](O2)CO)O)1)[C@H](O)[C@H](O)C(C)O1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
64 70 0 0 0 0 0 0 0 0999 V2000
-0.4460 0.2818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1744 0.6400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1744 1.3563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4460 1.7145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0664 1.3563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0664 0.6400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8178 0.2685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4613 0.6400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4613 1.3829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8178 1.7544 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8178 -0.2807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4460 -0.1979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7715 1.3873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3794 1.7383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3794 2.4402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7715 2.7912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1636 2.4402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1636 1.7383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7831 2.6733 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9593 0.3524 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5119 1.6135 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3270 1.5193 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.6908 2.1495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3948 1.9650 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-4.0945 2.1495 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.7307 1.5193 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.0267 1.7036 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-4.5265 2.3989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1364 1.2353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6935 1.1266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6935 0.4138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1993 0.1285 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1319 0.1607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1319 -0.4774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5759 -0.7337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1823 -0.3836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7888 -0.7337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7888 -1.4340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1823 -1.7841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5759 -1.4340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2069 -1.6754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7220 -0.5102 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.9962 -0.3157 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.3826 -0.9602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3826 -1.7116 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.1085 -1.9062 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.7220 -1.2617 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.9912 -2.3437 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5829 0.0089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6460 0.7933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0324 0.4390 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.7198 0.2670 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
4.2235 -0.2313 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.8372 0.1229 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.1498 0.2950 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
4.0922 0.9120 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5006 0.5406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2383 0.2689 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4731 -0.6636 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1352 -1.2617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5795 2.7467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8517 3.4324 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1523 -2.7912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5690 -2.2079 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 3 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 18 1 0 0 0 0
18 13 2 0 0 0 0
9 18 1 0 0 0 0
15 19 1 0 0 0 0
8 20 1 0 0 0 0
5 21 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 1 0 0 0
26 27 1 1 0 0 0
22 27 1 1 0 0 0
21 22 1 0 0 0 0
25 28 1 0 0 0 0
26 29 1 0 0 0 0
27 30 1 0 0 0 0
30 31 1 0 0 0 0
31 32 2 0 0 0 0
31 33 1 0 0 0 0
33 34 2 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 40 1 0 0 0 0
40 35 2 0 0 0 0
38 41 1 0 0 0 0
42 43 1 0 0 0 0
43 44 1 1 0 0 0
45 44 1 1 0 0 0
46 45 1 1 0 0 0
46 47 1 0 0 0 0
47 42 1 0 0 0 0
20 43 1 0 0 0 0
46 48 1 0 0 0 0
42 49 1 0 0 0 0
50 51 1 0 0 0 0
51 52 1 1 0 0 0
53 52 1 1 0 0 0
54 53 1 1 0 0 0
54 55 1 0 0 0 0
55 50 1 0 0 0 0
49 51 1 0 0 0 0
52 56 1 0 0 0 0
55 57 1 0 0 0 0
54 58 1 0 0 0 0
53 59 1 0 0 0 0
47 60 1 0 0 0 0
24 61 1 0 0 0 0
61 62 1 0 0 0 0
45 63 1 0 0 0 0
63 64 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 63 64
M SBL 2 1 69
M SMT 2 CH2OH
M SVB 2 69 2.1523 -2.7912
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 61 62
M SBL 1 1 67
M SMT 1 CH2OH
M SVB 1 67 -3.5795 2.7467
S SKP 8
ID FL5FAAGL0100
KNApSAcK_ID C00005919
NAME Kaempferol 3-neohesperidoside-7-(2''-p-coumarylglucoside)
CAS_RN 173268-23-0
FORMULA C42H46O22
EXACTMASS 902.248073156
AVERAGEMASS 902.80144
SMILES [C@H](O)([C@@H](OC([C@H](OC(=C6c(c7)ccc(O)c7)C(=O)c(c3O6)c(cc(O[C@@H]([C@H]4OC(C=Cc(c5)ccc(O)c5)=O)OC(CO)[C@H](O)[C@@H]4O)c3)O)2)C([C@@H](O)[C@H](O2)CO)O)1)[C@H](O)[C@H](O)C(C)O1
M END