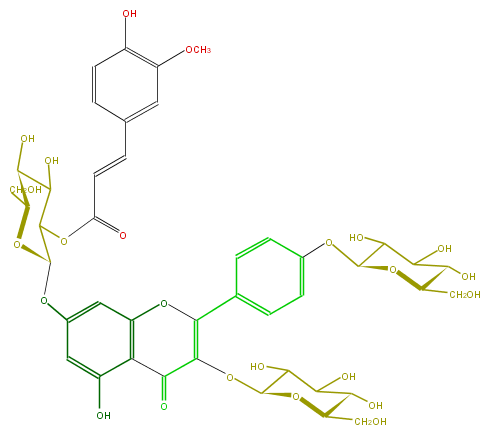
Mol:FL5FAAGL0104
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 67 73 0 0 0 0 0 0 0 0999 V2000 | + | 67 73 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.3435 -1.8646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3435 -1.8646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3435 -2.7070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3435 -2.7070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6140 -3.1281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6140 -3.1281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8844 -2.7070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8844 -2.7070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8844 -1.8646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8844 -1.8646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6140 -1.4434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6140 -1.4434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1549 -3.1281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1549 -3.1281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4254 -2.7070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4254 -2.7070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4254 -1.8646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4254 -1.8646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1549 -1.4434 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1549 -1.4434 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1549 -3.7850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1549 -3.7850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4932 -1.2813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4932 -1.2813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2368 -1.7105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2368 -1.7105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9802 -1.2813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9802 -1.2813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9802 -0.4227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9802 -0.4227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2368 0.0066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2368 0.0066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4932 -0.4227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4932 -0.4227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6140 -3.9703 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6140 -3.9703 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5776 -0.0543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5776 -0.0543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8849 -1.3820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8849 -1.3820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3442 -3.1193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3442 -3.1193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6613 -2.9911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6613 -2.9911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0569 -3.4983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0569 -3.4983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8439 -3.5539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8439 -3.5539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4869 -4.0188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4869 -4.0188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0914 -3.5116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0914 -3.5116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3044 -3.4558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3044 -3.4558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0496 -2.8742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0496 -2.8742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9411 -3.0974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9411 -3.0974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5579 -3.7497 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5579 -3.7497 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8523 -0.1160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8523 -0.1160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2479 -0.6232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2479 -0.6232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0348 -0.6788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0348 -0.6788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6778 -1.1437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6778 -1.1437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2823 -0.6365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2823 -0.6365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4953 -0.5808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4953 -0.5808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2837 0.0518 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2837 0.0518 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1320 -0.2017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1320 -0.2017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6664 -0.8334 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6664 -0.8334 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4778 -0.1076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4778 -0.1076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7331 -0.5376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7331 -0.5376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9694 0.2892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9694 0.2892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7331 1.1211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7331 1.1211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4778 1.5511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4778 1.5511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2415 0.7243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2415 0.7243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3380 2.2966 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3380 2.2966 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7952 1.8068 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7952 1.8068 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4218 -0.0270 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4218 -0.0270 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7250 0.4793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7250 0.4793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0794 0.1064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0794 0.1064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7250 1.4493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7250 1.4493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0232 1.8546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0232 1.8546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0232 2.6643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0232 2.6643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3087 3.0767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3087 3.0767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3087 3.9015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3087 3.9015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0232 4.3141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0232 4.3141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7375 3.9015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7375 3.9015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7375 3.0767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7375 3.0767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0232 5.1382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0232 5.1382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2348 -4.1214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2348 -4.1214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8648 -5.1382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8648 -5.1382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6937 1.1328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6937 1.1328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6664 0.2534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6664 0.2534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5951 4.3136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5951 4.3136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3419 3.7726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3419 3.7726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3859 -1.2477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3859 -1.2477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0158 -2.2645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0158 -2.2645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 23 21 1 0 0 0 0 | + | 23 21 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 32 19 1 0 0 0 0 | + | 32 19 1 0 0 0 0 |
| − | 41 40 1 1 0 0 0 | + | 41 40 1 1 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 45 40 1 1 0 0 0 | + | 45 40 1 1 0 0 0 |
| − | 44 46 1 0 0 0 0 | + | 44 46 1 0 0 0 0 |
| − | 43 47 1 0 0 0 0 | + | 43 47 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 41 20 1 0 0 0 0 | + | 41 20 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 49 51 1 0 0 0 0 | + | 49 51 1 0 0 0 0 |
| − | 51 52 2 0 0 0 0 | + | 51 52 2 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 53 54 2 0 0 0 0 | + | 53 54 2 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 55 56 2 0 0 0 0 | + | 55 56 2 0 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 57 58 2 0 0 0 0 | + | 57 58 2 0 0 0 0 |
| − | 58 53 1 0 0 0 0 | + | 58 53 1 0 0 0 0 |
| − | 56 59 1 0 0 0 0 | + | 56 59 1 0 0 0 0 |
| − | 60 61 1 0 0 0 0 | + | 60 61 1 0 0 0 0 |
| − | 25 60 1 0 0 0 0 | + | 25 60 1 0 0 0 0 |
| − | 62 63 1 0 0 0 0 | + | 62 63 1 0 0 0 0 |
| − | 45 62 1 0 0 0 0 | + | 45 62 1 0 0 0 0 |
| − | 64 65 1 0 0 0 0 | + | 64 65 1 0 0 0 0 |
| − | 55 64 1 0 0 0 0 | + | 55 64 1 0 0 0 0 |
| − | 66 67 1 0 0 0 0 | + | 66 67 1 0 0 0 0 |
| − | 34 66 1 0 0 0 0 | + | 34 66 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 60 61 | + | M SAL 1 2 60 61 |
| − | M SBL 1 1 67 | + | M SBL 1 1 67 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 67 -0.7480 0.1027 | + | M SBV 1 67 -0.7480 0.1027 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 62 63 | + | M SAL 2 2 62 63 |
| − | M SBL 2 1 69 | + | M SBL 2 1 69 |
| − | M SMT 2 ^ CH2OH | + | M SMT 2 ^ CH2OH |
| − | M SBV 2 69 0.4523 -0.4085 | + | M SBV 2 69 0.4523 -0.4085 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 64 65 | + | M SAL 3 2 64 65 |
| − | M SBL 3 1 71 | + | M SBL 3 1 71 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SBV 3 71 -0.7136 -0.4120 | + | M SBV 3 71 -0.7136 -0.4120 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 66 67 | + | M SAL 4 2 66 67 |
| − | M SBL 4 1 73 | + | M SBL 4 1 73 |
| − | M SMT 4 CH2OH | + | M SMT 4 CH2OH |
| − | M SBV 4 73 -0.7081 0.1040 | + | M SBV 4 73 -0.7081 0.1040 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGL0104 | + | ID FL5FAAGL0104 |
| − | FORMULA C43H48O24 | + | FORMULA C43H48O24 |
| − | EXACTMASS 948.253552464 | + | EXACTMASS 948.253552464 |
| − | AVERAGEMASS 948.82682 | + | AVERAGEMASS 948.82682 |
| − | SMILES C(OC(=C(c(c6)ccc(OC(O7)C(O)C(C(C(CO)7)O)O)c6)5)C(=O)c(c2O5)c(cc(OC(C(OC(C=Cc(c4)cc(c(O)c4)OC)=O)3)OC(C(O)C3O)CO)c2)O)(O1)C(O)C(O)C(C(CO)1)O | + | SMILES C(OC(=C(c(c6)ccc(OC(O7)C(O)C(C(C(CO)7)O)O)c6)5)C(=O)c(c2O5)c(cc(OC(C(OC(C=Cc(c4)cc(c(O)c4)OC)=O)3)OC(C(O)C3O)CO)c2)O)(O1)C(O)C(O)C(C(CO)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
67 73 0 0 0 0 0 0 0 0999 V2000
-3.3435 -1.8646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3435 -2.7070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6140 -3.1281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8844 -2.7070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8844 -1.8646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6140 -1.4434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1549 -3.1281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4254 -2.7070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4254 -1.8646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1549 -1.4434 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1549 -3.7850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4932 -1.2813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2368 -1.7105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9802 -1.2813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9802 -0.4227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2368 0.0066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4932 -0.4227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6140 -3.9703 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5776 -0.0543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8849 -1.3820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3442 -3.1193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6613 -2.9911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0569 -3.4983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8439 -3.5539 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4869 -4.0188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0914 -3.5116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3044 -3.4558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0496 -2.8742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9411 -3.0974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5579 -3.7497 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8523 -0.1160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2479 -0.6232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0348 -0.6788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6778 -1.1437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2823 -0.6365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4953 -0.5808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2837 0.0518 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1320 -0.2017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.6664 -0.8334 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4778 -0.1076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7331 -0.5376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9694 0.2892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7331 1.1211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4778 1.5511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2415 0.7243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3380 2.2966 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7952 1.8068 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4218 -0.0270 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7250 0.4793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0794 0.1064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7250 1.4493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0232 1.8546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0232 2.6643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3087 3.0767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3087 3.9015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0232 4.3141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7375 3.9015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7375 3.0767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0232 5.1382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2348 -4.1214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8648 -5.1382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6937 1.1328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.6664 0.2534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5951 4.3136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3419 3.7726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3859 -1.2477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0158 -2.2645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
1 20 1 0 0 0 0
21 8 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
23 21 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
36 38 1 0 0 0 0
35 39 1 0 0 0 0
32 19 1 0 0 0 0
41 40 1 1 0 0 0
41 42 1 0 0 0 0
42 43 1 0 0 0 0
43 44 1 0 0 0 0
44 45 1 1 0 0 0
45 40 1 1 0 0 0
44 46 1 0 0 0 0
43 47 1 0 0 0 0
42 48 1 0 0 0 0
41 20 1 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
49 51 1 0 0 0 0
51 52 2 0 0 0 0
52 53 1 0 0 0 0
53 54 2 0 0 0 0
54 55 1 0 0 0 0
55 56 2 0 0 0 0
56 57 1 0 0 0 0
57 58 2 0 0 0 0
58 53 1 0 0 0 0
56 59 1 0 0 0 0
60 61 1 0 0 0 0
25 60 1 0 0 0 0
62 63 1 0 0 0 0
45 62 1 0 0 0 0
64 65 1 0 0 0 0
55 64 1 0 0 0 0
66 67 1 0 0 0 0
34 66 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 60 61
M SBL 1 1 67
M SMT 1 CH2OH
M SBV 1 67 -0.7480 0.1027
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 62 63
M SBL 2 1 69
M SMT 2 ^ CH2OH
M SBV 2 69 0.4523 -0.4085
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 64 65
M SBL 3 1 71
M SMT 3 OCH3
M SBV 3 71 -0.7136 -0.4120
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 66 67
M SBL 4 1 73
M SMT 4 CH2OH
M SBV 4 73 -0.7081 0.1040
S SKP 5
ID FL5FAAGL0104
FORMULA C43H48O24
EXACTMASS 948.253552464
AVERAGEMASS 948.82682
SMILES C(OC(=C(c(c6)ccc(OC(O7)C(O)C(C(C(CO)7)O)O)c6)5)C(=O)c(c2O5)c(cc(OC(C(OC(C=Cc(c4)cc(c(O)c4)OC)=O)3)OC(C(O)C3O)CO)c2)O)(O1)C(O)C(O)C(C(CO)1)O
M END