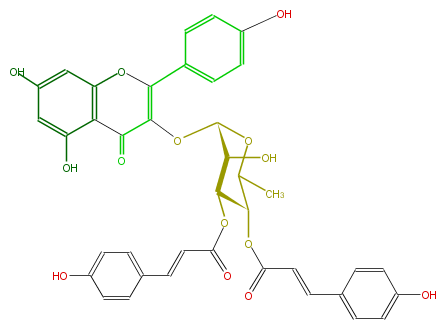
Mol:FL5FAAGS0056
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 53 58 0 0 0 0 0 0 0 0999 V2000 | + | 53 58 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.6204 1.5769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6204 1.5769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6204 0.9345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6204 0.9345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0641 0.6133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0641 0.6133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5078 0.9345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5078 0.9345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5078 1.5769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5078 1.5769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0641 1.8981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0641 1.8981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9515 0.6133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9515 0.6133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3952 0.9345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3952 0.9345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3952 1.5769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3952 1.5769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9515 1.8981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9515 1.8981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9515 0.1125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9515 0.1125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6948 2.0217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6948 2.0217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1278 1.6944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1278 1.6944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4392 2.0217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4392 2.0217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4392 2.6764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4392 2.6764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1278 3.0037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1278 3.0037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6948 2.6764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6948 2.6764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0641 -0.0288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0641 -0.0288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2329 3.0557 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2329 3.0557 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1154 1.8981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1154 1.8981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8319 0.5250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8319 0.5250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0473 -0.5275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0473 -0.5275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5809 -0.8902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5809 -0.8902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3815 -0.1927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3815 -0.1927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5809 0.5090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5809 0.5090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0473 0.8718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0473 0.8718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1519 0.1742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1519 0.1742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9223 0.1742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9223 0.1742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1140 -1.1297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1140 -1.1297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5809 -1.5395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5809 -1.5395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0018 -0.5508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0018 -0.5508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1513 -1.7025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1513 -1.7025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1683 -2.1778 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1683 -2.1778 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8826 -2.0621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8826 -2.0621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5770 -2.5914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5770 -2.5914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4931 -2.0621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4931 -2.0621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7855 -2.5686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7855 -2.5686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4282 -2.5686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4282 -2.5686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7094 -3.0557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7094 -3.0557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2718 -3.0557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2718 -3.0557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5530 -2.5686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5530 -2.5686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2718 -2.0815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2718 -2.0815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7094 -2.0815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7094 -2.0815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1154 -2.5686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1154 -2.5686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7288 -1.7025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7288 -1.7025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0105 -2.1904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0105 -2.1904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5545 -2.1904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5545 -2.1904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8141 -1.7408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8141 -1.7408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3332 -1.7408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3332 -1.7408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5927 -2.1904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5927 -2.1904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3332 -2.6399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3332 -2.6399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8141 -2.6399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8141 -2.6399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1111 -2.1904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1111 -2.1904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 1 1 0 0 0 0 | + | 20 1 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 22 1 1 0 0 0 | + | 27 22 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 29 32 1 0 0 0 0 | + | 29 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 30 34 1 0 0 0 0 | + | 30 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 2 0 0 0 0 | + | 42 43 2 0 0 0 0 |
| − | 43 38 1 0 0 0 0 | + | 43 38 1 0 0 0 0 |
| − | 41 44 1 0 0 0 0 | + | 41 44 1 0 0 0 0 |
| − | 32 45 1 0 0 0 0 | + | 32 45 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 2 0 0 0 0 | + | 51 52 2 0 0 0 0 |
| − | 52 47 1 0 0 0 0 | + | 52 47 1 0 0 0 0 |
| − | 50 53 1 0 0 0 0 | + | 50 53 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FAAGS0056 | + | ID FL5FAAGS0056 |
| − | KNApSAcK_ID C00005869 | + | KNApSAcK_ID C00005869 |
| − | NAME Kaempferol 3-(3'',4''-di-p-coumarylrhamnoside) | + | NAME Kaempferol 3-(3'',4''-di-p-coumarylrhamnoside) |
| − | CAS_RN 166321-99-9 | + | CAS_RN 166321-99-9 |
| − | FORMULA C39H32O14 | + | FORMULA C39H32O14 |
| − | EXACTMASS 724.179205732 | + | EXACTMASS 724.179205732 |
| − | AVERAGEMASS 724.6629800000001 | + | AVERAGEMASS 724.6629800000001 |
| − | SMILES Oc(c6)ccc(c6)C(O4)=C(C(c(c5O)c(cc(c5)O)4)=O)OC(O2)C(O)C(OC(=O)C=Cc(c3)ccc(c3)O)C(C(C)2)OC(C=Cc(c1)ccc(c1)O)=O | + | SMILES Oc(c6)ccc(c6)C(O4)=C(C(c(c5O)c(cc(c5)O)4)=O)OC(O2)C(O)C(OC(=O)C=Cc(c3)ccc(c3)O)C(C(C)2)OC(C=Cc(c1)ccc(c1)O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
53 58 0 0 0 0 0 0 0 0999 V2000
-3.6204 1.5769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6204 0.9345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0641 0.6133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5078 0.9345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5078 1.5769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0641 1.8981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9515 0.6133 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3952 0.9345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3952 1.5769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9515 1.8981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9515 0.1125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6948 2.0217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1278 1.6944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4392 2.0217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4392 2.6764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1278 3.0037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6948 2.6764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0641 -0.0288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2329 3.0557 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1154 1.8981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8319 0.5250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0473 -0.5275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5809 -0.8902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3815 -0.1927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5809 0.5090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0473 0.8718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1519 0.1742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9223 0.1742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1140 -1.1297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5809 -1.5395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0018 -0.5508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1513 -1.7025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1683 -2.1778 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8826 -2.0621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5770 -2.5914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4931 -2.0621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7855 -2.5686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4282 -2.5686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7094 -3.0557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2718 -3.0557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5530 -2.5686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2718 -2.0815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7094 -2.0815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1154 -2.5686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7288 -1.7025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0105 -2.1904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5545 -2.1904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8141 -1.7408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3332 -1.7408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5927 -2.1904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3332 -2.6399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8141 -2.6399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1111 -2.1904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 1 1 0 0 0 0
21 8 1 0 0 0 0
4 3 1 0 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 1 0 0 0
27 22 1 1 0 0 0
27 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
26 21 1 0 0 0 0
29 32 1 0 0 0 0
32 33 2 0 0 0 0
30 34 1 0 0 0 0
34 35 2 0 0 0 0
34 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 40 1 0 0 0 0
40 41 2 0 0 0 0
41 42 1 0 0 0 0
42 43 2 0 0 0 0
43 38 1 0 0 0 0
41 44 1 0 0 0 0
32 45 1 0 0 0 0
45 46 2 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 51 1 0 0 0 0
51 52 2 0 0 0 0
52 47 1 0 0 0 0
50 53 1 0 0 0 0
S SKP 8
ID FL5FAAGS0056
KNApSAcK_ID C00005869
NAME Kaempferol 3-(3'',4''-di-p-coumarylrhamnoside)
CAS_RN 166321-99-9
FORMULA C39H32O14
EXACTMASS 724.179205732
AVERAGEMASS 724.6629800000001
SMILES Oc(c6)ccc(c6)C(O4)=C(C(c(c5O)c(cc(c5)O)4)=O)OC(O2)C(O)C(OC(=O)C=Cc(c3)ccc(c3)O)C(C(C)2)OC(C=Cc(c1)ccc(c1)O)=O
M END