Mol:FL5FAAGS0061
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 53 58 0 0 0 0 0 0 0 0999 V2000 | + | 53 58 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.8927 2.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8927 2.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8927 1.4300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8927 1.4300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3364 1.1088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3364 1.1088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7801 1.4300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7801 1.4300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7801 2.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7801 2.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3364 2.3935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3364 2.3935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2238 1.1088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2238 1.1088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6674 1.4300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6674 1.4300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6674 2.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6674 2.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2238 2.3935 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2238 2.3935 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2238 0.6080 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2238 0.6080 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0330 2.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0330 2.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6000 2.1898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6000 2.1898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1670 2.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1670 2.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1670 3.1719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1670 3.1719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6000 3.4992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6000 3.4992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0330 3.1719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0330 3.1719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3364 0.4667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3364 0.4667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7738 3.5222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7738 3.5222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4083 2.5261 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4083 2.5261 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0380 1.0666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0380 1.0666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1033 -0.9495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1033 -0.9495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4321 -1.2586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4321 -1.2586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2622 -0.6642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2622 -0.6642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4321 -0.0662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4321 -0.0662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1033 0.2430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1033 0.2430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0665 -0.3515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0665 -0.3515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7231 -0.3515 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7231 -0.3515 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0342 -1.4628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0342 -1.4628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3029 -1.7407 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3029 -1.7407 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7908 -0.9694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7908 -0.9694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7311 -0.0425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7311 -0.0425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4303 -0.5635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4303 -0.5635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0087 -0.3982 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0087 -0.3982 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5907 -0.5635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5907 -0.5635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8916 -0.0425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8916 -0.0425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3131 -0.2078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3131 -0.2078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1724 -0.1922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1724 -0.1922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5513 0.2048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5513 0.2048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3257 -0.2931 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3257 -0.2931 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1423 -0.5635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1423 -0.5635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4083 -1.4751 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4083 -1.4751 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9942 -2.4647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9942 -2.4647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2532 -2.9133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2532 -2.9133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4385 -2.4647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4385 -2.4647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1337 -2.9927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1337 -2.9927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3246 -2.9927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3246 -2.9927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0189 -2.4631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0189 -2.4631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4074 -2.4631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4074 -2.4631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1017 -2.9927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1017 -2.9927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4074 -3.5222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4074 -3.5222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0189 -3.5222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0189 -3.5222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4303 -2.9927 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4303 -2.9927 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 22 1 1 0 0 0 | + | 27 22 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 37 39 1 0 0 0 0 | + | 37 39 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 33 28 1 0 0 0 0 | + | 33 28 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 2 0 0 0 0 | + | 51 52 2 0 0 0 0 |
| − | 52 47 1 0 0 0 0 | + | 52 47 1 0 0 0 0 |
| − | 50 53 1 0 0 0 0 | + | 50 53 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FAAGS0061 | + | ID FL5FAAGS0061 |
| − | KNApSAcK_ID C00005884 | + | KNApSAcK_ID C00005884 |
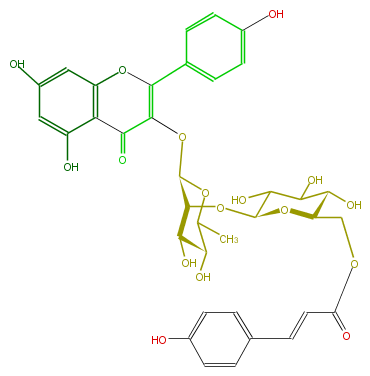
| − | NAME Kaempferol 3-[6'''-p-coumarylglucosyl-(1->2)-rhamnoside] | + | NAME Kaempferol 3-[6'''-p-coumarylglucosyl-(1->2)-rhamnoside] |
| − | CAS_RN 107190-70-5,111957-48-3 | + | CAS_RN 107190-70-5,111957-48-3 |
| − | FORMULA C36H36O17 | + | FORMULA C36H36O17 |
| − | EXACTMASS 740.1952497259999 | + | EXACTMASS 740.1952497259999 |
| − | AVERAGEMASS 740.66084 | + | AVERAGEMASS 740.66084 |
| − | SMILES O(C(O4)C(OC(O5)C(C(C(C5COC(C=Cc(c6)ccc(c6)O)=O)O)O)O)C(O)C(O)C4C)C(C2=O)=C(Oc(c3)c2c(cc3O)O)c(c1)ccc(O)c1 | + | SMILES O(C(O4)C(OC(O5)C(C(C(C5COC(C=Cc(c6)ccc(c6)O)=O)O)O)O)C(O)C(O)C4C)C(C2=O)=C(Oc(c3)c2c(cc3O)O)c(c1)ccc(O)c1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
53 58 0 0 0 0 0 0 0 0999 V2000
-2.8927 2.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8927 1.4300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3364 1.1088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7801 1.4300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7801 2.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3364 2.3935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2238 1.1088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6674 1.4300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6674 2.0724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2238 2.3935 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2238 0.6080 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0330 2.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6000 2.1898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1670 2.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1670 3.1719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6000 3.4992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0330 3.1719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3364 0.4667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7738 3.5222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4083 2.5261 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0380 1.0666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1033 -0.9495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4321 -1.2586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2622 -0.6642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4321 -0.0662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1033 0.2430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0665 -0.3515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7231 -0.3515 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0342 -1.4628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3029 -1.7407 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7908 -0.9694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7311 -0.0425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4303 -0.5635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0087 -0.3982 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5907 -0.5635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8916 -0.0425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3131 -0.2078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1724 -0.1922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5513 0.2048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3257 -0.2931 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1423 -0.5635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4083 -1.4751 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9942 -2.4647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2532 -2.9133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4385 -2.4647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1337 -2.9927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3246 -2.9927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0189 -2.4631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4074 -2.4631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1017 -2.9927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4074 -3.5222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0189 -3.5222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4303 -2.9927 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
1 20 1 0 0 0 0
21 8 1 0 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 1 0 0 0
27 22 1 1 0 0 0
27 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
26 21 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
32 38 1 0 0 0 0
37 39 1 0 0 0 0
36 40 1 0 0 0 0
35 41 1 0 0 0 0
33 28 1 0 0 0 0
41 42 1 0 0 0 0
42 43 1 0 0 0 0
43 44 2 0 0 0 0
43 45 1 0 0 0 0
45 46 2 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 51 1 0 0 0 0
51 52 2 0 0 0 0
52 47 1 0 0 0 0
50 53 1 0 0 0 0
S SKP 8
ID FL5FAAGS0061
KNApSAcK_ID C00005884
NAME Kaempferol 3-[6'''-p-coumarylglucosyl-(1->2)-rhamnoside]
CAS_RN 107190-70-5,111957-48-3
FORMULA C36H36O17
EXACTMASS 740.1952497259999
AVERAGEMASS 740.66084
SMILES O(C(O4)C(OC(O5)C(C(C(C5COC(C=Cc(c6)ccc(c6)O)=O)O)O)O)C(O)C(O)C4C)C(C2=O)=C(Oc(c3)c2c(cc3O)O)c(c1)ccc(O)c1
M END