Mol:FL5FABGI0008
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 49 53 0 0 0 0 0 0 0 0999 V2000 | + | 49 53 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.1408 -0.3209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1408 -0.3209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1408 -0.9633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1408 -0.9633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6971 -1.2845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6971 -1.2845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2534 -0.9633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2534 -0.9633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2534 -0.3209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2534 -0.3209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6971 0.0002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6971 0.0002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8097 -1.2845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8097 -1.2845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3660 -0.9633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3660 -0.9633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3660 -0.3209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3660 -0.3209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8097 0.0002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8097 0.0002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8097 -1.7853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8097 -1.7853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9221 0.0001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9221 0.0001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4891 -0.3272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4891 -0.3272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0561 0.0001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0561 0.0001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0561 0.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0561 0.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4891 0.9821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4891 0.9821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9221 0.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9221 0.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4153 0.0001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4153 0.0001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7602 -1.3522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7602 -1.3522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6971 -1.9266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6971 -1.9266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6971 0.6424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6971 0.6424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1410 0.9634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1410 0.9634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1410 1.6055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1410 1.6055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6971 1.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6971 1.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4151 1.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4151 1.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4723 -0.4064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4723 -0.4064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1011 -0.8964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1011 -0.8964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5666 -0.6885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5666 -0.6885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0508 -0.6830 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0508 -0.6830 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4256 -0.3081 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4256 -0.3081 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8932 -0.5549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8932 -0.5549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9861 -0.7031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9861 -0.7031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6797 -0.9246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6797 -0.9246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2603 -1.2026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2603 -1.2026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8235 -1.0871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8235 -1.0871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4523 -1.5770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4523 -1.5770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9178 -1.3692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9178 -1.3692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4021 -1.3636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4021 -1.3636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7769 -0.9887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7769 -0.9887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2445 -1.2355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2445 -1.2355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3374 -1.3837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3374 -1.3837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0310 -1.6052 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0310 -1.6052 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6116 -1.8833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6116 -1.8833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2917 0.2293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2917 0.2293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4948 0.0158 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4948 0.0158 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6429 -0.4513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6429 -0.4513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8460 -0.6649 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8460 -0.6649 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6229 0.9821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6229 0.9821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3374 0.5696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3374 0.5696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 6 21 1 0 0 0 0 | + | 6 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 2 0 0 0 0 | + | 22 23 2 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 23 25 1 0 0 0 0 | + | 23 25 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 28 1 1 0 0 0 | + | 27 28 1 1 0 0 0 |
| − | 29 28 1 1 0 0 0 | + | 29 28 1 1 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 26 1 0 0 0 0 | + | 31 26 1 0 0 0 0 |
| − | 26 32 1 0 0 0 0 | + | 26 32 1 0 0 0 0 |
| − | 27 33 1 0 0 0 0 | + | 27 33 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 29 18 1 0 0 0 0 | + | 29 18 1 0 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 38 37 1 1 0 0 0 | + | 38 37 1 1 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 35 1 0 0 0 0 | + | 40 35 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | 37 43 1 0 0 0 0 | + | 37 43 1 0 0 0 0 |
| − | 38 32 1 0 0 0 0 | + | 38 32 1 0 0 0 0 |
| − | 31 44 1 0 0 0 0 | + | 31 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 40 46 1 0 0 0 0 | + | 40 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 15 48 1 0 0 0 0 | + | 15 48 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 44 45 | + | M SAL 1 2 44 45 |
| − | M SBL 1 1 48 | + | M SBL 1 1 48 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 48 -7.4310 4.1345 | + | M SBV 1 48 -7.4310 4.1345 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 46 47 | + | M SAL 2 2 46 47 |
| − | M SBL 2 1 50 | + | M SBL 2 1 50 |
| − | M SMT 2 ^CH2OH | + | M SMT 2 ^CH2OH |
| − | M SBV 2 50 -7.4310 4.1345 | + | M SBV 2 50 -7.4310 4.1345 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 48 49 | + | M SAL 3 2 48 49 |
| − | M SBL 3 1 52 | + | M SBL 3 1 52 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SBV 3 52 -6.4657 3.6776 | + | M SBV 3 52 -6.4657 3.6776 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FABGI0008 | + | ID FL5FABGI0008 |
| − | KNApSAcK_ID C00005825 | + | KNApSAcK_ID C00005825 |
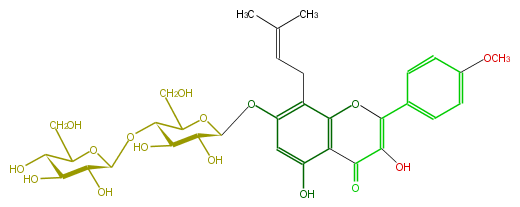
| − | NAME Cuhuoside;8-Prenylkaempferol 4'-methyl ether 7-cellobioside;7-[(4-O-beta-D-Glucopyranosyl-beta-D-glucopyranosyl)oxy]-3,5-dihydroxy-2-(4-methoxyphenyl)-8-(3-methyl-2-butenyl)-4H-1-benzopyran-4-one | + | NAME Cuhuoside;8-Prenylkaempferol 4'-methyl ether 7-cellobioside;7-[(4-O-beta-D-Glucopyranosyl-beta-D-glucopyranosyl)oxy]-3,5-dihydroxy-2-(4-methoxyphenyl)-8-(3-methyl-2-butenyl)-4H-1-benzopyran-4-one |
| − | CAS_RN 150903-46-1 | + | CAS_RN 150903-46-1 |
| − | FORMULA C33H40O16 | + | FORMULA C33H40O16 |
| − | EXACTMASS 692.2316352319999 | + | EXACTMASS 692.2316352319999 |
| − | AVERAGEMASS 692.6611 | + | AVERAGEMASS 692.6611 |
| − | SMILES c(OC)(c5)ccc(c5)C(=C(O)1)Oc(c2CC=C(C)C)c(c(O)cc2OC(C3O)OC(C(OC(O4)C(C(C(C4CO)O)O)O)C(O)3)CO)C1=O | + | SMILES c(OC)(c5)ccc(c5)C(=C(O)1)Oc(c2CC=C(C)C)c(c(O)cc2OC(C3O)OC(C(OC(O4)C(C(C(C4CO)O)O)O)C(O)3)CO)C1=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
49 53 0 0 0 0 0 0 0 0999 V2000
0.1408 -0.3209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1408 -0.9633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6971 -1.2845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2534 -0.9633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2534 -0.3209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6971 0.0002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8097 -1.2845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3660 -0.9633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3660 -0.3209 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8097 0.0002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8097 -1.7853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9221 0.0001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4891 -0.3272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0561 0.0001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0561 0.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4891 0.9821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9221 0.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4153 0.0001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7602 -1.3522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6971 -1.9266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6971 0.6424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1410 0.9634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1410 1.6055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6971 1.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4151 1.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4723 -0.4064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1011 -0.8964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5666 -0.6885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0508 -0.6830 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4256 -0.3081 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8932 -0.5549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9861 -0.7031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6797 -0.9246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2603 -1.2026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8235 -1.0871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4523 -1.5770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9178 -1.3692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4021 -1.3636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7769 -0.9887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2445 -1.2355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.3374 -1.3837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0310 -1.6052 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6116 -1.8833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2917 0.2293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4948 0.0158 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6429 -0.4513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8460 -0.6649 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6229 0.9821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.3374 0.5696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 0 0 0 0
3 20 1 0 0 0 0
6 21 1 0 0 0 0
21 22 1 0 0 0 0
22 23 2 0 0 0 0
23 24 1 0 0 0 0
23 25 1 0 0 0 0
26 27 1 1 0 0 0
27 28 1 1 0 0 0
29 28 1 1 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
31 26 1 0 0 0 0
26 32 1 0 0 0 0
27 33 1 0 0 0 0
28 34 1 0 0 0 0
29 18 1 0 0 0 0
35 36 1 1 0 0 0
36 37 1 1 0 0 0
38 37 1 1 0 0 0
38 39 1 0 0 0 0
39 40 1 0 0 0 0
40 35 1 0 0 0 0
35 41 1 0 0 0 0
36 42 1 0 0 0 0
37 43 1 0 0 0 0
38 32 1 0 0 0 0
31 44 1 0 0 0 0
44 45 1 0 0 0 0
40 46 1 0 0 0 0
46 47 1 0 0 0 0
15 48 1 0 0 0 0
48 49 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 44 45
M SBL 1 1 48
M SMT 1 ^CH2OH
M SBV 1 48 -7.4310 4.1345
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 46 47
M SBL 2 1 50
M SMT 2 ^CH2OH
M SBV 2 50 -7.4310 4.1345
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 48 49
M SBL 3 1 52
M SMT 3 OCH3
M SBV 3 52 -6.4657 3.6776
S SKP 8
ID FL5FABGI0008
KNApSAcK_ID C00005825
NAME Cuhuoside;8-Prenylkaempferol 4'-methyl ether 7-cellobioside;7-[(4-O-beta-D-Glucopyranosyl-beta-D-glucopyranosyl)oxy]-3,5-dihydroxy-2-(4-methoxyphenyl)-8-(3-methyl-2-butenyl)-4H-1-benzopyran-4-one
CAS_RN 150903-46-1
FORMULA C33H40O16
EXACTMASS 692.2316352319999
AVERAGEMASS 692.6611
SMILES c(OC)(c5)ccc(c5)C(=C(O)1)Oc(c2CC=C(C)C)c(c(O)cc2OC(C3O)OC(C(OC(O4)C(C(C(C4CO)O)O)O)C(O)3)CO)C1=O
M END