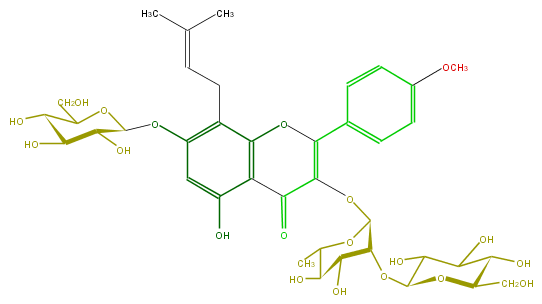
Mol:FL5FABGI0011
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 59 64 0 0 0 0 0 0 0 0999 V2000 | + | 59 64 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.9022 0.6752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9022 0.6752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9022 -0.1772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9022 -0.1772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1641 -0.6034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1641 -0.6034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4260 -0.1772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4260 -0.1772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4260 0.6752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4260 0.6752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1641 1.1012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1641 1.1012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3121 -0.6034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3121 -0.6034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0503 -0.1772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0503 -0.1772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0503 0.6752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0503 0.6752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3121 1.1012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3121 1.1012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3245 -1.4584 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3245 -1.4584 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7881 1.1011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7881 1.1011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5404 0.6668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5404 0.6668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2927 1.1011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2927 1.1011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2927 1.9698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2927 1.9698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5404 2.4042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5404 2.4042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7881 1.9698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7881 1.9698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6400 1.1011 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6400 1.1011 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8442 -0.6341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8442 -0.6341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1641 -1.4553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1641 -1.4553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1641 1.9533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1641 1.9533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9019 2.3792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9019 2.3792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9019 3.2313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9019 3.2313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1641 3.6572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1641 3.6572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6398 3.6572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6398 3.6572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2050 1.2883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2050 1.2883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7125 0.6382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7125 0.6382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0033 0.9140 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0033 0.9140 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3191 0.9213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3191 0.9213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8164 1.4188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8164 1.4188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4368 1.0913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4368 1.0913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7610 1.1699 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7610 1.1699 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4237 0.6230 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4237 0.6230 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4513 0.4987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4513 0.4987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5639 -1.9771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5639 -1.9771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1648 -2.6684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1648 -2.6684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9324 -2.4490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9324 -2.4490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7045 -2.6684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7045 -2.6684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1037 -1.9771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1037 -1.9771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3362 -2.1964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3362 -2.1964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9890 -2.0740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9890 -2.0740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9096 -1.5695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9096 -1.5695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7610 -2.1064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7610 -2.1064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1728 -1.7938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1728 -1.7938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1728 -2.4727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1728 -2.4727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6448 -1.9847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6448 -1.9847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3069 -1.8179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3069 -1.8179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3071 -1.1389 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3071 -1.1389 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8348 -1.6269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8348 -1.6269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5310 -2.7587 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5310 -2.7587 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6930 -2.4869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6930 -2.4869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7025 -2.1034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7025 -2.1034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6180 -2.4133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6180 -2.4133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4088 -2.5624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4088 -2.5624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4088 -3.6572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4088 -3.6572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0448 2.4040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0448 2.4040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9928 1.8567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9928 1.8567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9418 1.6033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9418 1.6033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3162 2.6319 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3162 2.6319 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 6 21 1 0 0 0 0 | + | 6 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 2 0 0 0 0 | + | 22 23 2 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 23 25 1 0 0 0 0 | + | 23 25 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 28 1 1 0 0 0 | + | 27 28 1 1 0 0 0 |
| − | 29 28 1 1 0 0 0 | + | 29 28 1 1 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 26 1 0 0 0 0 | + | 31 26 1 0 0 0 0 |
| − | 26 32 1 0 0 0 0 | + | 26 32 1 0 0 0 0 |
| − | 27 33 1 0 0 0 0 | + | 27 33 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 29 18 1 0 0 0 0 | + | 29 18 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 37 38 1 1 0 0 0 | + | 37 38 1 1 0 0 0 |
| − | 39 38 1 1 0 0 0 | + | 39 38 1 1 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 35 1 0 0 0 0 | + | 40 35 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 40 42 1 0 0 0 0 | + | 40 42 1 0 0 0 0 |
| − | 39 43 1 0 0 0 0 | + | 39 43 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 1 0 0 0 | + | 45 46 1 1 0 0 0 |
| − | 46 47 1 1 0 0 0 | + | 46 47 1 1 0 0 0 |
| − | 48 47 1 1 0 0 0 | + | 48 47 1 1 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 44 1 0 0 0 0 | + | 49 44 1 0 0 0 0 |
| − | 46 50 1 0 0 0 0 | + | 46 50 1 0 0 0 0 |
| − | 45 51 1 0 0 0 0 | + | 45 51 1 0 0 0 0 |
| − | 44 52 1 0 0 0 0 | + | 44 52 1 0 0 0 0 |
| − | 47 53 1 0 0 0 0 | + | 47 53 1 0 0 0 0 |
| − | 48 19 1 0 0 0 0 | + | 48 19 1 0 0 0 0 |
| − | 36 53 1 0 0 0 0 | + | 36 53 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 38 54 1 0 0 0 0 | + | 38 54 1 0 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 15 56 1 0 0 0 0 | + | 15 56 1 0 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 31 58 1 0 0 0 0 | + | 31 58 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 54 55 | + | M SAL 1 2 54 55 |
| − | M SBL 1 1 60 | + | M SBL 1 1 60 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 60 -0.7043 -0.1059 | + | M SBV 1 60 -0.7043 -0.1059 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 56 57 | + | M SAL 2 2 56 57 |
| − | M SBL 2 1 62 | + | M SBL 2 1 62 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 62 -0.7520 -0.4343 | + | M SBV 2 62 -0.7520 -0.4343 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 58 59 | + | M SAL 3 2 58 59 |
| − | M SBL 3 1 64 | + | M SBL 3 1 64 |
| − | M SMT 3 ^ CH2OH | + | M SMT 3 ^ CH2OH |
| − | M SBV 3 64 0.5049 -0.5120 | + | M SBV 3 64 0.5049 -0.5120 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FABGI0011 | + | ID FL5FABGI0011 |
| − | FORMULA C39H50O20 | + | FORMULA C39H50O20 |
| − | EXACTMASS 838.28954404 | + | EXACTMASS 838.28954404 |
| − | AVERAGEMASS 838.8023000000001 | + | AVERAGEMASS 838.8023000000001 |
| − | SMILES O(C(O5)C(OC(C6O)OC(C(O)C(O)6)CO)C(O)C(C5C)O)C(C(=O)2)=C(Oc(c3CC=C(C)C)c2c(O)cc3OC(O4)C(C(O)C(O)C4CO)O)c(c1)ccc(c1)OC | + | SMILES O(C(O5)C(OC(C6O)OC(C(O)C(O)6)CO)C(O)C(C5C)O)C(C(=O)2)=C(Oc(c3CC=C(C)C)c2c(O)cc3OC(O4)C(C(O)C(O)C4CO)O)c(c1)ccc(c1)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
59 64 0 0 0 0 0 0 0 0999 V2000
-1.9022 0.6752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9022 -0.1772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1641 -0.6034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4260 -0.1772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4260 0.6752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1641 1.1012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3121 -0.6034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0503 -0.1772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0503 0.6752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3121 1.1012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3245 -1.4584 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7881 1.1011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5404 0.6668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2927 1.1011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2927 1.9698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5404 2.4042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7881 1.9698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6400 1.1011 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8442 -0.6341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1641 -1.4553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1641 1.9533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9019 2.3792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9019 3.2313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1641 3.6572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6398 3.6572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2050 1.2883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7125 0.6382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0033 0.9140 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3191 0.9213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8164 1.4188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4368 1.0913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7610 1.1699 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.4237 0.6230 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4513 0.4987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5639 -1.9771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1648 -2.6684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9324 -2.4490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7045 -2.6684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1037 -1.9771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3362 -2.1964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9890 -2.0740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9096 -1.5695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.7610 -2.1064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1728 -1.7938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1728 -2.4727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6448 -1.9847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3069 -1.8179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3071 -1.1389 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8348 -1.6269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5310 -2.7587 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6930 -2.4869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7025 -2.1034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6180 -2.4133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.4088 -2.5624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4088 -3.6572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0448 2.4040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9928 1.8567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9418 1.6033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.3162 2.6319 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 0 0 0 0
3 20 1 0 0 0 0
6 21 1 0 0 0 0
21 22 1 0 0 0 0
22 23 2 0 0 0 0
23 24 1 0 0 0 0
23 25 1 0 0 0 0
26 27 1 1 0 0 0
27 28 1 1 0 0 0
29 28 1 1 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
31 26 1 0 0 0 0
26 32 1 0 0 0 0
27 33 1 0 0 0 0
28 34 1 0 0 0 0
29 18 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 1 0 0 0
37 38 1 1 0 0 0
39 38 1 1 0 0 0
39 40 1 0 0 0 0
40 35 1 0 0 0 0
35 41 1 0 0 0 0
40 42 1 0 0 0 0
39 43 1 0 0 0 0
44 45 1 0 0 0 0
45 46 1 1 0 0 0
46 47 1 1 0 0 0
48 47 1 1 0 0 0
48 49 1 0 0 0 0
49 44 1 0 0 0 0
46 50 1 0 0 0 0
45 51 1 0 0 0 0
44 52 1 0 0 0 0
47 53 1 0 0 0 0
48 19 1 0 0 0 0
36 53 1 0 0 0 0
54 55 1 0 0 0 0
38 54 1 0 0 0 0
56 57 1 0 0 0 0
15 56 1 0 0 0 0
58 59 1 0 0 0 0
31 58 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 54 55
M SBL 1 1 60
M SMT 1 CH2OH
M SBV 1 60 -0.7043 -0.1059
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 56 57
M SBL 2 1 62
M SMT 2 OCH3
M SBV 2 62 -0.7520 -0.4343
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 58 59
M SBL 3 1 64
M SMT 3 ^ CH2OH
M SBV 3 64 0.5049 -0.5120
S SKP 5
ID FL5FABGI0011
FORMULA C39H50O20
EXACTMASS 838.28954404
AVERAGEMASS 838.8023000000001
SMILES O(C(O5)C(OC(C6O)OC(C(O)C(O)6)CO)C(O)C(C5C)O)C(C(=O)2)=C(Oc(c3CC=C(C)C)c2c(O)cc3OC(O4)C(C(O)C(O)C4CO)O)c(c1)ccc(c1)OC
M END