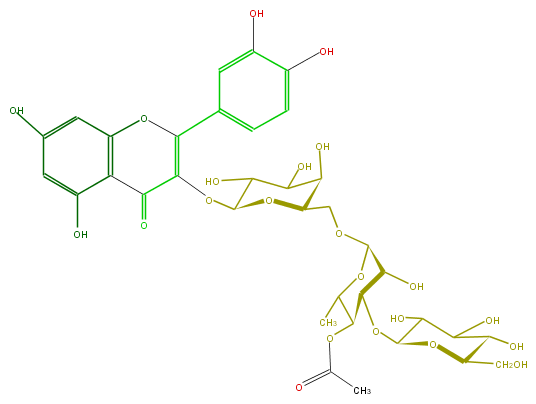
Mol:FL5FACGA0041
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 57 62 0 0 0 0 0 0 0 0999 V2000 | + | 57 62 0 0 0 0 0 0 0 0999 V2000 |
| − | -4.9877 1.7090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9877 1.7090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9877 0.8330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9877 0.8330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2289 0.3949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2289 0.3949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4702 0.8330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4702 0.8330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4702 1.7090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4702 1.7090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2289 2.1470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2289 2.1470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7114 0.3949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7114 0.3949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9527 0.8330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9527 0.8330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9527 1.7090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9527 1.7090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7114 2.1470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7114 2.1470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7114 -0.2882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7114 -0.2882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9975 2.3156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9975 2.3156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2242 1.8693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2242 1.8693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5491 2.3156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5491 2.3156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5491 3.2085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5491 3.2085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2242 3.6551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2242 3.6551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9975 3.2085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9975 3.2085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2289 -0.4810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2289 -0.4810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3767 3.6864 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3767 3.6864 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6908 2.3280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6908 2.3280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2242 0.2921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2242 0.2921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2453 0.7653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2453 0.7653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6555 0.0547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6555 0.0547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1334 0.2804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1334 0.2804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9270 0.0547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9270 0.0547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3374 0.7653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3374 0.7653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5485 0.5401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5485 0.5401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0715 0.7131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0715 0.7131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8695 1.0543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8695 1.0543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2732 1.5167 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2732 1.5167 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5056 0.1190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5056 0.1190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7404 -0.4708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7404 -0.4708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7184 -1.9312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7184 -1.9312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0675 -2.5356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0675 -2.5356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2369 -1.8587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2369 -1.8587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7406 -1.3698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7406 -1.3698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3916 -0.7654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3916 -0.7654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2221 -1.4424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2221 -1.4424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5670 -2.6902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5670 -2.6902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5194 -2.8564 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5194 -2.8564 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3581 -2.5030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3581 -2.5030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4194 -1.6750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4194 -1.6750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5300 -3.6334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5300 -3.6334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8132 -3.9746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8132 -3.9746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1290 -4.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1290 -4.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6320 -2.4283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6320 -2.4283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0517 -3.0085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0517 -3.0085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8722 -2.9949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8722 -2.9949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5804 -3.4180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5804 -3.4180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1608 -2.8380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1608 -2.8380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3402 -2.8516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3402 -2.8516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1303 -2.4008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1303 -2.4008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1515 -2.4373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1515 -2.4373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6908 -3.0395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6908 -3.0395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2242 4.5477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2242 4.5477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3719 -3.4608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3719 -3.4608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0808 -4.5477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0808 -4.5477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 23 21 1 0 0 0 0 | + | 23 21 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 40 43 1 0 0 0 0 | + | 40 43 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 1 1 0 0 0 | + | 47 48 1 1 0 0 0 |
| − | 48 49 1 1 0 0 0 | + | 48 49 1 1 0 0 0 |
| − | 50 49 1 1 0 0 0 | + | 50 49 1 1 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 46 1 0 0 0 0 | + | 51 46 1 0 0 0 0 |
| − | 46 52 1 0 0 0 0 | + | 46 52 1 0 0 0 0 |
| − | 51 53 1 0 0 0 0 | + | 51 53 1 0 0 0 0 |
| − | 50 54 1 0 0 0 0 | + | 50 54 1 0 0 0 0 |
| − | 47 39 1 0 0 0 0 | + | 47 39 1 0 0 0 0 |
| − | 16 55 1 0 0 0 0 | + | 16 55 1 0 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 49 56 1 0 0 0 0 | + | 49 56 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 56 57 | + | M SAL 1 2 56 57 |
| − | M SBL 1 1 62 | + | M SBL 1 1 62 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 62 -0.7915 0.0428 | + | M SBV 1 62 -0.7915 0.0428 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FACGA0041 | + | ID FL5FACGA0041 |
| − | FORMULA C35H42O22 | + | FORMULA C35H42O22 |
| − | EXACTMASS 814.216773028 | + | EXACTMASS 814.216773028 |
| − | AVERAGEMASS 814.69478 | + | AVERAGEMASS 814.69478 |
| − | SMILES c(c6O)c(c5c(O)c6)OC(=C(C5=O)OC(C(O)4)OC(C(O)C4O)COC(C(O)2)OC(C(C2OC(O3)C(C(C(C3CO)O)O)O)OC(C)=O)C)c(c1)ccc(O)c1O | + | SMILES c(c6O)c(c5c(O)c6)OC(=C(C5=O)OC(C(O)4)OC(C(O)C4O)COC(C(O)2)OC(C(C2OC(O3)C(C(C(C3CO)O)O)O)OC(C)=O)C)c(c1)ccc(O)c1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
57 62 0 0 0 0 0 0 0 0999 V2000
-4.9877 1.7090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9877 0.8330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2289 0.3949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4702 0.8330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4702 1.7090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2289 2.1470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7114 0.3949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9527 0.8330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9527 1.7090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7114 2.1470 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7114 -0.2882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9975 2.3156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2242 1.8693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5491 2.3156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5491 3.2085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2242 3.6551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9975 3.2085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2289 -0.4810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3767 3.6864 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.6908 2.3280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2242 0.2921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2453 0.7653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6555 0.0547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1334 0.2804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9270 0.0547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3374 0.7653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5485 0.5401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0715 0.7131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8695 1.0543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2732 1.5167 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5056 0.1190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7404 -0.4708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7184 -1.9312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0675 -2.5356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2369 -1.8587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7406 -1.3698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3916 -0.7654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2221 -1.4424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5670 -2.6902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5194 -2.8564 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3581 -2.5030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4194 -1.6750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5300 -3.6334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8132 -3.9746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1290 -4.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6320 -2.4283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0517 -3.0085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8722 -2.9949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5804 -3.4180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1608 -2.8380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3402 -2.8516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1303 -2.4008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1515 -2.4373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.6908 -3.0395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2242 4.5477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.3719 -3.4608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0808 -4.5477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
1 20 1 0 0 0 0
21 8 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
25 31 1 0 0 0 0
23 21 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
35 36 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
37 32 1 0 0 0 0
35 39 1 0 0 0 0
34 40 1 0 0 0 0
33 41 1 0 0 0 0
36 42 1 0 0 0 0
31 32 1 0 0 0 0
40 43 1 0 0 0 0
43 44 2 0 0 0 0
43 45 1 0 0 0 0
46 47 1 0 0 0 0
47 48 1 1 0 0 0
48 49 1 1 0 0 0
50 49 1 1 0 0 0
50 51 1 0 0 0 0
51 46 1 0 0 0 0
46 52 1 0 0 0 0
51 53 1 0 0 0 0
50 54 1 0 0 0 0
47 39 1 0 0 0 0
16 55 1 0 0 0 0
56 57 1 0 0 0 0
49 56 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 56 57
M SBL 1 1 62
M SMT 1 CH2OH
M SBV 1 62 -0.7915 0.0428
S SKP 5
ID FL5FACGA0041
FORMULA C35H42O22
EXACTMASS 814.216773028
AVERAGEMASS 814.69478
SMILES c(c6O)c(c5c(O)c6)OC(=C(C5=O)OC(C(O)4)OC(C(O)C4O)COC(C(O)2)OC(C(C2OC(O3)C(C(C(C3CO)O)O)O)OC(C)=O)C)c(c1)ccc(O)c1O
M END