Mol:FL5FACGL0018
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.5360 -1.1720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5360 -1.1720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5360 -1.8143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5360 -1.8143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9797 -2.1355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9797 -2.1355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4234 -1.8143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4234 -1.8143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4234 -1.1720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4234 -1.1720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9797 -0.8508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9797 -0.8508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8671 -2.1355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8671 -2.1355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3108 -1.8143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3108 -1.8143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3108 -1.1720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3108 -1.1720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8671 -0.8508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8671 -0.8508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8671 -2.6363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8671 -2.6363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3897 -0.7271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3897 -0.7271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9567 -1.0545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9567 -1.0545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5236 -0.7271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5236 -0.7271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5236 -0.0725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5236 -0.0725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9567 0.2549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9567 0.2549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3897 -0.0725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3897 -0.0725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9797 -2.7776 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9797 -2.7776 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1305 0.2779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1305 0.2779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2525 -2.2238 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2525 -2.2238 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2005 -0.7883 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2005 -0.7883 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9567 0.9094 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9567 0.9094 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2627 -1.8431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2627 -1.8431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9619 -2.3641 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9619 -2.3641 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5404 -2.1988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5404 -2.1988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1224 -2.3641 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1224 -2.3641 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4233 -1.8431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4233 -1.8431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8447 -2.0084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8447 -2.0084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7041 -1.9928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7041 -1.9928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0829 -1.5958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0829 -1.5958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8573 -2.0937 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8573 -2.0937 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2087 2.4930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2087 2.4930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4474 1.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4474 1.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0002 1.5676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0002 1.5676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2625 1.1238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2625 1.1238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3998 1.6358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3998 1.6358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0478 1.9173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0478 1.9173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7225 2.7897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7225 2.7897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7611 2.4136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7611 2.4136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2923 1.0454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2923 1.0454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4860 -2.3772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4860 -2.3772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2005 -2.7897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2005 -2.7897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3818 2.4979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3818 2.4979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0963 2.0854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0963 2.0854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 21 1 0 0 0 0 | + | 1 21 1 0 0 0 0 |
| − | 16 22 1 0 0 0 0 | + | 16 22 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 28 30 1 0 0 0 0 | + | 28 30 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 24 20 1 0 0 0 0 | + | 24 20 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 22 35 1 0 0 0 0 | + | 22 35 1 0 0 0 0 |
| − | 26 41 1 0 0 0 0 | + | 26 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 37 43 1 0 0 0 0 | + | 37 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 45 -6.8295 6.2193 | + | M SBV 1 45 -6.8295 6.2193 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 43 44 | + | M SAL 2 2 43 44 |
| − | M SBL 2 1 47 | + | M SBL 2 1 47 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 47 -6.7635 6.8130 | + | M SBV 2 47 -6.7635 6.8130 |
| − | S SKP 8 | + | S SKP 8 |
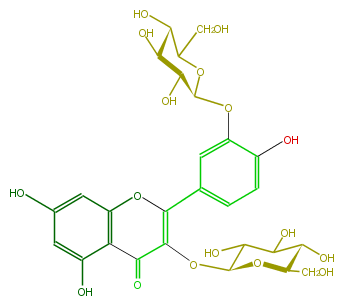
| − | ID FL5FACGL0018 | + | ID FL5FACGL0018 |
| − | KNApSAcK_ID C00005435 | + | KNApSAcK_ID C00005435 |
| − | NAME Quercetin 3,3'-diglucoside | + | NAME Quercetin 3,3'-diglucoside |
| − | CAS_RN 20188-84-5 | + | CAS_RN 20188-84-5 |
| − | FORMULA C27H30O17 | + | FORMULA C27H30O17 |
| − | EXACTMASS 626.148299534 | + | EXACTMASS 626.148299534 |
| − | AVERAGEMASS 626.5169000000001 | + | AVERAGEMASS 626.5169000000001 |
| − | SMILES O=C(C(OC(C(O)5)OC(C(C(O)5)O)CO)=1)c(c(O)4)c(cc(O)c4)OC(c(c2)ccc(c2OC(O3)C(O)C(C(O)C(CO)3)O)O)1 | + | SMILES O=C(C(OC(C(O)5)OC(C(C(O)5)O)CO)=1)c(c(O)4)c(cc(O)c4)OC(c(c2)ccc(c2OC(O3)C(O)C(C(O)C(CO)3)O)O)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-2.5360 -1.1720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5360 -1.8143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9797 -2.1355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4234 -1.8143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4234 -1.1720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9797 -0.8508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8671 -2.1355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3108 -1.8143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3108 -1.1720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8671 -0.8508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8671 -2.6363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3897 -0.7271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9567 -1.0545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5236 -0.7271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5236 -0.0725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9567 0.2549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3897 -0.0725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9797 -2.7776 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1305 0.2779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2525 -2.2238 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2005 -0.7883 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9567 0.9094 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2627 -1.8431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9619 -2.3641 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5404 -2.1988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1224 -2.3641 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4233 -1.8431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8447 -2.0084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7041 -1.9928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0829 -1.5958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8573 -2.0937 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2087 2.4930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4474 1.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0002 1.5676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2625 1.1238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3998 1.6358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0478 1.9173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7225 2.7897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7611 2.4136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2923 1.0454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4860 -2.3772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2005 -2.7897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3818 2.4979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0963 2.0854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 8 1 0 0 0 0
4 3 1 0 0 0 0
1 21 1 0 0 0 0
16 22 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 1 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
23 29 1 0 0 0 0
28 30 1 0 0 0 0
27 31 1 0 0 0 0
24 20 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
22 35 1 0 0 0 0
26 41 1 0 0 0 0
41 42 1 0 0 0 0
37 43 1 0 0 0 0
43 44 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 45
M SMT 1 CH2OH
M SBV 1 45 -6.8295 6.2193
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 43 44
M SBL 2 1 47
M SMT 2 CH2OH
M SBV 2 47 -6.7635 6.8130
S SKP 8
ID FL5FACGL0018
KNApSAcK_ID C00005435
NAME Quercetin 3,3'-diglucoside
CAS_RN 20188-84-5
FORMULA C27H30O17
EXACTMASS 626.148299534
AVERAGEMASS 626.5169000000001
SMILES O=C(C(OC(C(O)5)OC(C(C(O)5)O)CO)=1)c(c(O)4)c(cc(O)c4)OC(c(c2)ccc(c2OC(O3)C(O)C(C(O)C(CO)3)O)O)1
M END