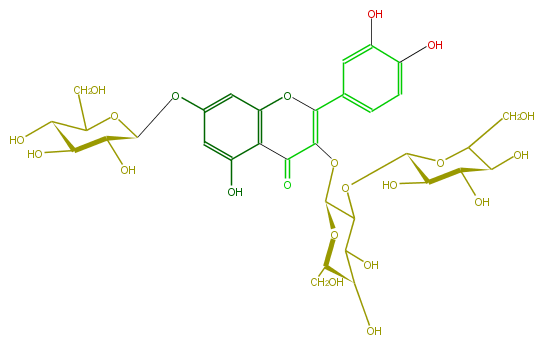
Mol:FL5FACGL0033
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 55 60 0 0 0 0 0 0 0 0999 V2000 | + | 55 60 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.3062 0.9657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3062 0.9657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3062 0.3233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3062 0.3233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7499 0.0021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7499 0.0021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1936 0.3233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1936 0.3233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1936 0.9657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1936 0.9657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7499 1.2869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7499 1.2869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3627 0.0021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3627 0.0021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9190 0.3233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9190 0.3233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9190 0.9657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9190 0.9657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3627 1.2869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3627 1.2869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3627 -0.4987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3627 -0.4987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4751 1.2867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4751 1.2867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0421 0.9594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0421 0.9594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6091 1.2867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6091 1.2867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6091 1.9414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6091 1.9414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0421 2.2688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0421 2.2688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4751 1.9414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4751 1.9414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8623 1.2867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8623 1.2867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3132 -0.0656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3132 -0.0656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7499 -0.6400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7499 -0.6400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2430 2.3074 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2430 2.3074 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7038 -1.1833 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.7038 -1.1833 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.1210 -0.8469 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.1210 -0.8469 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.3059 -1.4939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3059 -1.4939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1210 -2.1449 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.1210 -2.1449 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.7038 -2.4814 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.7038 -2.4814 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.5189 -1.8343 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.5189 -1.8343 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.5363 -0.5584 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5363 -0.5584 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9804 -2.1008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9804 -2.1008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0396 -3.4233 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0396 -3.4233 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7381 0.1188 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 2.7381 0.1188 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 3.1805 -0.4652 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.1805 -0.4652 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.8176 -0.2174 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.8176 -0.2174 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.4323 -0.2108 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.4323 -0.2108 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.9856 0.2360 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 3.9856 0.2360 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 3.4283 -0.0581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4283 -0.0581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4908 -0.4987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4908 -0.4987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1826 -0.8302 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1826 -0.8302 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9693 0.0993 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9693 0.0993 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0421 2.9233 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0421 2.9233 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3507 0.7570 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -4.3507 0.7570 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -3.9038 0.1671 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.9038 0.1671 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.2602 0.4174 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.2602 0.4174 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.6393 0.4241 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.6393 0.4241 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.0905 0.8754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0905 0.8754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6535 0.5782 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.6535 0.5782 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -4.9693 0.3998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9693 0.3998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6005 0.1332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6005 0.1332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8915 -0.2016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8915 -0.2016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7425 0.8895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7425 0.8895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5550 1.8718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5550 1.8718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8456 1.4076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8456 1.4076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8021 1.6993 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8021 1.6993 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9090 -2.4332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9090 -2.4332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0849 -2.3228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0849 -2.3228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 21 15 1 0 0 0 0 | + | 21 15 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 23 19 1 0 0 0 0 | + | 23 19 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 28 31 1 0 0 0 0 | + | 28 31 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 16 40 1 0 0 0 0 | + | 16 40 1 0 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 44 43 1 1 0 0 0 | + | 44 43 1 1 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 41 1 0 0 0 0 | + | 46 41 1 0 0 0 0 |
| − | 41 47 1 0 0 0 0 | + | 41 47 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 18 1 0 0 0 0 | + | 44 18 1 0 0 0 0 |
| − | 35 50 1 0 0 0 0 | + | 35 50 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 46 52 1 0 0 0 0 | + | 46 52 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 25 54 1 0 0 0 0 | + | 25 54 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 54 55 | + | M SAL 3 2 54 55 |
| − | M SBL 3 1 59 | + | M SBL 3 1 59 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 59 0.9622 -2.4657 | + | M SVB 3 59 0.9622 -2.4657 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 52 53 | + | M SAL 2 2 52 53 |
| − | M SBL 2 1 57 | + | M SBL 2 1 57 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 57 -3.8456 1.4076 | + | M SVB 2 57 -3.8456 1.4076 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 50 51 | + | M SAL 1 2 50 51 |
| − | M SBL 1 1 55 | + | M SBL 1 1 55 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 55 4.569 -0.1241 | + | M SVB 1 55 4.569 -0.1241 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FACGL0033 | + | ID FL5FACGL0033 |
| − | KNApSAcK_ID C00005464 | + | KNApSAcK_ID C00005464 |
| − | NAME Quercetin 3-sophoroside-7-glucoside;2-(3,4-Dihydroxyphenyl)-3-[(2-O-beta-D-glucopyranosyl-beta-D-glucopyranosyl)oxy]-7-(beta-D-glucopyranosyloxy)-5-hydroxy-4H-1-benzopyran-4-one | + | NAME Quercetin 3-sophoroside-7-glucoside;2-(3,4-Dihydroxyphenyl)-3-[(2-O-beta-D-glucopyranosyl-beta-D-glucopyranosyl)oxy]-7-(beta-D-glucopyranosyloxy)-5-hydroxy-4H-1-benzopyran-4-one |
| − | CAS_RN 42903-93-5 | + | CAS_RN 42903-93-5 |
| − | FORMULA C33H40O22 | + | FORMULA C33H40O22 |
| − | EXACTMASS 788.201122964 | + | EXACTMASS 788.201122964 |
| − | AVERAGEMASS 788.6575 | + | AVERAGEMASS 788.6575 |
| − | SMILES c(c6)(cc(c(c6)O)O)C(O3)=C(C(=O)c(c(O)4)c3cc(O[C@@H]([C@@H](O)5)OC(CO)[C@H](O)[C@@H]5O)c4)O[C@H](O2)C(C([C@@H](O)[C@H]2CO)O)O[C@H]([C@@H]1O)OC(CO)[C@H]([C@H]1O)O | + | SMILES c(c6)(cc(c(c6)O)O)C(O3)=C(C(=O)c(c(O)4)c3cc(O[C@@H]([C@@H](O)5)OC(CO)[C@H](O)[C@@H]5O)c4)O[C@H](O2)C(C([C@@H](O)[C@H]2CO)O)O[C@H]([C@@H]1O)OC(CO)[C@H]([C@H]1O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
55 60 0 0 0 0 0 0 0 0999 V2000
-1.3062 0.9657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3062 0.3233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7499 0.0021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1936 0.3233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1936 0.9657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7499 1.2869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3627 0.0021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9190 0.3233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9190 0.9657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3627 1.2869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3627 -0.4987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4751 1.2867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0421 0.9594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6091 1.2867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6091 1.9414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0421 2.2688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4751 1.9414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8623 1.2867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3132 -0.0656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7499 -0.6400 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2430 2.3074 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7038 -1.1833 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.1210 -0.8469 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.3059 -1.4939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1210 -2.1449 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.7038 -2.4814 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.5189 -1.8343 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.5363 -0.5584 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9804 -2.1008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0396 -3.4233 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7381 0.1188 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
3.1805 -0.4652 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.8176 -0.2174 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.4323 -0.2108 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.9856 0.2360 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
3.4283 -0.0581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4908 -0.4987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1826 -0.8302 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9693 0.0993 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0421 2.9233 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3507 0.7570 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-3.9038 0.1671 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.2602 0.4174 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.6393 0.4241 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.0905 0.8754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6535 0.5782 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-4.9693 0.3998 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6005 0.1332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8915 -0.2016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7425 0.8895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5550 1.8718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8456 1.4076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8021 1.6993 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9090 -2.4332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0849 -2.3228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 0 0 0 0
3 20 1 0 0 0 0
21 15 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
23 19 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
32 37 1 0 0 0 0
33 38 1 0 0 0 0
28 31 1 0 0 0 0
34 39 1 0 0 0 0
16 40 1 0 0 0 0
41 42 1 1 0 0 0
42 43 1 1 0 0 0
44 43 1 1 0 0 0
44 45 1 0 0 0 0
45 46 1 0 0 0 0
46 41 1 0 0 0 0
41 47 1 0 0 0 0
42 48 1 0 0 0 0
43 49 1 0 0 0 0
44 18 1 0 0 0 0
35 50 1 0 0 0 0
50 51 1 0 0 0 0
46 52 1 0 0 0 0
52 53 1 0 0 0 0
25 54 1 0 0 0 0
54 55 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 54 55
M SBL 3 1 59
M SMT 3 CH2OH
M SVB 3 59 0.9622 -2.4657
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 52 53
M SBL 2 1 57
M SMT 2 CH2OH
M SVB 2 57 -3.8456 1.4076
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 50 51
M SBL 1 1 55
M SMT 1 CH2OH
M SVB 1 55 4.569 -0.1241
S SKP 8
ID FL5FACGL0033
KNApSAcK_ID C00005464
NAME Quercetin 3-sophoroside-7-glucoside;2-(3,4-Dihydroxyphenyl)-3-[(2-O-beta-D-glucopyranosyl-beta-D-glucopyranosyl)oxy]-7-(beta-D-glucopyranosyloxy)-5-hydroxy-4H-1-benzopyran-4-one
CAS_RN 42903-93-5
FORMULA C33H40O22
EXACTMASS 788.201122964
AVERAGEMASS 788.6575
SMILES c(c6)(cc(c(c6)O)O)C(O3)=C(C(=O)c(c(O)4)c3cc(O[C@@H]([C@@H](O)5)OC(CO)[C@H](O)[C@@H]5O)c4)O[C@H](O2)C(C([C@@H](O)[C@H]2CO)O)O[C@H]([C@@H]1O)OC(CO)[C@H]([C@H]1O)O
M END