Mol:FL5FACGS0024
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.5891 1.1561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5891 1.1561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5891 0.3463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5891 0.3463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8879 -0.0585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8879 -0.0585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1867 0.3463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1867 0.3463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1867 1.1561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1867 1.1561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8879 1.5610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8879 1.5610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4855 -0.0585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4855 -0.0585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7843 0.3463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7843 0.3463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7843 1.1561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7843 1.1561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4855 1.5610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4855 1.5610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4855 -0.6898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4855 -0.6898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0833 1.5607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0833 1.5607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6314 1.1481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6314 1.1481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3461 1.5607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3461 1.5607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3461 2.3860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3461 2.3860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6314 2.7986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6314 2.7986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0833 2.3860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0833 2.3860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2901 1.5607 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2901 1.5607 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2874 -0.1439 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2874 -0.1439 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8879 -0.8679 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8879 -0.8679 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1451 2.8473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1451 2.8473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1431 -1.3465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1431 -1.3465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5915 -0.9225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5915 -0.9225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3585 -1.7380 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3585 -1.7380 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5915 -2.5586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5915 -2.5586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1431 -2.9828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1431 -2.9828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0900 -2.1671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0900 -2.1671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0983 -0.6839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0983 -0.6839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3694 -2.6075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3694 -2.6075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0327 -3.6236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0327 -3.6236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8281 0.0886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8281 0.0886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5051 -0.5396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5051 -0.5396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2416 -0.0925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2416 -0.0925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0033 0.0502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0033 0.0502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3510 0.5071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3510 0.5071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7235 0.0200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7235 0.0200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8433 -0.6716 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8433 -0.6716 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8922 -0.4447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8922 -0.4447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4198 0.4305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4198 0.4305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6314 3.6236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6314 3.6236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0458 0.8433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0458 0.8433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2901 0.6418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2901 0.6418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 21 15 1 0 0 0 0 | + | 21 15 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 23 19 1 0 0 0 0 | + | 23 19 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 28 31 1 0 0 0 0 | + | 28 31 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 16 40 1 0 0 0 0 | + | 16 40 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 46 -0.6948 -0.3361 | + | M SBV 1 46 -0.6948 -0.3361 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FACGS0024 | + | ID FL5FACGS0024 |
| − | FORMULA C26H28O16 | + | FORMULA C26H28O16 |
| − | EXACTMASS 596.137734848 | + | EXACTMASS 596.137734848 |
| − | AVERAGEMASS 596.49092 | + | AVERAGEMASS 596.49092 |
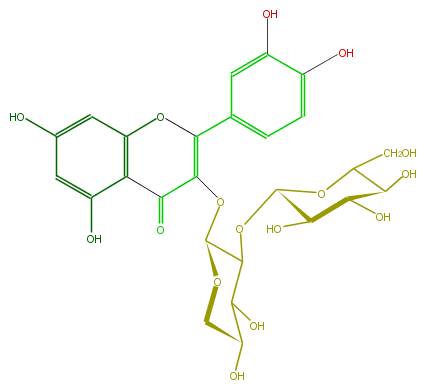
| − | SMILES c(c(O)5)cc(cc5O)C(O3)=C(C(c(c(O)4)c(cc(O)c4)3)=O)OC(O1)C(OC(C(O)2)OC(C(O)C(O)2)CO)C(O)C(C1)O | + | SMILES c(c(O)5)cc(cc5O)C(O3)=C(C(c(c(O)4)c(cc(O)c4)3)=O)OC(O1)C(OC(C(O)2)OC(C(O)C(O)2)CO)C(O)C(C1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-3.5891 1.1561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5891 0.3463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8879 -0.0585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1867 0.3463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1867 1.1561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8879 1.5610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4855 -0.0585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7843 0.3463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7843 1.1561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4855 1.5610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4855 -0.6898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0833 1.5607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6314 1.1481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3461 1.5607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3461 2.3860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6314 2.7986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0833 2.3860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2901 1.5607 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2874 -0.1439 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8879 -0.8679 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1451 2.8473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1431 -1.3465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5915 -0.9225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3585 -1.7380 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5915 -2.5586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1431 -2.9828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0900 -2.1671 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0983 -0.6839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3694 -2.6075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0327 -3.6236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8281 0.0886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5051 -0.5396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2416 -0.0925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0033 0.0502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3510 0.5071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7235 0.0200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8433 -0.6716 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8922 -0.4447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4198 0.4305 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6314 3.6236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0458 0.8433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2901 0.6418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 0 0 0 0
3 20 1 0 0 0 0
21 15 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
23 19 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
32 37 1 0 0 0 0
33 38 1 0 0 0 0
28 31 1 0 0 0 0
34 39 1 0 0 0 0
16 40 1 0 0 0 0
41 42 1 0 0 0 0
35 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 46
M SMT 1 CH2OH
M SBV 1 46 -0.6948 -0.3361
S SKP 5
ID FL5FACGS0024
FORMULA C26H28O16
EXACTMASS 596.137734848
AVERAGEMASS 596.49092
SMILES c(c(O)5)cc(cc5O)C(O3)=C(C(c(c(O)4)c(cc(O)c4)3)=O)OC(O1)C(OC(C(O)2)OC(C(O)C(O)2)CO)C(O)C(C1)O
M END