Mol:FL5FACGS0025
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -4.6473 -0.1105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6473 -0.1105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6473 -0.9355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6473 -0.9355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9329 -1.3480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9329 -1.3480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2185 -0.9355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2185 -0.9355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2185 -0.1105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2185 -0.1105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9329 0.3018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9329 0.3018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5041 -1.3480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5041 -1.3480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7896 -0.9355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7896 -0.9355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7896 -0.1105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7896 -0.1105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5041 0.3018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5041 0.3018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5041 -1.9912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5041 -1.9912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8900 0.4607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8900 0.4607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1619 0.0402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1619 0.0402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5663 0.4607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5663 0.4607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5663 1.3015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5663 1.3015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1619 1.7218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1619 1.7218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8900 1.3015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8900 1.3015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9329 -2.1726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9329 -2.1726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3456 1.7514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3456 1.7514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2829 0.3018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2829 0.3018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1261 -1.3907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1261 -1.3907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1619 2.5624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1619 2.5624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1215 -0.9286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1215 -0.9286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5077 -1.5979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5077 -1.5979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2353 -1.3855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2353 -1.3855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9827 -1.5978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9827 -1.5978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6009 -0.8878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6009 -0.8878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6260 -1.1409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6260 -1.1409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0574 -0.6261 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0574 -0.6261 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2748 -1.0464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2748 -1.0464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8086 -0.1636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8086 -0.1636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5989 -0.6741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5989 -0.6741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2590 -0.0953 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2590 -0.0953 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9978 0.1827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9978 0.1827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2626 0.5258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2626 0.5258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7192 -0.0741 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7192 -0.0741 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0523 -0.8553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0523 -0.8553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0936 -0.2014 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0936 -0.2014 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2829 0.4941 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2829 0.4941 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0971 -0.4270 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0971 -0.4270 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9397 0.9437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9397 0.9437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9397 2.0031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9397 2.0031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6969 -1.5029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6969 -1.5029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6970 -2.5624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6970 -2.5624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 1 1 0 0 0 0 | + | 20 1 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 16 22 1 0 0 0 0 | + | 16 22 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 21 24 1 0 0 0 0 | + | 21 24 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 31 30 1 0 0 0 0 | + | 31 30 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 23 40 1 0 0 0 0 | + | 23 40 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 26 43 1 0 0 0 0 | + | 26 43 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 46 -0.6771 -0.4180 | + | M SBV 1 46 -0.6771 -0.4180 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 43 44 | + | M SAL 2 2 43 44 |
| − | M SBL 2 1 48 | + | M SBL 2 1 48 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 48 -0.7143 -0.0948 | + | M SBV 2 48 -0.7143 -0.0948 |
| − | S SKP 5 | + | S SKP 5 |
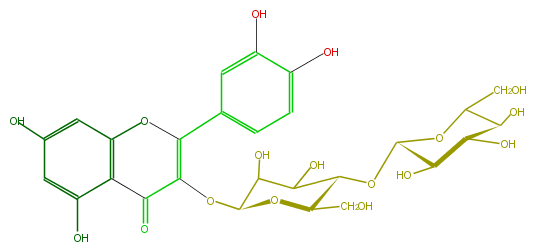
| − | ID FL5FACGS0025 | + | ID FL5FACGS0025 |
| − | FORMULA C27H30O17 | + | FORMULA C27H30O17 |
| − | EXACTMASS 626.148299534 | + | EXACTMASS 626.148299534 |
| − | AVERAGEMASS 626.5169000000001 | + | AVERAGEMASS 626.5169000000001 |
| − | SMILES c(c(O)5)cc(cc5O)C(=C1OC(C(O)3)OC(C(OC(O4)C(O)C(O)C(O)C4CO)C3O)CO)Oc(c2)c(c(O)cc2O)C1=O | + | SMILES c(c(O)5)cc(cc5O)C(=C1OC(C(O)3)OC(C(OC(O4)C(O)C(O)C(O)C4CO)C3O)CO)Oc(c2)c(c(O)cc2O)C1=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-4.6473 -0.1105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6473 -0.9355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9329 -1.3480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2185 -0.9355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2185 -0.1105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9329 0.3018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5041 -1.3480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7896 -0.9355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7896 -0.1105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5041 0.3018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5041 -1.9912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8900 0.4607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1619 0.0402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5663 0.4607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5663 1.3015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1619 1.7218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8900 1.3015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9329 -2.1726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3456 1.7514 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2829 0.3018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1261 -1.3907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1619 2.5624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1215 -0.9286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5077 -1.5979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2353 -1.3855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9827 -1.5978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6009 -0.8878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6260 -1.1409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0574 -0.6261 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2748 -1.0464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8086 -0.1636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5989 -0.6741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2590 -0.0953 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9978 0.1827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2626 0.5258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7192 -0.0741 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0523 -0.8553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0936 -0.2014 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2829 0.4941 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0971 -0.4270 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9397 0.9437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9397 2.0031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6969 -1.5029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6970 -2.5624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 1 1 0 0 0 0
21 8 1 0 0 0 0
4 3 1 0 0 0 0
16 22 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 1 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
28 29 1 0 0 0 0
27 30 1 0 0 0 0
21 24 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
32 37 1 0 0 0 0
33 38 1 0 0 0 0
31 30 1 0 0 0 0
34 39 1 0 0 0 0
23 40 1 0 0 0 0
41 42 1 0 0 0 0
35 41 1 0 0 0 0
43 44 1 0 0 0 0
26 43 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 46
M SMT 1 CH2OH
M SBV 1 46 -0.6771 -0.4180
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 43 44
M SBL 2 1 48
M SMT 2 CH2OH
M SBV 2 48 -0.7143 -0.0948
S SKP 5
ID FL5FACGS0025
FORMULA C27H30O17
EXACTMASS 626.148299534
AVERAGEMASS 626.5169000000001
SMILES c(c(O)5)cc(cc5O)C(=C1OC(C(O)3)OC(C(OC(O4)C(O)C(O)C(O)C4CO)C3O)CO)Oc(c2)c(c(O)cc2O)C1=O
M END