Mol:FL5FACGS0040
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 51 56 0 0 0 0 0 0 0 0999 V2000 | + | 51 56 0 0 0 0 0 0 0 0999 V2000 |
| − | -4.5184 1.0409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5184 1.0409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5184 0.2158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5184 0.2158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8039 -0.1967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8039 -0.1967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0895 0.2158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0895 0.2158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0895 1.0409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0895 1.0409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8039 1.4532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8039 1.4532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3750 -0.1967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3750 -0.1967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6606 0.2158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6606 0.2158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6606 1.0409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6606 1.0409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3750 1.4532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3750 1.4532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3750 -1.0179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3750 -1.0179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9671 1.4884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9671 1.4884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2389 1.0679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2389 1.0679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4891 1.4884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4891 1.4884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4891 2.3292 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4891 2.3292 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2389 2.7496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2389 2.7496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9671 2.3292 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9671 2.3292 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8039 -1.0182 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8039 -1.0182 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2686 2.7791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2686 2.7791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8872 -0.2134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8872 -0.2134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2389 3.5901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2389 3.5901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3718 1.5335 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3718 1.5335 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3139 0.1496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3139 0.1496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2832 -0.4476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2832 -0.4476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0260 -0.1586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0260 -0.1586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7429 -0.1509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7429 -0.1509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2220 0.3702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2220 0.3702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5720 0.0271 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5720 0.0271 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3947 -0.5307 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3947 -0.5307 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6583 -0.5236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6583 -0.5236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3110 0.1359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3110 0.1359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1925 -1.6881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1925 -1.6881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8192 -1.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8192 -1.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4953 -1.4715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4953 -1.4715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8459 -0.8321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8459 -0.8321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8146 -0.9146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8146 -0.9146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3388 -1.0064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3388 -1.0064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8530 -2.1088 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8530 -2.1088 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7753 -1.1908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7753 -1.1908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7080 -1.9606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7080 -1.9606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2916 -1.4541 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2916 -1.4541 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2195 -1.2907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2195 -1.2907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3718 -0.4891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3718 -0.4891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5425 -1.0673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5425 -1.0673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2062 -1.6683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2062 -1.6683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6591 -0.5830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6591 -0.5830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9709 0.0697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9709 0.0697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8535 0.5147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8535 0.5147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6028 1.2639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6028 1.2639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5624 -2.3288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5624 -2.3288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3207 -3.5901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3207 -3.5901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 1 22 1 0 0 0 0 | + | 1 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 24 29 1 0 0 0 0 | + | 24 29 1 0 0 0 0 |
| − | 25 30 1 0 0 0 0 | + | 25 30 1 0 0 0 0 |
| − | 20 23 1 0 0 0 0 | + | 20 23 1 0 0 0 0 |
| − | 26 31 1 0 0 0 0 | + | 26 31 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 32 1 0 0 0 0 | + | 36 32 1 0 0 0 0 |
| − | 33 37 1 0 0 0 0 | + | 33 37 1 0 0 0 0 |
| − | 34 38 1 0 0 0 0 | + | 34 38 1 0 0 0 0 |
| − | 35 31 1 0 0 0 0 | + | 35 31 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 1 1 0 0 0 | + | 40 41 1 1 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 43 42 1 1 0 0 0 | + | 43 42 1 1 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 39 1 0 0 0 0 | + | 44 39 1 0 0 0 0 |
| − | 39 45 1 0 0 0 0 | + | 39 45 1 0 0 0 0 |
| − | 44 46 1 0 0 0 0 | + | 44 46 1 0 0 0 0 |
| − | 43 47 1 0 0 0 0 | + | 43 47 1 0 0 0 0 |
| − | 40 38 1 0 0 0 0 | + | 40 38 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 27 48 1 0 0 0 0 | + | 27 48 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 32 50 1 0 0 0 0 | + | 32 50 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 48 49 | + | M SAL 1 2 48 49 |
| − | M SBL 1 1 54 | + | M SBL 1 1 54 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 54 -0.6315 -0.1445 | + | M SBV 1 54 -0.6315 -0.1445 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 50 51 | + | M SAL 2 2 50 51 |
| − | M SBL 2 1 56 | + | M SBL 2 1 56 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 56 -0.3699 0.6407 | + | M SBV 2 56 -0.3699 0.6407 |
| − | S SKP 5 | + | S SKP 5 |
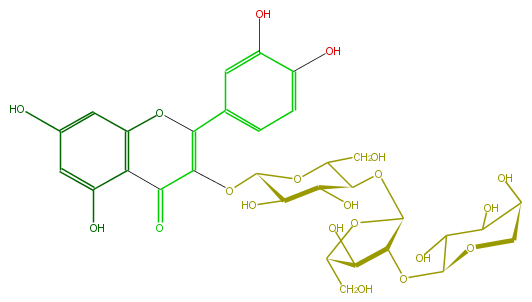
| − | ID FL5FACGS0040 | + | ID FL5FACGS0040 |
| − | FORMULA C31H36O20 | + | FORMULA C31H36O20 |
| − | EXACTMASS 728.179993592 | + | EXACTMASS 728.179993592 |
| − | AVERAGEMASS 728.60554 | + | AVERAGEMASS 728.60554 |
| − | SMILES C(CO)(O1)C(OC(C(OC(O6)C(C(O)C(O)C6)O)5)OC(C(O)5)CO)C(O)C(C(OC(C(=O)3)=C(Oc(c4)c3c(cc4O)O)c(c2)ccc(c2O)O)1)O | + | SMILES C(CO)(O1)C(OC(C(OC(O6)C(C(O)C(O)C6)O)5)OC(C(O)5)CO)C(O)C(C(OC(C(=O)3)=C(Oc(c4)c3c(cc4O)O)c(c2)ccc(c2O)O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
51 56 0 0 0 0 0 0 0 0999 V2000
-4.5184 1.0409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5184 0.2158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8039 -0.1967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0895 0.2158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0895 1.0409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8039 1.4532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3750 -0.1967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6606 0.2158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6606 1.0409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3750 1.4532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3750 -1.0179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9671 1.4884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2389 1.0679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4891 1.4884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4891 2.3292 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2389 2.7496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9671 2.3292 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8039 -1.0182 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2686 2.7791 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8872 -0.2134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2389 3.5901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.3718 1.5335 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3139 0.1496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2832 -0.4476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0260 -0.1586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7429 -0.1509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2220 0.3702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5720 0.0271 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3947 -0.5307 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6583 -0.5236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3110 0.1359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1925 -1.6881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8192 -1.8385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4953 -1.4715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8459 -0.8321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8146 -0.9146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3388 -1.0064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8530 -2.1088 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7753 -1.1908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7080 -1.9606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2916 -1.4541 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2195 -1.2907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3718 -0.4891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5425 -1.0673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2062 -1.6683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6591 -0.5830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9709 0.0697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8535 0.5147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6028 1.2639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5624 -2.3288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3207 -3.5901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 8 1 0 0 0 0
4 3 1 0 0 0 0
16 21 1 0 0 0 0
1 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
24 29 1 0 0 0 0
25 30 1 0 0 0 0
20 23 1 0 0 0 0
26 31 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 32 1 0 0 0 0
33 37 1 0 0 0 0
34 38 1 0 0 0 0
35 31 1 0 0 0 0
39 40 1 0 0 0 0
40 41 1 1 0 0 0
41 42 1 1 0 0 0
43 42 1 1 0 0 0
43 44 1 0 0 0 0
44 39 1 0 0 0 0
39 45 1 0 0 0 0
44 46 1 0 0 0 0
43 47 1 0 0 0 0
40 38 1 0 0 0 0
48 49 1 0 0 0 0
27 48 1 0 0 0 0
50 51 1 0 0 0 0
32 50 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 48 49
M SBL 1 1 54
M SMT 1 CH2OH
M SBV 1 54 -0.6315 -0.1445
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 50 51
M SBL 2 1 56
M SMT 2 CH2OH
M SBV 2 56 -0.3699 0.6407
S SKP 5
ID FL5FACGS0040
FORMULA C31H36O20
EXACTMASS 728.179993592
AVERAGEMASS 728.60554
SMILES C(CO)(O1)C(OC(C(OC(O6)C(C(O)C(O)C6)O)5)OC(C(O)5)CO)C(O)C(C(OC(C(=O)3)=C(Oc(c4)c3c(cc4O)O)c(c2)ccc(c2O)O)1)O
M END