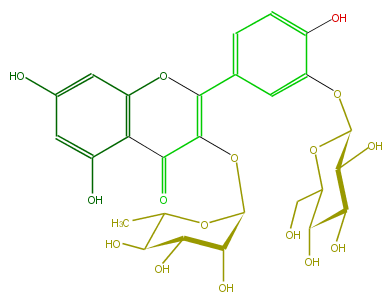
Mol:FL5FACGS0072
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.7719 2.3227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7719 2.3227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7719 1.4977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7719 1.4977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4863 1.0853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4863 1.0853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2008 1.4977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2008 1.4977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2008 2.3227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2008 2.3227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4863 2.7352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4863 2.7352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0575 1.0853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0575 1.0853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6570 1.4977 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6570 1.4977 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3715 1.0853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3715 1.0853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3715 0.2603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3715 0.2603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6570 -0.1522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6570 -0.1522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0575 0.2603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0575 0.2603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0858 1.4977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0858 1.4977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8003 1.0853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8003 1.0853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8003 0.2603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8003 0.2603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0858 -0.1522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0858 -0.1522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6570 -0.9772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6570 -0.9772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5148 1.4977 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5148 1.4977 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7719 -0.1522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7719 -0.1522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0858 -0.9772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0858 -0.9772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7927 2.6644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7927 2.6644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8294 1.1349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8294 1.1349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2569 -1.5801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2569 -1.5801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9908 -1.2029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9908 -1.2029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8207 -0.3953 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8207 -0.3953 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0859 0.3862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0859 0.3862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3519 0.0089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3519 0.0089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5219 -0.7987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5219 -0.7987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0131 -1.0315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0131 -1.0315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8574 -1.6829 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8574 -1.6829 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2444 -2.2335 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2444 -2.2335 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7734 -1.9491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7734 -1.9491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5148 0.1022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5148 0.1022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6404 -1.2783 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6404 -1.2783 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0530 -1.9930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0530 -1.9930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2595 -1.7663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2595 -1.7663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5388 -1.9755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5388 -1.9755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9515 -1.2608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9515 -1.2608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1579 -1.4875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1579 -1.4875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5341 -2.7352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5341 -2.7352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7549 -2.3695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7549 -2.3695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5855 -1.8576 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5855 -1.8576 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3955 -1.4437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3955 -1.4437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 26 22 1 0 0 0 0 | + | 26 22 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 38 37 1 1 0 0 0 | + | 38 37 1 1 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 37 40 1 0 0 0 0 | + | 37 40 1 0 0 0 0 |
| − | 36 41 1 0 0 0 0 | + | 36 41 1 0 0 0 0 |
| − | 35 42 1 0 0 0 0 | + | 35 42 1 0 0 0 0 |
| − | 34 43 1 0 0 0 0 | + | 34 43 1 0 0 0 0 |
| − | 38 19 1 0 0 0 0 | + | 38 19 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FACGS0072 | + | ID FL5FACGS0072 |
| − | FORMULA C27H30O16 | + | FORMULA C27H30O16 |
| − | EXACTMASS 610.153384912 | + | EXACTMASS 610.153384912 |
| − | AVERAGEMASS 610.5175 | + | AVERAGEMASS 610.5175 |
| − | SMILES c(c4)(ccc(c4OC(O5)C(O)C(C(O)C(CO)5)O)O)C(=C(OC(C(O)3)OC(C)C(C3O)O)1)Oc(c2)c(c(O)cc2O)C1=O | + | SMILES c(c4)(ccc(c4OC(O5)C(O)C(C(O)C(CO)5)O)O)C(=C(OC(C(O)3)OC(C)C(C3O)O)1)Oc(c2)c(c(O)cc2O)C1=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
0.7719 2.3227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7719 1.4977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4863 1.0853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2008 1.4977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2008 2.3227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4863 2.7352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0575 1.0853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6570 1.4977 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3715 1.0853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3715 0.2603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6570 -0.1522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0575 0.2603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0858 1.4977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8003 1.0853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8003 0.2603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0858 -0.1522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6570 -0.9772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5148 1.4977 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7719 -0.1522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0858 -0.9772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7927 2.6644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8294 1.1349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2569 -1.5801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9908 -1.2029 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8207 -0.3953 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0859 0.3862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3519 0.0089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5219 -0.7987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0131 -1.0315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8574 -1.6829 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2444 -2.2335 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7734 -1.9491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5148 0.1022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6404 -1.2783 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0530 -1.9930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2595 -1.7663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5388 -1.9755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9515 -1.2608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1579 -1.4875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5341 -2.7352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7549 -2.3695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5855 -1.8576 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3955 -1.4437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
25 33 1 0 0 0 0
26 22 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 1 0 0 0
37 36 1 1 0 0 0
38 37 1 1 0 0 0
38 39 1 0 0 0 0
39 34 1 0 0 0 0
37 40 1 0 0 0 0
36 41 1 0 0 0 0
35 42 1 0 0 0 0
34 43 1 0 0 0 0
38 19 1 0 0 0 0
S SKP 5
ID FL5FACGS0072
FORMULA C27H30O16
EXACTMASS 610.153384912
AVERAGEMASS 610.5175
SMILES c(c4)(ccc(c4OC(O5)C(O)C(C(O)C(CO)5)O)O)C(=C(OC(C(O)3)OC(C)C(C3O)O)1)Oc(c2)c(c(O)cc2O)C1=O
M END