Mol:FL5FACGS0097
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 55 60 0 0 0 0 0 0 0 0999 V2000 | + | 55 60 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.5943 2.2694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5943 2.2694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8798 2.6819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8798 2.6819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8798 3.5069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8798 3.5069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5943 3.9194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5943 3.9194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3087 3.5069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3087 3.5069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3087 2.6819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3087 2.6819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1653 2.2694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1653 2.2694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5491 2.6819 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5491 2.6819 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2636 2.2694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2636 2.2694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2636 1.4444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2636 1.4444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5491 1.0319 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5491 1.0319 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1653 1.4444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1653 1.4444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9781 2.6819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9781 2.6819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6926 2.2694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6926 2.2694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6926 1.4444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6926 1.4444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9781 1.0319 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9781 1.0319 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5491 0.3711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5491 0.3711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4070 2.6819 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4070 2.6819 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7643 0.9989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7643 0.9989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9781 0.2069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9781 0.2069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9006 3.8487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9006 3.8487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5943 4.6452 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5943 4.6452 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2568 -1.6555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2568 -1.6555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5359 -2.0566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5359 -2.0566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5227 -2.8815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5227 -2.8815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2305 -3.3053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2305 -3.3053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9515 -2.9042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9515 -2.9042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9646 -2.0793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9646 -2.0793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6846 -1.6788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6846 -1.6788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6583 -3.3275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6583 -3.3275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2174 -4.1291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2174 -4.1291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8290 -1.6333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8290 -1.6333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1090 -2.0338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1090 -2.0338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8747 -1.3449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8747 -1.3449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0503 -1.3833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0503 -1.3833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2045 -0.5984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2045 -0.5984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8238 -0.0528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8238 -0.0528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0006 -0.0143 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0006 -0.0143 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2555 -0.7993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2555 -0.7993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8752 -0.6019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8752 -0.6019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3676 -0.8565 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3676 -0.8565 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3550 -1.4499 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3550 -1.4499 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7126 -1.9866 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7126 -1.9866 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8293 -0.7973 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8293 -0.7973 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5257 -2.5936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5257 -2.5936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2346 -2.1716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2346 -2.1716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9545 -2.5744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9545 -2.5744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9656 -3.3993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9656 -3.3993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2568 -3.8214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2568 -3.8214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5368 -3.4186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5368 -3.4186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8289 -3.8401 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8289 -3.8401 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2679 -4.6452 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2679 -4.6452 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6846 -3.8016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6846 -3.8016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2235 -1.3478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2235 -1.3478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9314 -0.9263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9314 -0.9263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 26 31 1 0 0 0 0 | + | 26 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 36 44 1 0 0 0 0 | + | 36 44 1 0 0 0 0 |
| − | 37 19 1 0 0 0 0 | + | 37 19 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 49 52 1 0 0 0 0 | + | 49 52 1 0 0 0 0 |
| − | 48 53 1 0 0 0 0 | + | 48 53 1 0 0 0 0 |
| − | 46 54 1 0 0 0 0 | + | 46 54 1 0 0 0 0 |
| − | 54 55 2 0 0 0 0 | + | 54 55 2 0 0 0 0 |
| − | 44 32 1 0 0 0 0 | + | 44 32 1 0 0 0 0 |
| − | 41 54 1 0 0 0 0 | + | 41 54 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FACGS0097 | + | ID FL5FACGS0097 |
| − | KNApSAcK_ID C00013869 | + | KNApSAcK_ID C00013869 |
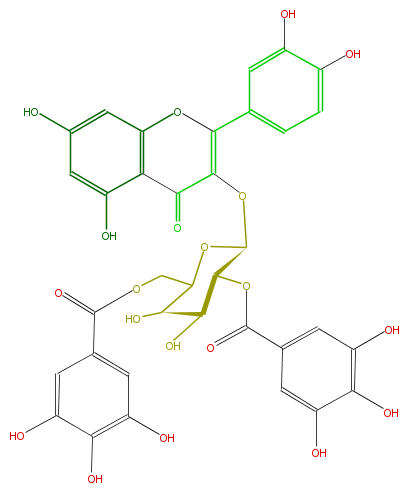
| − | NAME Quercetin 3-(2'',6''-digalloylgalactoside);3-[[2,6-Bis-O-(3,4,5-trihydroxybenzoyl)-beta-D-galactopyranosyl]oxy]-2-(3,4-dihydroxyphenyl)-5,7-dihydroxy-4H-1-benzopyran-4-one | + | NAME Quercetin 3-(2'',6''-digalloylgalactoside);3-[[2,6-Bis-O-(3,4,5-trihydroxybenzoyl)-beta-D-galactopyranosyl]oxy]-2-(3,4-dihydroxyphenyl)-5,7-dihydroxy-4H-1-benzopyran-4-one |
| − | CAS_RN 200860-88-4 | + | CAS_RN 200860-88-4 |
| − | FORMULA C35H28O20 | + | FORMULA C35H28O20 |
| − | EXACTMASS 768.117393336 | + | EXACTMASS 768.117393336 |
| − | AVERAGEMASS 768.58482 | + | AVERAGEMASS 768.58482 |
| − | SMILES c(c1C(=C(OC(C(OC(c(c6)cc(c(O)c(O)6)O)=O)4)OC(COC(=O)c(c5)cc(c(c(O)5)O)O)C(O)C4O)3)Oc(c2C3=O)cc(cc2O)O)cc(O)c(c1)O | + | SMILES c(c1C(=C(OC(C(OC(c(c6)cc(c(O)c(O)6)O)=O)4)OC(COC(=O)c(c5)cc(c(c(O)5)O)O)C(O)C4O)3)Oc(c2C3=O)cc(cc2O)O)cc(O)c(c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
55 60 0 0 0 0 0 0 0 0999 V2000
1.5943 2.2694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8798 2.6819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8798 3.5069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5943 3.9194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3087 3.5069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3087 2.6819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1653 2.2694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5491 2.6819 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2636 2.2694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2636 1.4444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5491 1.0319 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1653 1.4444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9781 2.6819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6926 2.2694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6926 1.4444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9781 1.0319 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5491 0.3711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4070 2.6819 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7643 0.9989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9781 0.2069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9006 3.8487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5943 4.6452 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2568 -1.6555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5359 -2.0566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5227 -2.8815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2305 -3.3053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9515 -2.9042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9646 -2.0793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6846 -1.6788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6583 -3.3275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2174 -4.1291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8290 -1.6333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1090 -2.0338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8747 -1.3449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0503 -1.3833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2045 -0.5984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8238 -0.0528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0006 -0.0143 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2555 -0.7993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8752 -0.6019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3676 -0.8565 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3550 -1.4499 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7126 -1.9866 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8293 -0.7973 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5257 -2.5936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2346 -2.1716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9545 -2.5744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9656 -3.3993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2568 -3.8214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5368 -3.4186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8289 -3.8401 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2679 -4.6452 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6846 -3.8016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2235 -1.3478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9314 -0.9263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
23 24 2 0 0 0 0
24 25 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
27 28 2 0 0 0 0
28 23 1 0 0 0 0
28 29 1 0 0 0 0
27 30 1 0 0 0 0
26 31 1 0 0 0 0
24 32 1 0 0 0 0
32 33 2 0 0 0 0
34 35 1 1 0 0 0
35 36 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
39 34 1 0 0 0 0
39 40 1 0 0 0 0
40 41 1 0 0 0 0
34 42 1 0 0 0 0
35 43 1 0 0 0 0
36 44 1 0 0 0 0
37 19 1 0 0 0 0
45 46 2 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 45 1 0 0 0 0
50 51 1 0 0 0 0
49 52 1 0 0 0 0
48 53 1 0 0 0 0
46 54 1 0 0 0 0
54 55 2 0 0 0 0
44 32 1 0 0 0 0
41 54 1 0 0 0 0
S SKP 8
ID FL5FACGS0097
KNApSAcK_ID C00013869
NAME Quercetin 3-(2'',6''-digalloylgalactoside);3-[[2,6-Bis-O-(3,4,5-trihydroxybenzoyl)-beta-D-galactopyranosyl]oxy]-2-(3,4-dihydroxyphenyl)-5,7-dihydroxy-4H-1-benzopyran-4-one
CAS_RN 200860-88-4
FORMULA C35H28O20
EXACTMASS 768.117393336
AVERAGEMASS 768.58482
SMILES c(c1C(=C(OC(C(OC(c(c6)cc(c(O)c(O)6)O)=O)4)OC(COC(=O)c(c5)cc(c(c(O)5)O)O)C(O)C4O)3)Oc(c2C3=O)cc(cc2O)O)cc(O)c(c1)O
M END