Mol:FL5FALNS0015
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 28 30 0 0 0 0 0 0 0 0999 V2000 | + | 28 30 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.4197 0.1167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4197 0.1167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4197 -0.5256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4197 -0.5256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8634 -0.8468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8634 -0.8468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3070 -0.5256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3070 -0.5256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3070 0.1167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3070 0.1167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8634 0.4379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8634 0.4379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7507 -0.8468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7507 -0.8468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1944 -0.5256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1944 -0.5256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1944 0.1167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1944 0.1167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7507 0.4379 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7507 0.4379 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7507 -1.3476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7507 -1.3476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3617 0.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3617 0.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9286 0.1105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9286 0.1105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4956 0.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4956 0.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4956 1.0925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4956 1.0925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9286 1.4198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9286 1.4198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3617 1.0925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3617 1.0925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2052 1.4197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2052 1.4197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6716 -1.0256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6716 -1.0256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5377 -1.5255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5377 -1.5255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2998 -1.0073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2998 -1.0073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5853 -1.4198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5853 -1.4198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7769 0.7355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7769 0.7355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2768 1.6016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2768 1.6016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0624 1.4197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0624 1.4197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7769 1.0072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7769 1.0072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3616 -0.0622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3616 -0.0622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2276 -0.5622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2276 -0.5622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 3 21 1 0 0 0 0 | + | 3 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 1 23 1 0 0 0 0 | + | 1 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 15 25 1 0 0 0 0 | + | 15 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 14 27 1 0 0 0 0 | + | 14 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | M STY 1 5 SUP | + | M STY 1 5 SUP |
| − | M SLB 1 5 5 | + | M SLB 1 5 5 |
| − | M SAL 5 2 27 28 | + | M SAL 5 2 27 28 |
| − | M SBL 5 1 29 | + | M SBL 5 1 29 |
| − | M SMT 5 OCH3 | + | M SMT 5 OCH3 |
| − | M SVB 5 29 1.7052 0.2316 | + | M SVB 5 29 1.7052 0.2316 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 25 26 | + | M SAL 4 2 25 26 |
| − | M SBL 4 1 27 | + | M SBL 4 1 27 |
| − | M SMT 4 OCH3 | + | M SMT 4 OCH3 |
| − | M SVB 4 27 2.0624 1.4197 | + | M SVB 4 27 2.0624 1.4197 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 23 24 | + | M SAL 3 2 23 24 |
| − | M SBL 3 1 25 | + | M SBL 3 1 25 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 25 -2.7769 0.7355 | + | M SVB 3 25 -2.7769 0.7355 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 21 22 | + | M SAL 2 2 21 22 |
| − | M SBL 2 1 23 | + | M SBL 2 1 23 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 23 -2.2998 -1.0073 | + | M SVB 2 23 -2.2998 -1.0073 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 19 20 | + | M SAL 1 2 19 20 |
| − | M SBL 1 1 21 | + | M SBL 1 1 21 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 21 0.0044 -0.7319 | + | M SVB 1 21 0.0044 -0.7319 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FALNS0015 | + | ID FL5FALNS0015 |
| − | KNApSAcK_ID C00004756 | + | KNApSAcK_ID C00004756 |
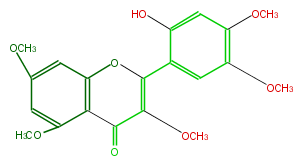
| − | NAME 2'-Hydroxy-3,5,7,4',5'-pentamethoxyflavone | + | NAME 2'-Hydroxy-3,5,7,4',5'-pentamethoxyflavone |
| − | CAS_RN 72947-61-6 | + | CAS_RN 72947-61-6 |
| − | FORMULA C20H20O8 | + | FORMULA C20H20O8 |
| − | EXACTMASS 388.11581761599996 | + | EXACTMASS 388.11581761599996 |
| − | AVERAGEMASS 388.368 | + | AVERAGEMASS 388.368 |
| − | SMILES c(c1OC)(C3=O)c(OC(=C3OC)c(c2)c(cc(OC)c(OC)2)O)cc(c1)OC | + | SMILES c(c1OC)(C3=O)c(OC(=C3OC)c(c2)c(cc(OC)c(OC)2)O)cc(c1)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
28 30 0 0 0 0 0 0 0 0999 V2000
-2.4197 0.1167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4197 -0.5256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8634 -0.8468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3070 -0.5256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3070 0.1167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8634 0.4379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7507 -0.8468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1944 -0.5256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1944 0.1167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7507 0.4379 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7507 -1.3476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3617 0.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9286 0.1105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4956 0.4378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4956 1.0925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9286 1.4198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3617 1.0925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2052 1.4197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6716 -1.0256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5377 -1.5255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2998 -1.0073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5853 -1.4198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7769 0.7355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2768 1.6016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0624 1.4197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7769 1.0072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3616 -0.0622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2276 -0.5622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
17 18 1 0 0 0 0
8 19 1 0 0 0 0
19 20 1 0 0 0 0
3 21 1 0 0 0 0
21 22 1 0 0 0 0
1 23 1 0 0 0 0
23 24 1 0 0 0 0
15 25 1 0 0 0 0
25 26 1 0 0 0 0
14 27 1 0 0 0 0
27 28 1 0 0 0 0
M STY 1 5 SUP
M SLB 1 5 5
M SAL 5 2 27 28
M SBL 5 1 29
M SMT 5 OCH3
M SVB 5 29 1.7052 0.2316
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 25 26
M SBL 4 1 27
M SMT 4 OCH3
M SVB 4 27 2.0624 1.4197
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 23 24
M SBL 3 1 25
M SMT 3 OCH3
M SVB 3 25 -2.7769 0.7355
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 21 22
M SBL 2 1 23
M SMT 2 OCH3
M SVB 2 23 -2.2998 -1.0073
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 19 20
M SBL 1 1 21
M SMT 1 OCH3
M SVB 1 21 0.0044 -0.7319
S SKP 8
ID FL5FALNS0015
KNApSAcK_ID C00004756
NAME 2'-Hydroxy-3,5,7,4',5'-pentamethoxyflavone
CAS_RN 72947-61-6
FORMULA C20H20O8
EXACTMASS 388.11581761599996
AVERAGEMASS 388.368
SMILES c(c1OC)(C3=O)c(OC(=C3OC)c(c2)c(cc(OC)c(OC)2)O)cc(c1)OC
M END