Mol:FL5FCAGL0004
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.8369 1.9769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8369 1.9769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8369 1.1673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8369 1.1673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1358 0.7624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1358 0.7624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4347 1.1673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4347 1.1673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4347 1.9769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4347 1.9769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1358 2.3816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1358 2.3816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7335 0.7624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7335 0.7624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0324 1.1673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0324 1.1673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0324 1.9769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0324 1.9769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7335 2.3816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7335 2.3816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7335 0.1313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7335 0.1313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6684 2.3814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6684 2.3814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3830 1.9689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3830 1.9689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0975 2.3814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0975 2.3814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0975 3.2066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0975 3.2066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3830 3.6191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3830 3.6191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6684 3.2066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6684 3.2066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1358 -0.0468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1358 -0.0468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8120 3.6191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8120 3.6191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7545 0.7170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7545 0.7170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0039 0.8888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0039 0.8888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1740 0.3730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1740 0.3730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1291 0.1666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1291 0.1666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8358 -0.5160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8358 -0.5160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6659 -0.0003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6659 -0.0003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7107 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7107 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3693 0.4895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3693 0.4895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2027 -0.4760 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2027 -0.4760 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3487 -2.9434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3487 -2.9434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9644 -2.2675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9644 -2.2675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0519 -2.3252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0519 -2.3252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2415 -1.8909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2415 -1.8909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6258 -2.5668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6258 -2.5668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5383 -2.5091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5383 -2.5091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2810 1.0156 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2810 1.0156 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6711 -0.7456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6711 -0.7456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8773 -1.5039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8773 -1.5039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2701 -3.6191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2701 -3.6191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4485 -1.8143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4485 -1.8143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1410 -2.2151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1410 -2.2151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8577 -2.9870 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8577 -2.9870 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5726 2.4016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5726 2.4016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2027 3.4931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2027 3.4931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 25 28 1 0 0 0 0 | + | 25 28 1 0 0 0 0 |
| − | 30 29 1 1 0 0 0 | + | 30 29 1 1 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 29 1 1 0 0 0 | + | 34 29 1 1 0 0 0 |
| − | 20 22 1 0 0 0 0 | + | 20 22 1 0 0 0 0 |
| − | 21 35 1 0 0 0 0 | + | 21 35 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 30 1 0 0 0 0 | + | 37 30 1 0 0 0 0 |
| − | 29 38 1 0 0 0 0 | + | 29 38 1 0 0 0 0 |
| − | 32 39 1 0 0 0 0 | + | 32 39 1 0 0 0 0 |
| − | 33 40 1 0 0 0 0 | + | 33 40 1 0 0 0 0 |
| − | 34 41 1 0 0 0 0 | + | 34 41 1 0 0 0 0 |
| − | 24 36 1 0 0 0 0 | + | 24 36 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 1 42 1 0 0 0 0 | + | 1 42 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 42 43 | + | M SAL 1 2 42 43 |
| − | M SBL 1 1 47 | + | M SBL 1 1 47 |
| − | M SMT 1 ^ OCH3 | + | M SMT 1 ^ OCH3 |
| − | M SBV 1 47 0.7357 -0.4248 | + | M SBV 1 47 0.7357 -0.4248 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FCAGL0004 | + | ID FL5FCAGL0004 |
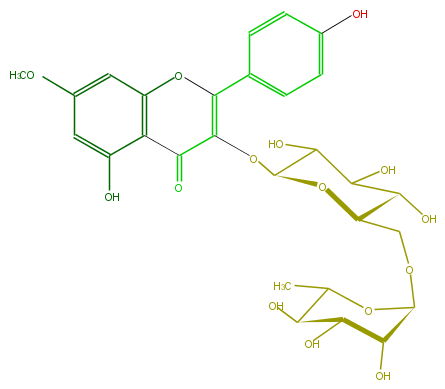
| − | FORMULA C28H32O15 | + | FORMULA C28H32O15 |
| − | EXACTMASS 608.174120354 | + | EXACTMASS 608.174120354 |
| − | AVERAGEMASS 608.54468 | + | AVERAGEMASS 608.54468 |
| − | SMILES OC(C(O)4)C(OC(COC(O5)C(C(C(O)C(C)5)O)O)C4O)OC(C1=O)=C(c(c3)ccc(O)c3)Oc(c2)c(c(O)cc2OC)1 | + | SMILES OC(C(O)4)C(OC(COC(O5)C(C(C(O)C(C)5)O)O)C4O)OC(C1=O)=C(c(c3)ccc(O)c3)Oc(c2)c(c(O)cc2OC)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-2.8369 1.9769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8369 1.1673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1358 0.7624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4347 1.1673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4347 1.9769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1358 2.3816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7335 0.7624 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0324 1.1673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0324 1.9769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7335 2.3816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7335 0.1313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6684 2.3814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3830 1.9689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0975 2.3814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0975 3.2066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3830 3.6191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6684 3.2066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1358 -0.0468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8120 3.6191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7545 0.7170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0039 0.8888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1740 0.3730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1291 0.1666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8358 -0.5160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6659 -0.0003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7107 0.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3693 0.4895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2027 -0.4760 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3487 -2.9434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9644 -2.2675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0519 -2.3252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2415 -1.8909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6258 -2.5668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5383 -2.5091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2810 1.0156 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6711 -0.7456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8773 -1.5039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2701 -3.6191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4485 -1.8143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1410 -2.2151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8577 -2.9870 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5726 2.4016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2027 3.4931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
15 19 1 0 0 0 0
8 20 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
26 27 1 0 0 0 0
25 28 1 0 0 0 0
30 29 1 1 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 1 0 0 0
34 29 1 1 0 0 0
20 22 1 0 0 0 0
21 35 1 0 0 0 0
36 37 1 0 0 0 0
37 30 1 0 0 0 0
29 38 1 0 0 0 0
32 39 1 0 0 0 0
33 40 1 0 0 0 0
34 41 1 0 0 0 0
24 36 1 0 0 0 0
42 43 1 0 0 0 0
1 42 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 42 43
M SBL 1 1 47
M SMT 1 ^ OCH3
M SBV 1 47 0.7357 -0.4248
S SKP 5
ID FL5FCAGL0004
FORMULA C28H32O15
EXACTMASS 608.174120354
AVERAGEMASS 608.54468
SMILES OC(C(O)4)C(OC(COC(O5)C(C(C(O)C(C)5)O)O)C4O)OC(C1=O)=C(c(c3)ccc(O)c3)Oc(c2)c(c(O)cc2OC)1
M END