Mol:FL5FCAGS0010
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 51 56 0 0 0 0 0 0 0 0999 V2000 | + | 51 56 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.0563 2.5366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0563 2.5366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0563 1.7116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0563 1.7116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7708 1.2991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7708 1.2991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4853 1.7116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4853 1.7116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4853 2.5366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4853 2.5366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7708 2.9491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7708 2.9491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3418 1.2991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3418 1.2991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3726 1.7116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3726 1.7116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0871 1.2991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0871 1.2991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0871 0.4741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0871 0.4741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3726 0.0616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3726 0.0616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3418 0.4741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3418 0.4741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8016 1.7116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8016 1.7116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5160 1.2991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5160 1.2991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5160 0.4741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5160 0.4741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8016 0.0616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8016 0.0616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3726 -0.6567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3726 -0.6567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2305 1.7116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2305 1.7116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0195 0.0393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0195 0.0393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0860 2.8834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0860 2.8834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8016 -0.6380 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8016 -0.6380 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9631 1.2886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9631 1.2886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9119 -0.1929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9119 -0.1929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6136 0.2414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6136 0.2414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3801 1.0330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3801 1.0330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5825 1.8330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5825 1.8330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8808 1.3988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8808 1.3988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1143 0.6071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1143 0.6071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6034 0.2911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6034 0.2911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5335 -0.4590 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5335 -0.4590 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9119 -0.8846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9119 -0.8846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4032 -0.5437 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4032 -0.5437 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9631 1.6160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9631 1.6160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2967 -1.9851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2967 -1.9851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6068 -2.5222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6068 -2.5222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6788 -2.0169 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6788 -2.0169 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0110 -1.3131 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0110 -1.3131 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5362 -0.9842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5362 -0.9842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1889 -1.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1889 -1.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3622 -1.4686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3622 -1.4686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5614 -1.1693 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5614 -1.1693 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6554 -1.3177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6554 -1.3177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2031 -2.3638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2031 -2.3638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2031 -2.9485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2031 -2.9485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0191 -2.9491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0191 -2.9491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1869 -1.6248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1869 -1.6248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6512 -2.0037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6512 -2.0037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5683 -2.0037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5683 -2.0037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0191 -2.3638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0191 -2.3638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6091 -1.7451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6091 -1.7451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6811 -1.3395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6811 -1.3395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 18 22 1 0 0 0 0 | + | 18 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 40 34 1 1 0 0 0 | + | 40 34 1 1 0 0 0 |
| − | 39 34 1 1 0 0 0 | + | 39 34 1 1 0 0 0 |
| − | 38 40 1 1 0 0 0 | + | 38 40 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 40 36 1 0 0 0 0 | + | 40 36 1 0 0 0 0 |
| − | 41 38 1 0 0 0 0 | + | 41 38 1 0 0 0 0 |
| − | 37 42 1 0 0 0 0 | + | 37 42 1 0 0 0 0 |
| − | 39 41 1 0 0 0 0 | + | 39 41 1 0 0 0 0 |
| − | 34 37 1 0 0 0 0 | + | 34 37 1 0 0 0 0 |
| − | 26 20 1 0 0 0 0 | + | 26 20 1 0 0 0 0 |
| − | 38 19 1 0 0 0 0 | + | 38 19 1 0 0 0 0 |
| − | 49 43 1 1 0 0 0 | + | 49 43 1 1 0 0 0 |
| − | 48 43 1 1 0 0 0 | + | 48 43 1 1 0 0 0 |
| − | 47 49 1 1 0 0 0 | + | 47 49 1 1 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 49 45 1 0 0 0 0 | + | 49 45 1 0 0 0 0 |
| − | 50 47 1 0 0 0 0 | + | 50 47 1 0 0 0 0 |
| − | 46 51 1 0 0 0 0 | + | 46 51 1 0 0 0 0 |
| − | 48 50 1 0 0 0 0 | + | 48 50 1 0 0 0 0 |
| − | 43 46 1 0 0 0 0 | + | 43 46 1 0 0 0 0 |
| − | 42 47 1 0 0 0 0 | + | 42 47 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
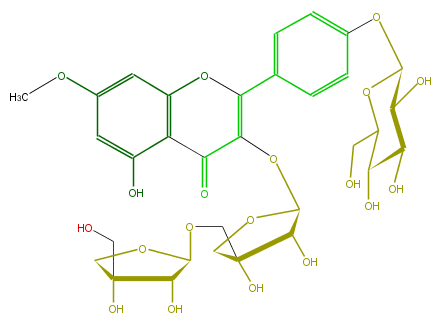
| − | ID FL5FCAGS0010 | + | ID FL5FCAGS0010 |
| − | FORMULA C32H38O19 | + | FORMULA C32H38O19 |
| − | EXACTMASS 726.200729034 | + | EXACTMASS 726.200729034 |
| − | AVERAGEMASS 726.6327200000001 | + | AVERAGEMASS 726.6327200000001 |
| − | SMILES c(c(OC)6)c(O1)c(c(O)c6)C(C(OC(C(O)4)OCC4(O)COC(C(O)5)OCC(O)5CO)=C(c(c3)ccc(c3)OC(O2)C(C(C(O)C2CO)O)O)1)=O | + | SMILES c(c(OC)6)c(O1)c(c(O)c6)C(C(OC(C(O)4)OCC4(O)COC(C(O)5)OCC(O)5CO)=C(c(c3)ccc(c3)OC(O2)C(C(C(O)C2CO)O)O)1)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
51 56 0 0 0 0 0 0 0 0999 V2000
1.0563 2.5366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0563 1.7116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7708 1.2991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4853 1.7116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4853 2.5366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7708 2.9491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3418 1.2991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3726 1.7116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0871 1.2991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0871 0.4741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3726 0.0616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3418 0.4741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8016 1.7116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5160 1.2991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5160 0.4741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8016 0.0616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3726 -0.6567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2305 1.7116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0195 0.0393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0860 2.8834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8016 -0.6380 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9631 1.2886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9119 -0.1929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6136 0.2414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3801 1.0330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5825 1.8330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8808 1.3988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1143 0.6071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6034 0.2911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5335 -0.4590 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9119 -0.8846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4032 -0.5437 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9631 1.6160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2967 -1.9851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6068 -2.5222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6788 -2.0169 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0110 -1.3131 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5362 -0.9842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1889 -1.8204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3622 -1.4686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5614 -1.1693 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6554 -1.3177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2031 -2.3638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2031 -2.9485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0191 -2.9491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1869 -1.6248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6512 -2.0037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5683 -2.0037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0191 -2.3638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6091 -1.7451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6811 -1.3395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
5 20 1 0 0 0 0
16 21 1 0 0 0 0
18 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
25 33 1 0 0 0 0
40 34 1 1 0 0 0
39 34 1 1 0 0 0
38 40 1 1 0 0 0
34 35 1 0 0 0 0
40 36 1 0 0 0 0
41 38 1 0 0 0 0
37 42 1 0 0 0 0
39 41 1 0 0 0 0
34 37 1 0 0 0 0
26 20 1 0 0 0 0
38 19 1 0 0 0 0
49 43 1 1 0 0 0
48 43 1 1 0 0 0
47 49 1 1 0 0 0
43 44 1 0 0 0 0
49 45 1 0 0 0 0
50 47 1 0 0 0 0
46 51 1 0 0 0 0
48 50 1 0 0 0 0
43 46 1 0 0 0 0
42 47 1 0 0 0 0
S SKP 5
ID FL5FCAGS0010
FORMULA C32H38O19
EXACTMASS 726.200729034
AVERAGEMASS 726.6327200000001
SMILES c(c(OC)6)c(O1)c(c(O)c6)C(C(OC(C(O)4)OCC4(O)COC(C(O)5)OCC(O)5CO)=C(c(c3)ccc(c3)OC(O2)C(C(C(O)C2CO)O)O)1)=O
M END