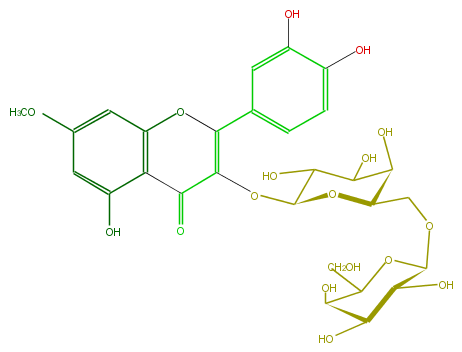
Mol:FL5FCCGA0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 45 49 0 0 0 0 0 0 0 0999 V2000 | + | 45 49 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.0093 0.8853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0093 0.8853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0093 0.0604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0093 0.0604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2948 -0.3522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2948 -0.3522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5803 0.0604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5803 0.0604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5803 0.8853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5803 0.8853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2948 1.2978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2948 1.2978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8659 -0.3522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8659 -0.3522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1514 0.0604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1514 0.0604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1514 0.8853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1514 0.8853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8659 1.2978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8659 1.2978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8659 -1.0802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8659 -1.0802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5628 1.2977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5628 1.2977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2910 0.8772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2910 0.8772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0192 1.2977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0192 1.2977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0192 2.1385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0192 2.1385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2910 2.5588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2910 2.5588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5628 2.1385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5628 2.1385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2948 -1.1010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2948 -1.1010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7094 2.5370 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7094 2.5370 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8018 0.1169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8018 0.1169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3996 -0.5799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3996 -0.5799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1731 -0.3588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1731 -0.3588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9514 -0.5799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9514 -0.5799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3538 0.1169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3538 0.1169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5801 -0.1042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5801 -0.1042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9910 0.0202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9910 0.0202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8226 0.3642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8226 0.3642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1514 0.8949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1514 0.8949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6903 -0.4369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6903 -0.4369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0254 -2.5623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0254 -2.5623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8622 -2.9149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8622 -2.9149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3924 -2.2542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3924 -2.2542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0482 -1.8661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0482 -1.8661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2918 -1.6634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2918 -1.6634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7381 -2.3175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7381 -2.3175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1184 -3.2511 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1184 -3.2511 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3675 -2.1665 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3675 -2.1665 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1060 -0.9835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1060 -0.9835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6112 -0.3789 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6112 -0.3789 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2910 3.2511 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2910 3.2511 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0286 -2.2260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0286 -2.2260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0507 -1.7998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0507 -1.7998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5802 -2.7172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5802 -2.7172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7254 1.2988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7254 1.2988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3675 2.4111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3675 2.4111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 25 27 1 0 0 0 0 | + | 25 27 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 31 36 1 0 0 0 0 | + | 31 36 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 38 29 1 0 0 0 0 | + | 38 29 1 0 0 0 0 |
| − | 21 39 1 0 0 0 0 | + | 21 39 1 0 0 0 0 |
| − | 39 8 1 0 0 0 0 | + | 39 8 1 0 0 0 0 |
| − | 16 40 1 0 0 0 0 | + | 16 40 1 0 0 0 0 |
| − | 30 41 1 0 0 0 0 | + | 30 41 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 35 42 1 0 0 0 0 | + | 35 42 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 1 44 1 0 0 0 0 | + | 1 44 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 42 43 | + | M SAL 1 2 42 43 |
| − | M SBL 1 1 47 | + | M SBL 1 1 47 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 47 0.6874 -0.5177 | + | M SBV 1 47 0.6874 -0.5177 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 44 45 | + | M SAL 2 2 44 45 |
| − | M SBL 2 1 49 | + | M SBL 2 1 49 |
| − | M SMT 2 ^ OCH3 | + | M SMT 2 ^ OCH3 |
| − | M SBV 2 49 0.7162 -0.4135 | + | M SBV 2 49 0.7162 -0.4135 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FCCGA0003 | + | ID FL5FCCGA0003 |
| − | FORMULA C28H32O17 | + | FORMULA C28H32O17 |
| − | EXACTMASS 640.163949598 | + | EXACTMASS 640.163949598 |
| − | AVERAGEMASS 640.54348 | + | AVERAGEMASS 640.54348 |
| − | SMILES c(c(OC)1)c(c(C2=O)c(OC(c(c5)cc(c(c5)O)O)=C2OC(C(O)3)OC(COC(C(O)4)OC(CO)C(O)C4O)C(O)C3O)c1)O | + | SMILES c(c(OC)1)c(c(C2=O)c(OC(c(c5)cc(c(c5)O)O)=C2OC(C(O)3)OC(COC(C(O)4)OC(CO)C(O)C4O)C(O)C3O)c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
45 49 0 0 0 0 0 0 0 0999 V2000
-3.0093 0.8853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0093 0.0604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2948 -0.3522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5803 0.0604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5803 0.8853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2948 1.2978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8659 -0.3522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1514 0.0604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1514 0.8853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8659 1.2978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8659 -1.0802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5628 1.2977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2910 0.8772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0192 1.2977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0192 2.1385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2910 2.5588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5628 2.1385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2948 -1.1010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7094 2.5370 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8018 0.1169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3996 -0.5799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1731 -0.3588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9514 -0.5799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3538 0.1169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5801 -0.1042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9910 0.0202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8226 0.3642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1514 0.8949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6903 -0.4369 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0254 -2.5623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8622 -2.9149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3924 -2.2542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0482 -1.8661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2918 -1.6634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7381 -2.3175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1184 -3.2511 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3675 -2.1665 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1060 -0.9835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6112 -0.3789 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2910 3.2511 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0286 -2.2260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0507 -1.7998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5802 -2.7172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7254 1.2988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3675 2.4111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 21 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
25 27 1 0 0 0 0
24 28 1 0 0 0 0
23 29 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
31 36 1 0 0 0 0
32 37 1 0 0 0 0
33 38 1 0 0 0 0
38 29 1 0 0 0 0
21 39 1 0 0 0 0
39 8 1 0 0 0 0
16 40 1 0 0 0 0
30 41 1 0 0 0 0
42 43 1 0 0 0 0
35 42 1 0 0 0 0
44 45 1 0 0 0 0
1 44 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 42 43
M SBL 1 1 47
M SMT 1 ^ CH2OH
M SBV 1 47 0.6874 -0.5177
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 44 45
M SBL 2 1 49
M SMT 2 ^ OCH3
M SBV 2 49 0.7162 -0.4135
S SKP 5
ID FL5FCCGA0003
FORMULA C28H32O17
EXACTMASS 640.163949598
AVERAGEMASS 640.54348
SMILES c(c(OC)1)c(c(C2=O)c(OC(c(c5)cc(c(c5)O)O)=C2OC(C(O)3)OC(COC(C(O)4)OC(CO)C(O)C4O)C(O)C3O)c1)O
M END