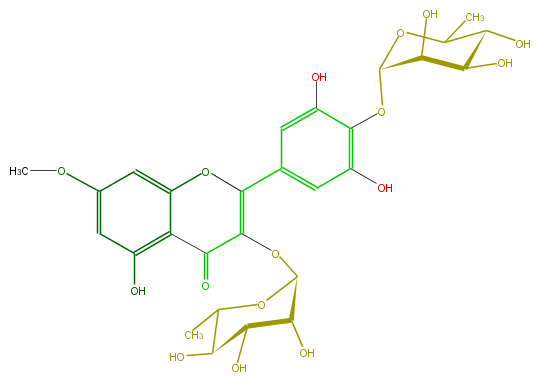
Mol:FL5FCGGS0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.7573 -1.2683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7573 -1.2683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0429 -0.8558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0429 -0.8558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0429 -0.0308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0429 -0.0308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7573 0.3817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7573 0.3817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4718 -0.0308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4718 -0.0308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4718 -0.8558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4718 -0.8558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3283 -1.2683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3283 -1.2683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6138 -0.8558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6138 -0.8558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6138 -0.0308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6138 -0.0308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3283 0.3817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3283 0.3817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1766 0.4256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1766 0.4256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8713 0.0244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8713 0.0244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5660 0.4256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5660 0.4256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5660 1.2278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5660 1.2278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8713 1.6288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8713 1.6288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1766 1.2278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1766 1.2278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2143 0.3980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2143 0.3980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7573 -1.9941 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7573 -1.9941 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3283 -1.8890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3283 -1.8890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8713 2.2890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8713 2.2890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1806 0.0707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1806 0.0707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1956 1.5913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1956 1.5913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0733 -1.2525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0733 -1.2525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0816 -2.3891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0816 -2.3891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1999 -3.2660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1999 -3.2660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5052 -2.7181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5052 -2.7181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3720 -2.6006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3720 -2.6006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4904 -1.7236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4904 -1.7236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2045 -2.2716 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2045 -2.2716 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6912 -2.8778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6912 -2.8778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8396 -3.3155 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8396 -3.3155 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7355 -3.5442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7355 -3.5442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6332 -3.2318 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6332 -3.2318 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5737 3.1741 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5737 3.1741 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1313 2.4077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1313 2.4077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9821 2.6509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9821 2.6509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8381 2.4265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8381 2.4265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2807 3.1929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2807 3.1929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4298 2.9498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4298 2.9498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9313 3.4743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9313 3.4743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9536 2.9531 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9536 2.9531 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5932 2.5567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5932 2.5567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0866 3.5442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0866 3.5442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9536 0.3980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9536 0.3980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 3 1 0 0 0 0 | + | 10 3 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 5 17 1 0 0 0 0 | + | 5 17 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 7 19 2 0 0 0 0 | + | 7 19 2 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 13 21 1 0 0 0 0 | + | 13 21 1 0 0 0 0 |
| − | 14 22 1 0 0 0 0 | + | 14 22 1 0 0 0 0 |
| − | 8 23 1 0 0 0 0 | + | 8 23 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 28 27 1 1 0 0 0 | + | 28 27 1 1 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 24 1 0 0 0 0 | + | 29 24 1 0 0 0 0 |
| − | 23 28 1 0 0 0 0 | + | 23 28 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 26 32 1 0 0 0 0 | + | 26 32 1 0 0 0 0 |
| − | 27 33 1 0 0 0 0 | + | 27 33 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 38 37 1 1 0 0 0 | + | 38 37 1 1 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 22 35 1 0 0 0 0 | + | 22 35 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 38 41 1 0 0 0 0 | + | 38 41 1 0 0 0 0 |
| − | 37 42 1 0 0 0 0 | + | 37 42 1 0 0 0 0 |
| − | 36 43 1 0 0 0 0 | + | 36 43 1 0 0 0 0 |
| − | 17 44 1 0 0 0 0 | + | 17 44 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FCGGS0002 | + | ID FL5FCGGS0002 |
| − | FORMULA C28H32O16 | + | FORMULA C28H32O16 |
| − | EXACTMASS 624.1690349759999 | + | EXACTMASS 624.1690349759999 |
| − | AVERAGEMASS 624.54408 | + | AVERAGEMASS 624.54408 |
| − | SMILES OC(C1O)C(C(OC(Oc(c5O)c(cc(c5)C(O3)=C(OC(O4)C(O)C(O)C(C(C)4)O)C(c(c32)c(cc(OC)c2)O)=O)O)1)C)O | + | SMILES OC(C1O)C(C(OC(Oc(c5O)c(cc(c5)C(O3)=C(OC(O4)C(O)C(O)C(C(C)4)O)C(c(c32)c(cc(OC)c2)O)=O)O)1)C)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-2.7573 -1.2683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0429 -0.8558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0429 -0.0308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7573 0.3817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4718 -0.0308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4718 -0.8558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3283 -1.2683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6138 -0.8558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6138 -0.0308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3283 0.3817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1766 0.4256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8713 0.0244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5660 0.4256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5660 1.2278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8713 1.6288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1766 1.2278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2143 0.3980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7573 -1.9941 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3283 -1.8890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8713 2.2890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1806 0.0707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1956 1.5913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0733 -1.2525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0816 -2.3891 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1999 -3.2660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5052 -2.7181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3720 -2.6006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4904 -1.7236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2045 -2.2716 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6912 -2.8778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8396 -3.3155 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7355 -3.5442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6332 -3.2318 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5737 3.1741 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1313 2.4077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9821 2.6509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8381 2.4265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2807 3.1929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4298 2.9498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9313 3.4743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9536 2.9531 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5932 2.5567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0866 3.5442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9536 0.3980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 3 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
5 17 1 0 0 0 0
1 18 1 0 0 0 0
7 19 2 0 0 0 0
15 20 1 0 0 0 0
13 21 1 0 0 0 0
14 22 1 0 0 0 0
8 23 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
28 27 1 1 0 0 0
28 29 1 0 0 0 0
29 24 1 0 0 0 0
23 28 1 0 0 0 0
24 30 1 0 0 0 0
25 31 1 0 0 0 0
26 32 1 0 0 0 0
27 33 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 1 0 0 0
37 36 1 1 0 0 0
38 37 1 1 0 0 0
38 39 1 0 0 0 0
39 34 1 0 0 0 0
22 35 1 0 0 0 0
39 40 1 0 0 0 0
38 41 1 0 0 0 0
37 42 1 0 0 0 0
36 43 1 0 0 0 0
17 44 1 0 0 0 0
S SKP 5
ID FL5FCGGS0002
FORMULA C28H32O16
EXACTMASS 624.1690349759999
AVERAGEMASS 624.54408
SMILES OC(C1O)C(C(OC(Oc(c5O)c(cc(c5)C(O3)=C(OC(O4)C(O)C(O)C(C(C)4)O)C(c(c32)c(cc(OC)c2)O)=O)O)1)C)O
M END