Mol:FL5FEAGS0016
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 36 39 0 0 0 0 0 0 0 0999 V2000 | + | 36 39 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.4794 -3.5077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4794 -3.5077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0127 -3.9207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0127 -3.9207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6163 -3.7010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6163 -3.7010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7279 -3.0684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7279 -3.0684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2358 -2.6554 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2358 -2.6554 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3679 -2.8751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3679 -2.8751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3315 -2.8487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3315 -2.8487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4430 -2.2161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4430 -2.2161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9509 -1.8031 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9509 -1.8031 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3473 -2.0228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3473 -2.0228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0976 -3.4915 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0976 -3.4915 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0625 -1.1708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0625 -1.1708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6777 -0.9468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6777 -0.9468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7914 -0.3021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7914 -0.3021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2899 0.1187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2899 0.1187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6746 -0.1053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6746 -0.1053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5610 -0.7500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5610 -0.7500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1082 -4.1137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1082 -4.1137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2818 0.7575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2818 0.7575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0045 1.5650 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.0045 1.5650 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.7091 1.0533 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.7091 1.0533 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.2772 1.2157 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2772 1.2157 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8486 1.0533 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.8486 1.0533 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.1441 1.5650 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.1441 1.5650 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.5760 1.4027 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.5760 1.4027 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.7080 2.0788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7080 2.0788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6168 1.8687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6168 1.8687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5703 1.3189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5703 1.3189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5934 -4.3098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5934 -4.3098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0665 -4.9856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0665 -4.9856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1829 -3.3837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1829 -3.3837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1677 -3.2102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1677 -3.2102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3364 -2.4455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3364 -2.4455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3050 -2.6942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3050 -2.6942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0434 0.7696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0434 0.7696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0433 -0.0554 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0433 -0.0554 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 25 27 1 0 0 0 0 | + | 25 27 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 21 19 1 0 0 0 0 | + | 21 19 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 2 29 1 0 0 0 0 | + | 2 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 1 31 1 0 0 0 0 | + | 1 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 8 33 1 0 0 0 0 | + | 8 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 23 35 1 0 0 0 0 | + | 23 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 35 36 | + | M SAL 4 2 35 36 |
| − | M SBL 4 1 38 | + | M SBL 4 1 38 |
| − | M SMT 4 CH2OH | + | M SMT 4 CH2OH |
| − | M SVB 4 38 3.1917 1.0801 | + | M SVB 4 38 3.1917 1.0801 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 33 34 | + | M SAL 3 2 33 34 |
| − | M SBL 3 1 36 | + | M SBL 3 1 36 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 36 -0.568 -1.6763 | + | M SVB 3 36 -0.568 -1.6763 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 31 32 | + | M SAL 2 2 31 32 |
| − | M SBL 2 1 34 | + | M SBL 2 1 34 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 34 -3.5489 0.4166 | + | M SVB 2 34 -3.5489 0.4166 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 29 30 | + | M SAL 1 2 29 30 |
| − | M SBL 1 1 32 | + | M SBL 1 1 32 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 32 -3.9061 -0.753 | + | M SVB 1 32 -3.9061 -0.753 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FEAGS0016 | + | ID FL5FEAGS0016 |
| − | KNApSAcK_ID C00005332 | + | KNApSAcK_ID C00005332 |
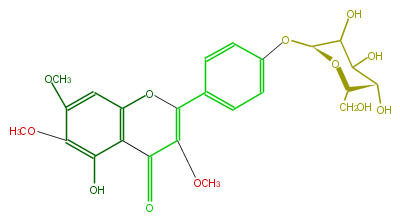
| − | NAME Pendulin | + | NAME Pendulin |
| − | CAS_RN 14801-84-4 | + | CAS_RN 14801-84-4 |
| − | FORMULA C24H26O12 | + | FORMULA C24H26O12 |
| − | EXACTMASS 506.142426296 | + | EXACTMASS 506.142426296 |
| − | AVERAGEMASS 506.45604 | + | AVERAGEMASS 506.45604 |
| − | SMILES O(C)c(c4OC)c(c(c1c4)C(C(OC)=C(c(c3)ccc(c3)O[C@@H](C(O)2)O[C@@H]([C@H](O)C(O)2)CO)O1)=O)O | + | SMILES O(C)c(c4OC)c(c(c1c4)C(C(OC)=C(c(c3)ccc(c3)O[C@@H](C(O)2)O[C@@H]([C@H](O)C(O)2)CO)O1)=O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
36 39 0 0 0 0 0 0 0 0999 V2000
-0.4794 -3.5077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0127 -3.9207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6163 -3.7010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7279 -3.0684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2358 -2.6554 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3679 -2.8751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3315 -2.8487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4430 -2.2161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9509 -1.8031 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3473 -2.0228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0976 -3.4915 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0625 -1.1708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6777 -0.9468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7914 -0.3021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2899 0.1187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6746 -0.1053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5610 -0.7500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1082 -4.1137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2818 0.7575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0045 1.5650 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.7091 1.0533 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.2772 1.2157 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8486 1.0533 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.1441 1.5650 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.5760 1.4027 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.7080 2.0788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6168 1.8687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5703 1.3189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5934 -4.3098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0665 -4.9856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1829 -3.3837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1677 -3.2102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3364 -2.4455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3050 -2.6942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0434 0.7696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0433 -0.0554 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
20 21 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
25 27 1 0 0 0 0
24 28 1 0 0 0 0
21 19 1 0 0 0 0
19 15 1 0 0 0 0
2 29 1 0 0 0 0
29 30 1 0 0 0 0
1 31 1 0 0 0 0
31 32 1 0 0 0 0
8 33 1 0 0 0 0
33 34 1 0 0 0 0
23 35 1 0 0 0 0
35 36 1 0 0 0 0
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 35 36
M SBL 4 1 38
M SMT 4 CH2OH
M SVB 4 38 3.1917 1.0801
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 33 34
M SBL 3 1 36
M SMT 3 OCH3
M SVB 3 36 -0.568 -1.6763
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 31 32
M SBL 2 1 34
M SMT 2 OCH3
M SVB 2 34 -3.5489 0.4166
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 29 30
M SBL 1 1 32
M SMT 1 OCH3
M SVB 1 32 -3.9061 -0.753
S SKP 8
ID FL5FEAGS0016
KNApSAcK_ID C00005332
NAME Pendulin
CAS_RN 14801-84-4
FORMULA C24H26O12
EXACTMASS 506.142426296
AVERAGEMASS 506.45604
SMILES O(C)c(c4OC)c(c(c1c4)C(C(OC)=C(c(c3)ccc(c3)O[C@@H](C(O)2)O[C@@H]([C@H](O)C(O)2)CO)O1)=O)O
M END